Alignement de séquences (2/2)
|
|
|
- Marie-Agnès Dupuis
- il y a 7 ans
- Total affichages :
Transcription
1 Alignement de séquences (2/2) Observation à l aide de l outil graphique : le dotplot. Simple, visuel, Très informatif : Permet de repérer une similarité globale Permet de repérer des similarités locales Permet de repérer des répétitions Oui mais!!!! Pas de mesure quantitative de similarité D1
2 Alignement de séquences identité Insertion / Délétion substitution D2 Alignement : mise en correspondance de deux séquences Quantifier et localiser la similarité dans une paire de séquences Trouver la meilleure mise en correspondance des résidus qui conserve l ordre des séquences Utilisation de la méthode des scores Trouver le meilleur score
3 Alignement de séquences Calcul de score? Le score de l alignement est la somme des scores des événements élémentaires identité substitution Insertion / Délétion D3 Matrice de similarité Pénalités
4 Alignement de séquences Rappel sur les acides aminés. D4 B Aspartic Acid ou Glutamic Acid Z Glutamine ou Glutamic Acid X inconnu Proprités physico-chimiques diagramme Venn
5 Alignement de séquences Calcul de score : matrices de similarité Blosum 62 A R N D P W A G K M H C W A Total = 13 Matrices protéiques : BLOSUM (Henikoff & Henikoff, 1992) PAM (Dayhoff, 1969) Choix de la matrice? Il n existe pas de matrice idéale!!! Blosum62 semble être la plus générale. D5
6 Alignement des séquences Calcul de score : matrices de similarité Matrice d acides nucléiques : Matrice d ADN Mésappariement de 2 purines ou 2 pyrimidines D6
7 Alignement des séquences Calcul de score : matrices de similarité D7
8 Alignement de séquences Calcul de score : pénalité des indel Ouverture Extensions pénalités ouverture extension Ajustement des pénalités? augmenter le score en fonction de la longueur du «gap» : choisir une pénalité d ouverture > à la pénalité d extension, D8 faire en sorte de ne pas affecter le score en fonction de la longueur du «gap» : pénaliser juste l ouverture du «gap», très peu ou pas du tout l extension.
9 Alignement de séquences Calcul de score : exercices Calculer les scores pour chacun des alignements et selon les 2 matrices de similarité : BLOSUM62 et BLOSUM50 Blosum 62 D9 Blosum 50
10 Alignement de séquences On distingue différents types d alignements : L alignement par paires : alignement global, alignement sur la totalité de la longueur de deux séquences nécessité d ajout d indel dans l une des séquences Algorithme de Needleman-Wunsch alignement local, identification de régions de forte homologie alignement sur les régions conservées seulement Algorithme de Smith-Waterman D10 L alignement multiple Alignement global entre plus de 2 séquences.
11 Alignement de séquences Construire des alignements de séquence? Calcul informatique (1) : approche exhaustive (naïve) o les différentes solutions alternatives d alignement possibles sont proposées, o Les scores sont calculés pour chacune de ces alternatives, o Est conservé le meilleur alignement, càd celui qui a le score le plus élevé. D11 Simple, mais temps de calcul bien trop élevé Op. x n n!!!!! Estimation du temps de calcul : alignement de 2 séquences de longueur n=20, temps de calcul (1 itération Op.=0,1 s) 300 millions d années.
12 Alignement de séquences Construire des alignements de séquence? Calcul informatique (2) : approche dynamique o Optimiser le score pour chaque paire de résidus (m*n paires), o Le meilleur score est la somme des meilleurs scores de chaque paire. D12 Exact, rapide [temps de calcul Op. x(m*n)], mais gourmand en mémoire!!!!! Estimation du temps de calcul : alignement de 2 séquences de longueur n=20, temps de calcul (1 itération Op.=0,1ms) 40 s.
13 Alignement de séquences Construire des alignements de séquence? Programmation dynamique : Alignement global de Needlemann & Wunsch (1970) Etape 1 : Construction de la matrice de comparaison. Matrice(m,n) Etape 2 : Transformation de la matrice par addition des scores. D13
14 Alignement de séquences Construire des alignements de séquence? Programmation dynamique : Alignement global de Needlemann & Wunsch (1970) Matrice initiale : Avec : S(i,j) : score dans la case (i,j)de la matrice transformée. i j Etape 2 : Transformation de la matrice par addition des scores : o Initialisation de (m,0) et (0,n) o Addition des scores : Démonstration : construction de la matrice transformée D14 se(i,j) : score élémentaire de la case d indice i et j de la matrice initiale. + y x
15 Alignement de séquences Construire des alignements de séquence? Programmation dynamique : Alignement global de Needlemann & Wunsch (1970) Matrice initiale : Avec : S(i,j) : score dans la case (i,j)de la matrice transformée. i j Etape 2 : Transformation de la matrice par addition des scores : o Initialisation de (m,0) et (0,n) o Addition des scores : Démonstration : construction de la matrice transformée D15 se(i,j) : score élémentaire de la case d indice i et j de la matrice initiale. + y x
16 Alignement de séquences Construire des alignements de séquence? Programmation dynamique : Alignement global de Needlemann & Wunsch (1970) Etape 2 : Transformation de la matrice par addition des scores : o Initialisation de (m,0) et (0,n) o Addition des scores : Matrice transformée intermédiaire : Matrice transformée finale : D16 Processus ascendant
17 Alignement de séquences Construire des alignements de séquence? Programmation dynamique : Alignement global de Needlemann & Wunsch (1970) Etape 3 : Chemin des scores maxima i j D17 Processus descendant i j
18 Alignement de séquences Construire des alignements de séquence? Programmation dynamique : Alignement global de Needlemann & Wunsch (1970) Les deux séquences présentent une similarité que l alignement global ne révèle pas!!!!! Alignement local de Smith-Waterman (1981) D18
19 Alignement de séquences Construire des alignements de séquence? Programmation dynamique : Alignement local de Smith-Waterman (1981) Dans le cas de l alignement local : N importe quelle cellule de la matrice de comparaison peut être prise comme point de départ pour le calcul des scores sommes. Tout score somme qui devient négatif stoppe la progression du calcul. Cette nouvelle case peut être initialisée à 0 et constituer un nouveau point de départ. D19
20 Alignement de séquences Un outil d alignement : Align. Choix de la méthode : «needle» (global) Needleman-Wunsch «water» (local) Smith-Waterman D20
21 Alignement de séquences Un outil d alignement : Align. Etape 1 : Entrée des deux séquences à analyser D21 Etape 2 : Choix :. des penalités et. de la matrice de similarité. Etape 3 : Exécution.
22 Alignement de séquences Un outil d alignement : Align. Des Résultats : Identité : Proportion des paires de résidus identiques entre les deux séquences alignées (exprimée en %) Similarité : Mesure de la ressemblance entre les deux séquences alignées. Le degré de similitude entre les deux séquences est quantifié par un score basé sur le % de similarité (% d identité + % de substitutions conservatives). Score : Somme des scores des événements élémentaires. D22
23 Recherche de similitudes dans une base de séquences (base de données)??? Pourquoi? Savoir si ma séquence ressemble à d autres séquences déjà connues, Trouver toutes les séquences d une même famille, Rechercher toutes les séquences qui contiennent un motif donné. D23
24 Recherche de similitudes dans une base de séquences (base de données)??? Méthodes? Recherche à grande échelle (bases de données contenant des 10zaines de milliers de séquences) pas raisonnable d utiliser des programmes classiques d alignement D24 Utilisation d heuristiques : BLAST & FASTA Basic Local Alignment Search Tool (Altschul et al, 1990) Méthodes approximatives basées sur une idée de filtrage.
25 D25 BLAST : Basic Local Aligment Search Tool : Recherche de régions de similarité locales. L algorithme BLAST : Étape 1 : création d une liste de tous les fragments (mots) de taille k (avec k petit: 11 pour les acides nucléiques, 2 ou 3 pour les protéines) trouvés dans la séquence requête et qui obtiennent un score > à un seuil donné. Etape 2 : construction d un automate fini déterministe pour retrouver les positions de tous les mots dans toutes les séquences de la banque de données. A partir de ces positions, BLAST essaie d étendre l alignement local tant que le score reste au dessus d un seuil donné. Toutes ces positions dans les séquences de la banque permettent ainsi de construire la liste des segments les plus similaires ou HSP (High Scoring Segment Pairs). Étape 3 : ordonner les alignements locaux, appelés MSP (Maximal-scoring Segment Pairs) en fonction de leur score maximun.
26 Étape 1 : Création d une liste de tous les fragments (mots) de taille k trouvés avec un score > seuil Etape 2 : Construction d un automate retrouver les positions dans séquences de la BD. Extension de l alignement local score reste au dessus d un seuil donné =>construction de la liste des HSP, D26 Etape 3 : Construction de la liste de MSP (HSP à score maximal)
27 BLAST : Basic Local Aligment Search Tool : Recherche de régions de similarité locales. Evaluer les résultats de BLAST : les indicateurs Le score brute : est la somme des scores des MSP qui composent cet alignement. Le score modifié : scores bruts convertis du logarithme (utilisés pour la création de la matrice de scores) au logarithme à base 2. Cela permet de comparer les scores obtenus entre différents alignements. La E-value : donne les informations sur la significativité d un alignement donné. La E-value d un alignement indique le nombre d alignements que l on s attendrait à trouver dans les banques avec un score supérieur ou égal au score qu obtiendrait la séquence requête contre une banque de données aléatoire (probabilité d'observer au hasard ce score à travers la banque de séquences considérée). Plus la E-value est faible, plus l'alignement est significatif. D27
28 BLAST : Basic Local Aligment Search Tool : Recherche de régions de similarité locales. Evaluer les résultats de BLAST : les indicateurs D28
29 Implémentation de l algorithme BLAST : NCBI BLAST & WU-BLAST? NCBI BLAST & WU-BLAST : Utilisables en tant que serveurs Web ou paquetage logiciel téléchargeable. NCBI BLAST : disponible sur le serveur du NCBI. Pour les versions les plus récentes : profit du développement de méthodes permettant de comparer les profils de séquences multiples. WU-BLAST : version alternative développée et maintenue à partir de la version NCBI Interrogation de bases de données protéines. D29
30 BLAST : Basic Local Aligment Search Tool : Recherche de régions de similarité locales. Les différents programmes BLAST : blastp : séquence requête protéique contre banque de données de séquences protéiques. blastn : séquence requête nucléique contre banque de données de séquences nucléiques. blastx : séquence requête nucléique traduite dans les six phases de lecture contre banque de données de séquences protéiques. tblastn : séquence requête protéique contre banque de données de séquences nucléiques dynamiquement traduite dans les six phases de lecture. tblastx : séquence requête nucléiques traduite dans les six phases de lecture contre banque de données de séquences nucléiques dynamiquement traduites suivant les six phases de lecture. D30
31 BLAST : Basic Local Aligment Search Tool : Recherche de régions de similarité locales. Les différents programmes BLAST : D31
32 Choisir l espèce étudiée : Choisir le programme : Choisir la base de données : D32
33 Entrer la séquence requête : Ajuster la sélection de la base de données : Optimisez les contraintes de sélection : D33
34 Réglez les paramètres de votre recherche : D34
35 Les résultats!! D35
36 D36
37 D37
38 D38
39 D39
40 D40
41 D41
42 D42
43 D43
44 D44
45 D45
46 D46
47 D47
48 D48
49 D49
50 Alignement multiple de séquences? CLUSTALL : Algorithme de type progressif. Composé de trois étapes : D50 Alignement par paires A B C D Calcul d un arbre de guidage Alignement progressif. A B C D A B C D Matrice de similarité B D Arbre de A guidage C similarité
51 Alignement multiple de séquences? CLUSTALL : Algorithme de type progressif. Composé de trois étapes : Calcul d un arbre de guidage Alignement progressif. B D Arbre de guidage A C similarité D51 B D Alignement des paires les plus similaires Gaps pour optimiser l alignement A C B D A C Nouveaux gaps pour optimiser L alignement (BD) avec (AC)
52 D52
53 D53
54 Merci de votre attention!!!!!!!! D54
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST.
 La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
Big data et sciences du Vivant L'exemple du séquençage haut débit
 Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard bioinfo@genopole.toulouse.inra.fr INRA - MIAT - Plate-forme
Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard bioinfo@genopole.toulouse.inra.fr INRA - MIAT - Plate-forme
IMMUNOLOGIE. La spécificité des immunoglobulines et des récepteurs T. Informations scientifiques
 IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
Perl Orienté Objet BioPerl There is more than one way to do it
 Perl Orienté Objet BioPerl There is more than one way to do it Bérénice Batut, berenice.batut@udamail.fr DUT Génie Biologique Option Bioinformatique Année 2014-2015 Perl Orienté Objet - BioPerl Rappels
Perl Orienté Objet BioPerl There is more than one way to do it Bérénice Batut, berenice.batut@udamail.fr DUT Génie Biologique Option Bioinformatique Année 2014-2015 Perl Orienté Objet - BioPerl Rappels
Resolution limit in community detection
 Introduction Plan 2006 Introduction Plan Introduction Introduction Plan Introduction Point de départ : un graphe et des sous-graphes. But : quantifier le fait que les sous-graphes choisis sont des modules.
Introduction Plan 2006 Introduction Plan Introduction Introduction Plan Introduction Point de départ : un graphe et des sous-graphes. But : quantifier le fait que les sous-graphes choisis sont des modules.
Module Analyse de Génomes 2011-2012 Master 2 module FMBS 326 Immunoinformatique
 Module Analyse de Génomes 2011-2012 Master 2 module FMBS 326 Immunoinformatique Planning du Module : Date Heure Salle 12/12 9h-12h TD info TA1Z bat 25 13h-17h TD info TA1Z bat 25 13/12 9h-12h TD info TA1Z
Module Analyse de Génomes 2011-2012 Master 2 module FMBS 326 Immunoinformatique Planning du Module : Date Heure Salle 12/12 9h-12h TD info TA1Z bat 25 13h-17h TD info TA1Z bat 25 13/12 9h-12h TD info TA1Z
Le Data Mining au service du Scoring ou notation statistique des emprunteurs!
 France Le Data Mining au service du Scoring ou notation statistique des emprunteurs! Comme le rappelle la CNIL dans sa délibération n 88-083 du 5 Juillet 1988 portant adoption d une recommandation relative
France Le Data Mining au service du Scoring ou notation statistique des emprunteurs! Comme le rappelle la CNIL dans sa délibération n 88-083 du 5 Juillet 1988 portant adoption d une recommandation relative
Introduc)on à Ensembl/ Biomart : Par)e pra)que
 Introduc)on à Ensembl/ Biomart : Par)e pra)que Stéphanie Le Gras Jean Muller NAVIGUER DANS ENSEMBL : PARTIE PRATIQUE 2 Naviga)on dans Ensembl : Pra)que Exercice 1 1.a. Quelle est la version de l assemblage
Introduc)on à Ensembl/ Biomart : Par)e pra)que Stéphanie Le Gras Jean Muller NAVIGUER DANS ENSEMBL : PARTIE PRATIQUE 2 Naviga)on dans Ensembl : Pra)que Exercice 1 1.a. Quelle est la version de l assemblage
Objectifs. Clustering. Principe. Applications. Applications. Cartes de crédits. Remarques. Biologie, Génomique
 Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
CHAPITRE 3 LA SYNTHESE DES PROTEINES
 CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
Plus courts chemins, programmation dynamique
 1 Plus courts chemins, programmation dynamique 1. Plus courts chemins à partir d un sommet 2. Plus courts chemins entre tous les sommets 3. Semi-anneau 4. Programmation dynamique 5. Applications à la bio-informatique
1 Plus courts chemins, programmation dynamique 1. Plus courts chemins à partir d un sommet 2. Plus courts chemins entre tous les sommets 3. Semi-anneau 4. Programmation dynamique 5. Applications à la bio-informatique
Les algorithmes de base du graphisme
 Les algorithmes de base du graphisme Table des matières 1 Traçage 2 1.1 Segments de droites......................... 2 1.1.1 Algorithmes simples.................... 3 1.1.2 Algorithmes de Bresenham (1965).............
Les algorithmes de base du graphisme Table des matières 1 Traçage 2 1.1 Segments de droites......................... 2 1.1.1 Algorithmes simples.................... 3 1.1.2 Algorithmes de Bresenham (1965).............
Sauvegarde collaborative entre pairs Ludovic Courtès LAAS-CNRS
 Sauvegarde collaborative entre pairs 1 Sauvegarde collaborative entre pairs Ludovic Courtès LAAS-CNRS Sauvegarde collaborative entre pairs 2 Introduction Pourquoi pair à pair? Utilisation de ressources
Sauvegarde collaborative entre pairs 1 Sauvegarde collaborative entre pairs Ludovic Courtès LAAS-CNRS Sauvegarde collaborative entre pairs 2 Introduction Pourquoi pair à pair? Utilisation de ressources
Comment reproduire les résultats de l article : POP-Java : Parallélisme et distribution orienté objet
 Comment reproduire les résultats de l article : POP-Java : Parallélisme et distribution orienté objet Beat Wolf 1, Pierre Kuonen 1, Thomas Dandekar 2 1 icosys, Haute École Spécialisée de Suisse occidentale,
Comment reproduire les résultats de l article : POP-Java : Parallélisme et distribution orienté objet Beat Wolf 1, Pierre Kuonen 1, Thomas Dandekar 2 1 icosys, Haute École Spécialisée de Suisse occidentale,
Identification de nouveaux membres dans des familles d'interleukines
 Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
Big Data et Graphes : Quelques pistes de recherche
 Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci http://liris.cnrs.fr/hamamache.kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de
Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci http://liris.cnrs.fr/hamamache.kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de
Identification des incontournables à la démarche Lean
 Identification des incontournables à la démarche Lean Analyse des résultats 7/09/2010 Barbara Lyonnet, Laboratoire SYMME, Université de Savoie barbara.lyonnet@univ-savoie.fr 1. Introduction Toute entreprise
Identification des incontournables à la démarche Lean Analyse des résultats 7/09/2010 Barbara Lyonnet, Laboratoire SYMME, Université de Savoie barbara.lyonnet@univ-savoie.fr 1. Introduction Toute entreprise
WEBANALYTICS Sur le chemin de l excellence
 WEBANALYTICS Sur le chemin de l excellence Une démarche centrée sur l évaluation de sa propre performance! 2004-2008 Philippe FLOC H Strategic Consulting Tous droits réservés Reproduction interdite WEBMETRICS
WEBANALYTICS Sur le chemin de l excellence Une démarche centrée sur l évaluation de sa propre performance! 2004-2008 Philippe FLOC H Strategic Consulting Tous droits réservés Reproduction interdite WEBMETRICS
Plan 1/9/2013. Génération et exploitation de données. CEP et applications. Flux de données et notifications. Traitement des flux Implémentation
 Complex Event Processing Traitement de flux de données en temps réel Romain Colle R&D Project Manager Quartet FS Plan Génération et exploitation de données CEP et applications Flux de données et notifications
Complex Event Processing Traitement de flux de données en temps réel Romain Colle R&D Project Manager Quartet FS Plan Génération et exploitation de données CEP et applications Flux de données et notifications
Nouveau Barème W.B.F. de points de victoire 4 à 48 donnes
 Nouveau Barème W.B.F. de points de victoire 4 à 48 donnes Pages 4 à 48 barèmes 4 à 48 donnes Condensé en une page: Page 2 barèmes 4 à 32 ( nombre pair de donnes ) Page 3 Tous les autres barèmes ( PV de
Nouveau Barème W.B.F. de points de victoire 4 à 48 donnes Pages 4 à 48 barèmes 4 à 48 donnes Condensé en une page: Page 2 barèmes 4 à 32 ( nombre pair de donnes ) Page 3 Tous les autres barèmes ( PV de
Spécificités, Applications et Outils
 Spécificités, Applications et Outils Ricco Rakotomalala Université Lumière Lyon 2 Laboratoire ERIC Laboratoire ERIC 1 Ricco Rakotomalala ricco.rakotomalala@univ-lyon2.fr http://chirouble.univ-lyon2.fr/~ricco/data-mining
Spécificités, Applications et Outils Ricco Rakotomalala Université Lumière Lyon 2 Laboratoire ERIC Laboratoire ERIC 1 Ricco Rakotomalala ricco.rakotomalala@univ-lyon2.fr http://chirouble.univ-lyon2.fr/~ricco/data-mining
Quantification Scalaire et Prédictive
 Quantification Scalaire et Prédictive Marco Cagnazzo Département Traitement du Signal et des Images TELECOM ParisTech 7 Décembre 2012 M. Cagnazzo Quantification Scalaire et Prédictive 1/64 Plan Introduction
Quantification Scalaire et Prédictive Marco Cagnazzo Département Traitement du Signal et des Images TELECOM ParisTech 7 Décembre 2012 M. Cagnazzo Quantification Scalaire et Prédictive 1/64 Plan Introduction
Détection des duplications en tandem au niveau nucléique à l'aide de la théorie des flots
 Université Toulouse 3 Paul Sabatier(UT3 Paul Sabatier) Informatique Spécialité Bioinformatique Eric AUDEMARD lundi 28 novembre 2011 Détection des duplications en tandem au niveau nucléique à l'aide de
Université Toulouse 3 Paul Sabatier(UT3 Paul Sabatier) Informatique Spécialité Bioinformatique Eric AUDEMARD lundi 28 novembre 2011 Détection des duplications en tandem au niveau nucléique à l'aide de
Formavie 2010. 2 Différentes versions du format PDB...3. 3 Les champs dans les fichiers PDB...4. 4 Le champ «ATOM»...5. 6 Limites du format PDB...
 Formavie 2010 Les fichiers PDB Les fichiers PDB contiennent les informations qui vont permettre à des logiciels de visualisation moléculaire (ex : RasTop ou Jmol) d afficher les molécules. Un fichier au
Formavie 2010 Les fichiers PDB Les fichiers PDB contiennent les informations qui vont permettre à des logiciels de visualisation moléculaire (ex : RasTop ou Jmol) d afficher les molécules. Un fichier au
Cryptologie. Algorithmes à clé publique. Jean-Marc Robert. Génie logiciel et des TI
 Cryptologie Algorithmes à clé publique Jean-Marc Robert Génie logiciel et des TI Plan de la présentation Introduction Cryptographie à clé publique Les principes essentiels La signature électronique Infrastructures
Cryptologie Algorithmes à clé publique Jean-Marc Robert Génie logiciel et des TI Plan de la présentation Introduction Cryptographie à clé publique Les principes essentiels La signature électronique Infrastructures
à moyen Risque moyen Risq à élevé Risque élevé Risq e Risque faible à moyen Risq Risque moyen à élevé Risq
 e élevé Risque faible Risq à moyen Risque moyen Risq à élevé Risque élevé Risq e Risque faible à moyen Risq Risque moyen à élevé Risq L e s I n d i c e s F u n d a t a é Risque Les Indices de faible risque
e élevé Risque faible Risq à moyen Risque moyen Risq à élevé Risque élevé Risq e Risque faible à moyen Risq Risque moyen à élevé Risq L e s I n d i c e s F u n d a t a é Risque Les Indices de faible risque
TP3 Test immunologique et spécificité anticorps - déterminant antigénique
 TP3 Test immunologique et spécificité anticorps - déterminant antigénique Partie 1 : Spécificité d'un anticorps pour un déterminant antigénique du VIH La séropositivité pour le VIH correspond à la présence
TP3 Test immunologique et spécificité anticorps - déterminant antigénique Partie 1 : Spécificité d'un anticorps pour un déterminant antigénique du VIH La séropositivité pour le VIH correspond à la présence
Mieux comprendre les certificats SSL THAWTE EST L UN DES PRINCIPAUX FOURNISSEURS DE CERTIFICATS SSL DANS LE MONDE
 Mieux comprendre les certificats SSL THAWTE EST L UN DES PRINCIPAUX FOURNISSEURS DE CERTIFICATS SSL DANS LE MONDE sommaire MIEUX COMPRENDRE LES CERTIFICATS SSL...1 SSL et certificats SSL : définition...1
Mieux comprendre les certificats SSL THAWTE EST L UN DES PRINCIPAUX FOURNISSEURS DE CERTIFICATS SSL DANS LE MONDE sommaire MIEUX COMPRENDRE LES CERTIFICATS SSL...1 SSL et certificats SSL : définition...1
GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010
 GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010 Analyse de la diversité moléculaire des régions génomiques de 30 gènes du développement méristématique dans une core collection
GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010 Analyse de la diversité moléculaire des régions génomiques de 30 gènes du développement méristématique dans une core collection
Analyses de Variance à un ou plusieurs facteurs Régressions Analyse de Covariance Modèles Linéaires Généralisés
 Analyses de Variance à un ou plusieurs facteurs Régressions Analyse de Covariance Modèles Linéaires Généralisés Professeur Patrice Francour francour@unice.fr Une grande partie des illustrations viennent
Analyses de Variance à un ou plusieurs facteurs Régressions Analyse de Covariance Modèles Linéaires Généralisés Professeur Patrice Francour francour@unice.fr Une grande partie des illustrations viennent
Julien MATHEVET Alexandre BOISSY GSID 4. Rapport RE09. Load Balancing et migration
 Julien MATHEVET Alexandre BOISSY GSID 4 Rapport Load Balancing et migration Printemps 2001 SOMMAIRE INTRODUCTION... 3 SYNTHESE CONCERNANT LE LOAD BALANCING ET LA MIGRATION... 4 POURQUOI FAIRE DU LOAD BALANCING?...
Julien MATHEVET Alexandre BOISSY GSID 4 Rapport Load Balancing et migration Printemps 2001 SOMMAIRE INTRODUCTION... 3 SYNTHESE CONCERNANT LE LOAD BALANCING ET LA MIGRATION... 4 POURQUOI FAIRE DU LOAD BALANCING?...
DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION
 DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION PRINCIPES DE BASE SUR LES DONNEES ET LE CALCUL HAUTE PERFORMANCE Lois de Gray sur l ingénierie des données 1 : Les calculs scientifiques traitent des volumes considérables
DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION PRINCIPES DE BASE SUR LES DONNEES ET LE CALCUL HAUTE PERFORMANCE Lois de Gray sur l ingénierie des données 1 : Les calculs scientifiques traitent des volumes considérables
MABioVis. Bio-informatique et la
 MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
Objectif. Participant. Prérequis. Oracle BI Suite EE 10g R3 - Développer des référentiels. 5 Jours [35 Heures]
![Objectif. Participant. Prérequis. Oracle BI Suite EE 10g R3 - Développer des référentiels. 5 Jours [35 Heures] Objectif. Participant. Prérequis. Oracle BI Suite EE 10g R3 - Développer des référentiels. 5 Jours [35 Heures]](/thumbs/24/2319607.jpg) Objectif Utiliser les techniques de gestion de la mise en cache pour contrôler et améliorer les performances des requêtes Définir des mesures simples et des mesures calculées pour une table de faits Créer
Objectif Utiliser les techniques de gestion de la mise en cache pour contrôler et améliorer les performances des requêtes Définir des mesures simples et des mesures calculées pour une table de faits Créer
Base de données bibliographiques Pubmed-Medline
 Chapitre 1 ; Domaine 1 ; Documentation ; Champs référentiels 1.1.1, 1.1.2 et 1.1.3 Base de données bibliographiques Pubmed-Medline D r Patrick Deschamps,, 30 mai 2007 PLAN C2i métiers de la santé Introduction
Chapitre 1 ; Domaine 1 ; Documentation ; Champs référentiels 1.1.1, 1.1.2 et 1.1.3 Base de données bibliographiques Pubmed-Medline D r Patrick Deschamps,, 30 mai 2007 PLAN C2i métiers de la santé Introduction
Big Data et Graphes : Quelques pistes de recherche
 Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de Lyon/Université Claude Bernard Lyon 1/Université
Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de Lyon/Université Claude Bernard Lyon 1/Université
Modélisation multi-agents - Agents réactifs
 Modélisation multi-agents - Agents réactifs Syma cursus CSI / SCIA Julien Saunier - julien.saunier@ifsttar.fr Sources www-lih.univlehavre.fr/~olivier/enseignement/masterrecherche/cours/ support/algofourmis.pdf
Modélisation multi-agents - Agents réactifs Syma cursus CSI / SCIA Julien Saunier - julien.saunier@ifsttar.fr Sources www-lih.univlehavre.fr/~olivier/enseignement/masterrecherche/cours/ support/algofourmis.pdf
1 Modélisation d être mauvais payeur
 1 Modélisation d être mauvais payeur 1.1 Description Cet exercice est très largement inspiré d un document que M. Grégoire de Lassence de la société SAS m a transmis. Il est intitulé Guide de démarrage
1 Modélisation d être mauvais payeur 1.1 Description Cet exercice est très largement inspiré d un document que M. Grégoire de Lassence de la société SAS m a transmis. Il est intitulé Guide de démarrage
Cryptologie et physique quantique : Espoirs et menaces. Objectifs 2. distribué sous licence creative common détails sur www.matthieuamiguet.
 : Espoirs et menaces Matthieu Amiguet 2005 2006 Objectifs 2 Obtenir une compréhension de base des principes régissant le calcul quantique et la cryptographie quantique Comprendre les implications sur la
: Espoirs et menaces Matthieu Amiguet 2005 2006 Objectifs 2 Obtenir une compréhension de base des principes régissant le calcul quantique et la cryptographie quantique Comprendre les implications sur la
Francis BISSON (06 794 819) Kenny CÔTÉ (06 836 427) Pierre-Luc ROGER (06 801 883) IFT702 Planification en intelligence artificielle
 Francis BISSON (06 794 819) Kenny CÔTÉ (06 836 427) Pierre-Luc ROGER (06 801 883) PLANIFICATION DE TÂCHES DANS MS PROJECT IFT702 Planification en intelligence artificielle Présenté à M. Froduald KABANZA
Francis BISSON (06 794 819) Kenny CÔTÉ (06 836 427) Pierre-Luc ROGER (06 801 883) PLANIFICATION DE TÂCHES DANS MS PROJECT IFT702 Planification en intelligence artificielle Présenté à M. Froduald KABANZA
NOTE SUR LA MODELISATION DU RISQUE D INFLATION
 NOTE SUR LA MODELISATION DU RISQUE D INFLATION 1/ RESUME DE L ANALYSE Cette étude a pour objectif de modéliser l écart entre deux indices d inflation afin d appréhender le risque à très long terme qui
NOTE SUR LA MODELISATION DU RISQUE D INFLATION 1/ RESUME DE L ANALYSE Cette étude a pour objectif de modéliser l écart entre deux indices d inflation afin d appréhender le risque à très long terme qui
Projet Active Object
 Projet Active Object TAO Livrable de conception et validation Romain GAIDIER Enseignant : M. Noël PLOUZEAU, ISTIC / IRISA Pierre-François LEFRANC Master 2 Informatique parcours MIAGE Méthodes Informatiques
Projet Active Object TAO Livrable de conception et validation Romain GAIDIER Enseignant : M. Noël PLOUZEAU, ISTIC / IRISA Pierre-François LEFRANC Master 2 Informatique parcours MIAGE Méthodes Informatiques
Insulinothérapie et diabète de type 1
 Insulinothérapie et diabète de type 1 Introduction: la molécule d insuline L instauration de l insulinothérapie Dispositif d administration de l insuline Les propriétés de l insuline Insuline et schémas
Insulinothérapie et diabète de type 1 Introduction: la molécule d insuline L instauration de l insulinothérapie Dispositif d administration de l insuline Les propriétés de l insuline Insuline et schémas
Evaluation des performances de programmes parallèles haut niveau à base de squelettes
 Evaluation des performances de programmes parallèles haut niveau à base de squelettes Enhancing the Performance Predictability of Grid Applications with Patterns and Process Algebras A. Benoit, M. Cole,
Evaluation des performances de programmes parallèles haut niveau à base de squelettes Enhancing the Performance Predictability of Grid Applications with Patterns and Process Algebras A. Benoit, M. Cole,
données en connaissance et en actions?
 1 Partie 2 : Présentation de la plateforme SPSS Modeler : Comment transformer vos données en connaissance et en actions? SPSS Modeler : l atelier de data mining Large gamme de techniques d analyse (algorithmes)
1 Partie 2 : Présentation de la plateforme SPSS Modeler : Comment transformer vos données en connaissance et en actions? SPSS Modeler : l atelier de data mining Large gamme de techniques d analyse (algorithmes)
Initiation à LabView : Les exemples d applications :
 Initiation à LabView : Les exemples d applications : c) Type de variables : Créer un programme : Exemple 1 : Calcul de c= 2(a+b)(a-3b) ou a, b et c seront des réels. «Exemple1» nom du programme : «Exemple
Initiation à LabView : Les exemples d applications : c) Type de variables : Créer un programme : Exemple 1 : Calcul de c= 2(a+b)(a-3b) ou a, b et c seront des réels. «Exemple1» nom du programme : «Exemple
Jean-Philippe Préaux http://www.i2m.univ-amu.fr/~preaux
 Colonies de fourmis Comment procèdent les colonies de fourmi pour déterminer un chemin presque géodésique de la fourmilière à un stock de nourriture? Les premières fourmis se déplacent au hasard. Les fourmis
Colonies de fourmis Comment procèdent les colonies de fourmi pour déterminer un chemin presque géodésique de la fourmilière à un stock de nourriture? Les premières fourmis se déplacent au hasard. Les fourmis
www.habefast.ch contact@habefast.ch Agence web en Suisse romande CH-1260 Nyon 022 362 25 70
 1 By Agence web en Suisse romande 2 Qu est ce qu est le SEO? Le référencement naturel ou SEO (Search Engine Optimisation) est une pratique qui, comme son nom l indique, permet d optimiser un site web pour
1 By Agence web en Suisse romande 2 Qu est ce qu est le SEO? Le référencement naturel ou SEO (Search Engine Optimisation) est une pratique qui, comme son nom l indique, permet d optimiser un site web pour
Qualité du logiciel: Méthodes de test
 Qualité du logiciel: Méthodes de test Matthieu Amiguet 2004 2005 Analyse statique de code Analyse statique de code Étudier le programme source sans exécution Généralement réalisée avant les tests d exécution
Qualité du logiciel: Méthodes de test Matthieu Amiguet 2004 2005 Analyse statique de code Analyse statique de code Étudier le programme source sans exécution Généralement réalisée avant les tests d exécution
Journée SITG, Genève 15 octobre 2013. Nicolas Lachance-Bernard M.ATDR Doctorant, Laboratoire de systèmes d information géographique
 Monitorint spatio-temporel intégré de la mobilité urbaine Monitoring spatio-temporel de l ADN urbain Une réponse aux défis, problèmes, enjeux et risques des milieux urbains Nicolas Lachance-Bernard M.ATDR
Monitorint spatio-temporel intégré de la mobilité urbaine Monitoring spatio-temporel de l ADN urbain Une réponse aux défis, problèmes, enjeux et risques des milieux urbains Nicolas Lachance-Bernard M.ATDR
Tests de comparaison de moyennes. Dr Sahar BAYAT MASTER 1 année 2009-2010 UE «Introduction à la biostatistique»
 Tests de comparaison de moyennes Dr Sahar BAYAT MASTER 1 année 2009-2010 UE «Introduction à la biostatistique» Test de Z ou de l écart réduit Le test de Z : comparer des paramètres en testant leurs différences
Tests de comparaison de moyennes Dr Sahar BAYAT MASTER 1 année 2009-2010 UE «Introduction à la biostatistique» Test de Z ou de l écart réduit Le test de Z : comparer des paramètres en testant leurs différences
1 Recherche en table par balayage
 1 Recherche en table par balayage 1.1 Problème de la recherche en table Une table désigne une liste ou un tableau d éléments. Le problème de la recherche en table est celui de la recherche d un élément
1 Recherche en table par balayage 1.1 Problème de la recherche en table Une table désigne une liste ou un tableau d éléments. Le problème de la recherche en table est celui de la recherche d un élément
Autoroute A16. Système de Repérage de Base (SRB) - Localisation des Points de repère (PR) A16- A16+
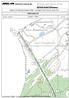 01 / 24 0 0!( 10 10 20 20 02 / 24 20 20 30 30 40 40 Système de Repérage de Base (SRB) - Localisation des Points de repère (PR) 03 / 24 40 40 50 50 60 60 60 60 04 / 24 70 70 80 80 80 80 Système de Repérage
01 / 24 0 0!( 10 10 20 20 02 / 24 20 20 30 30 40 40 Système de Repérage de Base (SRB) - Localisation des Points de repère (PR) 03 / 24 40 40 50 50 60 60 60 60 04 / 24 70 70 80 80 80 80 Système de Repérage
Chapitre V : La gestion de la mémoire. Hiérarchie de mémoires Objectifs Méthodes d'allocation Simulation de mémoire virtuelle Le mapping
 Chapitre V : La gestion de la mémoire Hiérarchie de mémoires Objectifs Méthodes d'allocation Simulation de mémoire virtuelle Le mapping Introduction Plusieurs dizaines de processus doivent se partager
Chapitre V : La gestion de la mémoire Hiérarchie de mémoires Objectifs Méthodes d'allocation Simulation de mémoire virtuelle Le mapping Introduction Plusieurs dizaines de processus doivent se partager
Routage AODV. Languignon - Mathe - Palancher - Pierdet - Robache. 20 décembre 2007. Une implémentation de la RFC3561
 20 décembre 2007 Une implémentation de la RFC3561 Présentation du groupe - plan Présentation Cahier des charges 1 CDC 2 Archi 3 Algorithme 4 Mini Appli de base Implémentation dans AODV 5 Difficultées rencontrées
20 décembre 2007 Une implémentation de la RFC3561 Présentation du groupe - plan Présentation Cahier des charges 1 CDC 2 Archi 3 Algorithme 4 Mini Appli de base Implémentation dans AODV 5 Difficultées rencontrées
DNS Poisoning. Pollution de cache sur des serveurs DNS. Xavier Dalem, Adrien Kunysz, Louis Plair. 15 mars 2007. Université de Liège
 Pollution de cache sur des serveurs DNS Université de Liège 15 mars 2007 Table des matières 1 2 3 4 DNS : rappel utilisé pour associer des données à un nom de domaine pas forcément qu une adresse (MX,
Pollution de cache sur des serveurs DNS Université de Liège 15 mars 2007 Table des matières 1 2 3 4 DNS : rappel utilisé pour associer des données à un nom de domaine pas forcément qu une adresse (MX,
Détection et prise en charge de la résistance aux antirétroviraux
 Détection et prise en charge de la résistance aux antirétroviraux Jean Ruelle, PhD AIDS Reference Laboratory, UCLouvain, Bruxelles Corata 2011, Namur, 10 juin 2011 Laboratoires de référence SIDA (Belgique)
Détection et prise en charge de la résistance aux antirétroviraux Jean Ruelle, PhD AIDS Reference Laboratory, UCLouvain, Bruxelles Corata 2011, Namur, 10 juin 2011 Laboratoires de référence SIDA (Belgique)
Politique scientifique, Recherche et Développement
 Ambassade de France à Washington Service pour la Science et la Technologie 4101 Reservoir Road NW, Washington, DC 20007 Tél. : +1 202 944 6246 Mail : info@france-science.org URL : http://france-science.org
Ambassade de France à Washington Service pour la Science et la Technologie 4101 Reservoir Road NW, Washington, DC 20007 Tél. : +1 202 944 6246 Mail : info@france-science.org URL : http://france-science.org
L exclusion mutuelle distribuée
 L exclusion mutuelle distribuée L algorithme de L Amport L algorithme est basé sur 2 concepts : L estampillage des messages La distribution d une file d attente sur l ensemble des sites du système distribué
L exclusion mutuelle distribuée L algorithme de L Amport L algorithme est basé sur 2 concepts : L estampillage des messages La distribution d une file d attente sur l ensemble des sites du système distribué
TD de Biochimie 4 : Coloration.
 TD de Biochimie 4 : Coloration. Synthèse de l expérience 2 Les questions posées durant l expérience 2 Exposé sur les méthodes de coloration des molécules : Générique Spécifique Autres Questions Pourquoi
TD de Biochimie 4 : Coloration. Synthèse de l expérience 2 Les questions posées durant l expérience 2 Exposé sur les méthodes de coloration des molécules : Générique Spécifique Autres Questions Pourquoi
Principes d implémentation des métaheuristiques
 Chapitre 2 Principes d implémentation des métaheuristiques Éric D. Taillard 1 2.1 Introduction Les métaheuristiques ont changé radicalement l élaboration d heuristiques : alors que l on commençait par
Chapitre 2 Principes d implémentation des métaheuristiques Éric D. Taillard 1 2.1 Introduction Les métaheuristiques ont changé radicalement l élaboration d heuristiques : alors que l on commençait par
Prédiction de la structure d une
 Prédiction de la structure d une protéine Soluscience Guillaume Chakroun guillaume chakroun@hotmail.com Copyright c 2004 Guillaume Chakroun TABLE DES MATIÈRES Table des matières 1 Les structures protéiques
Prédiction de la structure d une protéine Soluscience Guillaume Chakroun guillaume chakroun@hotmail.com Copyright c 2004 Guillaume Chakroun TABLE DES MATIÈRES Table des matières 1 Les structures protéiques
Circulaire 2015/1 «Comptabilité banques»
 Foire aux questions (FAQ) Circulaire 2015/1 «Comptabilité banques» (Dernière modification : 22 juillet 2015) 1. Comment faut-il traiter les provisions, constituées initialement pour des limites de crédit
Foire aux questions (FAQ) Circulaire 2015/1 «Comptabilité banques» (Dernière modification : 22 juillet 2015) 1. Comment faut-il traiter les provisions, constituées initialement pour des limites de crédit
Factorisation d entiers (première partie)
 Factorisation d entiers ÉCOLE DE THEORIE DES NOMBRES 0 Factorisation d entiers (première partie) Francesco Pappalardi Théorie des nombres et algorithmique 22 novembre, Bamako (Mali) Factorisation d entiers
Factorisation d entiers ÉCOLE DE THEORIE DES NOMBRES 0 Factorisation d entiers (première partie) Francesco Pappalardi Théorie des nombres et algorithmique 22 novembre, Bamako (Mali) Factorisation d entiers
T. Gasc 1,2,3, F. De Vuyst 1, R. Motte 3, M. Peybernes 4, R. Poncet 5
 Modélisation de la performance et optimisation d un algorithme hydrodynamique de type Lagrange-Projection sur processeurs multi-cœurs T. Gasc 1,2,3, F. De Vuyst 1, R. Motte 3, M. Peybernes 4, R. Poncet
Modélisation de la performance et optimisation d un algorithme hydrodynamique de type Lagrange-Projection sur processeurs multi-cœurs T. Gasc 1,2,3, F. De Vuyst 1, R. Motte 3, M. Peybernes 4, R. Poncet
Analyse de performance, monitoring
 Analyse de performance, monitoring Plan Principes de profilage Projet TPTP dans Eclipse Utilisation des profiling tools de TPTP Philippe Collet Master 1 Informatique 2009-2010 http://deptinfo.unice.fr/twiki/bin/view/minfo/gl
Analyse de performance, monitoring Plan Principes de profilage Projet TPTP dans Eclipse Utilisation des profiling tools de TPTP Philippe Collet Master 1 Informatique 2009-2010 http://deptinfo.unice.fr/twiki/bin/view/minfo/gl
Corrigé des TD 1 à 5
 Corrigé des TD 1 à 5 1 Premier Contact 1.1 Somme des n premiers entiers 1 (* Somme des n premiers entiers *) 2 program somme_entiers; n, i, somme: integer; 8 (* saisie du nombre n *) write( Saisissez un
Corrigé des TD 1 à 5 1 Premier Contact 1.1 Somme des n premiers entiers 1 (* Somme des n premiers entiers *) 2 program somme_entiers; n, i, somme: integer; 8 (* saisie du nombre n *) write( Saisissez un
Baccalauréat ES/L Amérique du Sud 21 novembre 2013
 Baccalauréat ES/L Amérique du Sud 21 novembre 2013 A. P. M. E. P. EXERCICE 1 Commun à tous les candidats 5 points Une entreprise informatique produit et vend des clés USB. La vente de ces clés est réalisée
Baccalauréat ES/L Amérique du Sud 21 novembre 2013 A. P. M. E. P. EXERCICE 1 Commun à tous les candidats 5 points Une entreprise informatique produit et vend des clés USB. La vente de ces clés est réalisée
Manuel des bonnes pratiques avec CATIA V.5
 2009 Manuel des bonnes pratiques avec CATIA V.5 Pour aller plus loin dans la maîtrise de l outil CAO Ce document a pour objectif de donner des méthodologies et règles de travail qui permettent de mener
2009 Manuel des bonnes pratiques avec CATIA V.5 Pour aller plus loin dans la maîtrise de l outil CAO Ce document a pour objectif de donner des méthodologies et règles de travail qui permettent de mener
Module BDR Master d Informatique (SAR)
 Module BDR Master d Informatique (SAR) Cours 9- Transactions réparties Anne Doucet Anne.Doucet@lip6.fr Transactions réparties Gestion de transactions Transactions dans un système réparti Protocoles de
Module BDR Master d Informatique (SAR) Cours 9- Transactions réparties Anne Doucet Anne.Doucet@lip6.fr Transactions réparties Gestion de transactions Transactions dans un système réparti Protocoles de
La mise en commun. (D après Michel Barlow, Le travail en groupes des élèves, Paris, A. Colin, 1993, pp. 38-44)
 La mise en commun (D après Michel Barlow, Le travail en groupes des élèves, Paris, A. Colin, 1993, pp. 38-44) 1. Définition et buts. Après le travail en groupe d élèves, la mise en commun permet de «reconstituer
La mise en commun (D après Michel Barlow, Le travail en groupes des élèves, Paris, A. Colin, 1993, pp. 38-44) 1. Définition et buts. Après le travail en groupe d élèves, la mise en commun permet de «reconstituer
SOCLE COMMUN - La Compétence 3 Les principaux éléments de mathématiques et la culture scientifique et technologique
 SOCLE COMMUN - La Compétence 3 Les principaux éléments de mathématiques et la culture scientifique et technologique DOMAINE P3.C3.D1. Pratiquer une démarche scientifique et technologique, résoudre des
SOCLE COMMUN - La Compétence 3 Les principaux éléments de mathématiques et la culture scientifique et technologique DOMAINE P3.C3.D1. Pratiquer une démarche scientifique et technologique, résoudre des
Samuel Bassetto 04/2010
 Industrialisation Lean manufacturing 4.2 Réalisé avec V. FIGENWALD - SIEMENS Samuel Bassetto 04/2010 Plan de la partie 2 : Vers une production Lean 1. Valeur Ajoutée et Gaspillages Muda walk 2. Temps de
Industrialisation Lean manufacturing 4.2 Réalisé avec V. FIGENWALD - SIEMENS Samuel Bassetto 04/2010 Plan de la partie 2 : Vers une production Lean 1. Valeur Ajoutée et Gaspillages Muda walk 2. Temps de
Figure 3.1- Lancement du Gambit
 3.1. Introduction Le logiciel Gambit est un mailleur 2D/3D; pré-processeur qui permet de mailler des domaines de géométrie d un problème de CFD (Computational Fluid Dynamics).Il génère des fichiers*.msh
3.1. Introduction Le logiciel Gambit est un mailleur 2D/3D; pré-processeur qui permet de mailler des domaines de géométrie d un problème de CFD (Computational Fluid Dynamics).Il génère des fichiers*.msh
Le ranking de Augure Influencers La méthodologie AIR en détails
 Le ranking de Augure Influencers La méthodologie AIR en détails V1.0 Octobre 2014 Oualid Abderrazek Product Marketing Sommaire 1. Contexte...3 2. L algorithme...3 a. Exposition...4 b. Echo...4 c. Niveau
Le ranking de Augure Influencers La méthodologie AIR en détails V1.0 Octobre 2014 Oualid Abderrazek Product Marketing Sommaire 1. Contexte...3 2. L algorithme...3 a. Exposition...4 b. Echo...4 c. Niveau
Présentation du PL/SQL
 I Présentation du PL/ Copyright Oracle Corporation, 1998. All rights reserved. Objectifs du Cours A la fin de ce chapitre, vous saurez : Décrire l intéret du PL/ Décrire l utilisation du PL/ pour le développeur
I Présentation du PL/ Copyright Oracle Corporation, 1998. All rights reserved. Objectifs du Cours A la fin de ce chapitre, vous saurez : Décrire l intéret du PL/ Décrire l utilisation du PL/ pour le développeur
Chapitre 3. Quelques fonctions usuelles. 1 Fonctions logarithme et exponentielle. 1.1 La fonction logarithme
 Chapitre 3 Quelques fonctions usuelles 1 Fonctions logarithme et eponentielle 1.1 La fonction logarithme Définition 1.1 La fonction 7! 1/ est continue sur ]0, +1[. Elle admet donc des primitives sur cet
Chapitre 3 Quelques fonctions usuelles 1 Fonctions logarithme et eponentielle 1.1 La fonction logarithme Définition 1.1 La fonction 7! 1/ est continue sur ]0, +1[. Elle admet donc des primitives sur cet
L analyse d images regroupe plusieurs disciplines que l on classe en deux catégories :
 La vision nous permet de percevoir et d interpreter le monde qui nous entoure. La vision artificielle a pour but de reproduire certaines fonctionnalités de la vision humaine au travers de l analyse d images.
La vision nous permet de percevoir et d interpreter le monde qui nous entoure. La vision artificielle a pour but de reproduire certaines fonctionnalités de la vision humaine au travers de l analyse d images.
Industrialiser la chaîne complète de fabrication 1ère partie - Les bénéfices de la solution logicielle IBM VisualAge Pacbase / Rational
 IBM Software Group Industrialiser la chaîne complète de fabrication 1ère partie - Les bénéfices de la solution logicielle IBM VisualAge Pacbase / Rational Fernard Bonaguidi fernand.bonaguidi@fr.ibm.com
IBM Software Group Industrialiser la chaîne complète de fabrication 1ère partie - Les bénéfices de la solution logicielle IBM VisualAge Pacbase / Rational Fernard Bonaguidi fernand.bonaguidi@fr.ibm.com
Qu est ce qu un réseau social. CNAM Séminaire de Statistiques Appliquées 13/11/2013. F.Soulié Fogelman 1. Utilisation des réseaux sociaux pour le
 Qui je suis Innovation Utilisation des réseaux sociaux pour le data mining Business & Decision Françoise Soulié Fogelman francoise.soulie@outlook.com Atos KDD_US CNAM Séminaire de Statistique appliquée
Qui je suis Innovation Utilisation des réseaux sociaux pour le data mining Business & Decision Françoise Soulié Fogelman francoise.soulie@outlook.com Atos KDD_US CNAM Séminaire de Statistique appliquée
Baccalauréat ES Antilles Guyane 12 septembre 2014 Corrigé
 Baccalauréat ES Antilles Guyane 12 septembre 2014 Corrigé EXERCICE 1 5 points Commun à tous les candidats 1. Réponse c : ln(10)+2 ln ( 10e 2) = ln(10)+ln ( e 2) = ln(10)+2 2. Réponse b : n 13 0,7 n 0,01
Baccalauréat ES Antilles Guyane 12 septembre 2014 Corrigé EXERCICE 1 5 points Commun à tous les candidats 1. Réponse c : ln(10)+2 ln ( 10e 2) = ln(10)+ln ( e 2) = ln(10)+2 2. Réponse b : n 13 0,7 n 0,01
Master IAD Module PS. Reconnaissance de la parole (suite) Alignement temporel et Programmation dynamique. Gaël RICHARD Février 2008
 Master IAD Module PS Reconnaissance de la parole (suite) Alignement temporel et Programmation dynamique Gaël RICHARD Février 2008 1 Reconnaissance de la parole Introduction Approches pour la reconnaissance
Master IAD Module PS Reconnaissance de la parole (suite) Alignement temporel et Programmation dynamique Gaël RICHARD Février 2008 1 Reconnaissance de la parole Introduction Approches pour la reconnaissance
Etude comparative de différents motifs utilisés pour le lancé de rayon
 Etude comparative de différents motifs utilisés pour le lancé de rayon Alexandre Bonhomme Université de Montréal 1 Introduction Au cours des dernières années les processeurs ont vu leurs capacités de calcul
Etude comparative de différents motifs utilisés pour le lancé de rayon Alexandre Bonhomme Université de Montréal 1 Introduction Au cours des dernières années les processeurs ont vu leurs capacités de calcul
Logiciel SCRATCH FICHE 02
 1. Reprise de la fiche 1: 1.1. Programme Figure : Logiciel SCRATCH FICHE 02 SANS ORDINATEUR : Dessiner à droite le dessin que donnera l'exécution de ce programme : Unité : 50 pas : Remarque : vous devez
1. Reprise de la fiche 1: 1.1. Programme Figure : Logiciel SCRATCH FICHE 02 SANS ORDINATEUR : Dessiner à droite le dessin que donnera l'exécution de ce programme : Unité : 50 pas : Remarque : vous devez
Parcours en deuxième année
 Parcours en deuxième année Unités d Enseignement (UE) ECTS Ingénierie des réseaux haut 4 débit Sécurité des réseaux et 4 télécoms Réseaux mobiles et sans fil 4 Réseaux télécoms et 4 convergence IP Infrastructure
Parcours en deuxième année Unités d Enseignement (UE) ECTS Ingénierie des réseaux haut 4 débit Sécurité des réseaux et 4 télécoms Réseaux mobiles et sans fil 4 Réseaux télécoms et 4 convergence IP Infrastructure
Application 1- VBA : Test de comportements d'investissements
 Application 1- VBA : Test de comportements d'investissements Notions mobilisées Chapitres 1 à 5 du cours - Exemple de récupération de cours en ligne 1ère approche des objets (feuilles et classeurs). Corps
Application 1- VBA : Test de comportements d'investissements Notions mobilisées Chapitres 1 à 5 du cours - Exemple de récupération de cours en ligne 1ère approche des objets (feuilles et classeurs). Corps
DEVELOPPEMENT ET MAINTENANCE DE LOGICIEL: OUTIL DE PILOTAGE
 DEVELOPPEMENT ET MAINTENANCE DE LOGICIEL: OUTIL DE PILOTAGE Développement et maintenance de logiciel Automne 2006 François-Xavier RIU Thomas POUPART Seng LAO Zhe WU SOMMAIRE Introduction Introduction INTRODUCTION
DEVELOPPEMENT ET MAINTENANCE DE LOGICIEL: OUTIL DE PILOTAGE Développement et maintenance de logiciel Automne 2006 François-Xavier RIU Thomas POUPART Seng LAO Zhe WU SOMMAIRE Introduction Introduction INTRODUCTION
FORMATION CONTINUE SUR L UTILISATION D EXCEL DANS L ENSEIGNEMENT Expérience de l E.N.S de Tétouan (Maroc)
 87 FORMATION CONTINUE SUR L UTILISATION D EXCEL DANS L ENSEIGNEMENT Expérience de l E.N.S de Tétouan (Maroc) Dans le cadre de la réforme pédagogique et de l intérêt que porte le Ministère de l Éducation
87 FORMATION CONTINUE SUR L UTILISATION D EXCEL DANS L ENSEIGNEMENT Expérience de l E.N.S de Tétouan (Maroc) Dans le cadre de la réforme pédagogique et de l intérêt que porte le Ministère de l Éducation
Le Processus RUP. H. Kadima. Tester. Analyst. Performance Engineer. Database Administrator. Release Engineer. Project Leader. Designer / Developer
 Le Processus RUP Database Administrator Project Leader H. Kadima Performance Engineer Release Engineer Analyst Designer / Developer Tester Table des matières 1. De l artisanat à l industrialisation de
Le Processus RUP Database Administrator Project Leader H. Kadima Performance Engineer Release Engineer Analyst Designer / Developer Tester Table des matières 1. De l artisanat à l industrialisation de
Séquence 6. Mais ces espèces pour autant ne sont pas identiques et parfois d ailleurs ne se ressemblent pas vraiment.
 Sommaire Séquence 6 Nous avons vu dans les séances précédentes qu au cours des temps géologiques des espèces différentes se sont succédé, leur apparition et leur disparition étant le résultat de modifications
Sommaire Séquence 6 Nous avons vu dans les séances précédentes qu au cours des temps géologiques des espèces différentes se sont succédé, leur apparition et leur disparition étant le résultat de modifications
LES OUTILS D ALIMENTATION DU REFERENTIEL DE DB-MAIN
 LES OUTILS D ALIMENTATION DU REFERENTIEL DE DB-MAIN Les contenues de ce document sont la propriété exclusive de la société REVER. Ils ne sont transmis qu à titre d information et ne peuvent en aucun cas
LES OUTILS D ALIMENTATION DU REFERENTIEL DE DB-MAIN Les contenues de ce document sont la propriété exclusive de la société REVER. Ils ne sont transmis qu à titre d information et ne peuvent en aucun cas
Table des matières PRESENTATION DU LANGAGE DS2 ET DE SES APPLICATIONS. Introduction
 PRESENTATION DU LANGAGE DS2 ET DE SES APPLICATIONS Depuis SAS 9.2 TS2M3, SAS propose un nouveau langage de programmation permettant de créer et gérer des tables SAS : le DS2 («Data Step 2»). Ces nouveautés
PRESENTATION DU LANGAGE DS2 ET DE SES APPLICATIONS Depuis SAS 9.2 TS2M3, SAS propose un nouveau langage de programmation permettant de créer et gérer des tables SAS : le DS2 («Data Step 2»). Ces nouveautés
8 Certifications Minergie
 8 Chapitre 8 Être Minergie, est-ce aussi être «autrement»? Pour de nombreux acteurs du marché immobilier, un label de durabilité devrait s accompagner d une appréciation de la valeur de leur immeuble,
8 Chapitre 8 Être Minergie, est-ce aussi être «autrement»? Pour de nombreux acteurs du marché immobilier, un label de durabilité devrait s accompagner d une appréciation de la valeur de leur immeuble,
Analyse des trajectoires acceptables en approche de virage assistance aux conducteurs
 DIVAS Analyse des trajectoires acceptables en approche de virage assistance aux conducteurs N 3.C.1 Décembre 2008 Projet financé par l Agence Nationale de la Recherche Responsable : S. Espié Projet ANR
DIVAS Analyse des trajectoires acceptables en approche de virage assistance aux conducteurs N 3.C.1 Décembre 2008 Projet financé par l Agence Nationale de la Recherche Responsable : S. Espié Projet ANR
Introduction à la B.I. Avec SQL Server 2008
 Introduction à la B.I. Avec SQL Server 2008 Version 1.0 VALENTIN Pauline 2 Introduction à la B.I. avec SQL Server 2008 Sommaire 1 Présentation de la B.I. et SQL Server 2008... 3 1.1 Présentation rapide
Introduction à la B.I. Avec SQL Server 2008 Version 1.0 VALENTIN Pauline 2 Introduction à la B.I. avec SQL Server 2008 Sommaire 1 Présentation de la B.I. et SQL Server 2008... 3 1.1 Présentation rapide
Définitions. Numéro à préciser. (Durée : )
 Numéro à préciser (Durée : ) On étudie dans ce problème l ordre lexicographique pour les mots sur un alphabet fini et plusieurs constructions des cycles de De Bruijn. Les trois parties sont largement indépendantes.
Numéro à préciser (Durée : ) On étudie dans ce problème l ordre lexicographique pour les mots sur un alphabet fini et plusieurs constructions des cycles de De Bruijn. Les trois parties sont largement indépendantes.
BACCALAURÉAT GÉNÉRAL SESSION 2012 OBLIGATOIRE MATHÉMATIQUES. Série S. Durée de l épreuve : 4 heures Coefficient : 7 ENSEIGNEMENT OBLIGATOIRE
 BACCALAURÉAT GÉNÉRAL SESSION 2012 MATHÉMATIQUES Série S Durée de l épreuve : 4 heures Coefficient : 7 ENSEIGNEMENT OBLIGATOIRE Les calculatrices électroniques de poche sont autorisées, conformément à la
BACCALAURÉAT GÉNÉRAL SESSION 2012 MATHÉMATIQUES Série S Durée de l épreuve : 4 heures Coefficient : 7 ENSEIGNEMENT OBLIGATOIRE Les calculatrices électroniques de poche sont autorisées, conformément à la
Introduction au Data-Mining
 Introduction au Data-Mining Alain Rakotomamonjy - Gilles Gasso. INSA Rouen -Département ASI Laboratoire PSI Introduction au Data-Mining p. 1/25 Data-Mining : Kèkecé? Traduction : Fouille de données. Terme
Introduction au Data-Mining Alain Rakotomamonjy - Gilles Gasso. INSA Rouen -Département ASI Laboratoire PSI Introduction au Data-Mining p. 1/25 Data-Mining : Kèkecé? Traduction : Fouille de données. Terme
Chaine de transmission
 Chaine de transmission Chaine de transmission 1. analogiques à l origine 2. convertis en signaux binaires Échantillonnage + quantification + codage 3. brassage des signaux binaires Multiplexage 4. séparation
Chaine de transmission Chaine de transmission 1. analogiques à l origine 2. convertis en signaux binaires Échantillonnage + quantification + codage 3. brassage des signaux binaires Multiplexage 4. séparation
Teste et mesure vos réseaux et vos applicatifs en toute indépendance
 Teste et mesure vos réseaux et vos applicatifs en toute indépendance 2013 J3TEL en quelques minutes Groupe HBG en bref : Siège social à Paris 1100 employés dans 6 pays 150 M d de CA en 2012 Des activités
Teste et mesure vos réseaux et vos applicatifs en toute indépendance 2013 J3TEL en quelques minutes Groupe HBG en bref : Siège social à Paris 1100 employés dans 6 pays 150 M d de CA en 2012 Des activités
