MÉMOIRE. Bases de données et outils d analyse pour la génomique bactérienne
|
|
|
- Beatrice Prudhomme
- il y a 10 ans
- Total affichages :
Transcription
1 MÉMOIRE présenté devant l Université Claude Bernard LYON 1 pour l obtention de L HABILITATION À DIRIGER DES RECHERCHES par Guy PERRIÈRE soutenance prévue le 27 juin 2000 Bases de données et outils d analyse pour la génomique bactérienne Jury : MM. A. BAIROCH A.J. COZZONE P. DESSEN C. GAUTIER D. KAHN B. LABEDAN J.-L. RISLER Laboratoire de Biométrie et Biologie Évolutive, UMR CNRS n 5558 Université Claude Bernard Lyon 1 43, bd. du 11 novembre Villeurbanne Cedx
2
3 TABLE DES MATIÈRES INTRODUCTION...1 I. BANQUES DE DONNÉES LES BANQUES GÉNÉRALISTES BANQUES DE SÉQUENCES NUCLÉOTIDIQUES DONNÉES FORMAT DE STOCKAGE DISTRIBUTION ET MISE À JOUR BANQUES DE SÉQUENCES PROTÉIQUES TREMBL ET GENPEPT SWISS-PROT PIR AUTRES BANQUES INTERÊT ET LIMITES DES BANQUES GÉNÉRALISTES BANQUES SPÉCIALISÉES BANQUES THÉMATIQUES STRUCTURES MOLÉCULAIRES SÉQUENCES ET STRUCTURES DE RNA FAMILLES DE GÈNES DOMAINES PROTÉIQUES SIGNATURES PEPTIDIQUES BANQUES GÉNOMIQUES PROCARYOTES SACCHAROMYCES CEREVISIAE DROSOPHILA MELANOGASTER HOMO SAPIENS AUTRES SYSTÈMES D INTERROGATION LA PRÉDOMINANCE D INTERNET ACNUC SRS ENTREZ ACEDB...27 II. DÉVELOPPEMENTS INFORMATIQUES LE SERVEUR DU PBIL MISE EN PLACE ACCÈS AUX BANQUES RÉCUPÉRATION DES SÉQUENCES RECHERCHE DE SIMILARITÉS ET ALIGNEMENTS AUTRES FONCTIONNALITÉS ANALYSE MULTIVARIÉE ANALYSE FACTORIELLE DES CORRESPONDANCES ANALYSE DISCRIMINANTE DES CORRESPONDANCES ANALYSE EN COORDONNÉES PRINCIPALES...36 i
4 III APPLICATIONS ASSOCIÉES BILAN DE L UTILISATION DU SERVEUR EMGLIB MOTIVATION CONTENU DE LA BANQUE SÉQUENCES USAGE DU CODE INFORMATIONS COMPLÉMENTAIRES ACCÈS AUX DONNÉES PERSPECTIVES HOBACGEN MOTIVATION CONSTRUCTION DE LA BANQUE ACCÈS AUX DONNÉES CLIENT JAVA SERVEUR ACCÈS PAR LE WEB COMPARAISON AVEC D AUTRES SYSTÈMES ÉVOLUTIONS FUTURES...56 RÉSULTATS BIOLOGIQUES LOCALISATION SUBCELLULAIRE DES PROTÉINES MATÉRIEL ET MÉTHODES RÉSULTATS DISCUSSION RÉPARTITION ASYMÉTRIQUE DES GÈNES MATÉRIEL ET MÉTHODES RÉSULTATS DISCUSSION VARIATIONS DE COMPOSITION EN BASES ET TRANSFERTS HORIZONTAUX DÉTECTION PAR APPROCHES INTRINSÈQUES MATÉRIEL ET MÉTHODES RÉSULTATS DISCUSSION...69 CONCLUSIONS PERSPECTIVES BANQUES DE DONNÉES DE GÈNES HOMOLOGUES TRANSFERTS HORIZONTAUX GÉNOMIQUE DES BACTÉRIES ENDOCYTOBIOTIQUES...73 RÉFÉRENCES BIBLIOGRAPHIQUES...75 ANNEXE : TITRES ET TRAVAUX...89 ii
5 INTRODUCTION INTRODUCTION Au cours des cinq dernières années, la bioinformatique moléculaire a connu un essor extraordinaire. Cet essor est bien sûr lié à l aboutissement de nombreux projets de séquençage, projets ayant conduit à l arrivée d énormes quantités de données dont il faut maintenant tirer le plus d informations possibles. Si, dans un premier temps, les génomes séquencés étaient ceux de procaryotes, nous arrivons maintenant au stade où des génomes d eucaryotes pluricellulaires commencent à être disponibles : Caenorhabditis elegans, Drosophila melanogaster, et bientôt l Homme. Cependant, même dans le cas d organismes au génome compact comme les bactéries et les archées, les quantités de données brutes disponibles sont déjà trop importantes pour pouvoir être analysées manuellement (en particulier pour tout ce qui concerne l annotation des séquences). L outil informatique est donc désormais considéré comme un complément indispensable de la biologie moléculaire expérimentale. Ce développement de la bioinformatique a été rendu possible par les énormes progrès réalisés au niveau des capacités de calcul et de stockage des ordinateurs. Sans ces progrès, il n eût pas été envisageable de construire des banques capables de manipuler l intégralité des séquences biologiques publiées ou de développer des logiciels susceptibles d effectuer des traitements sur de très larges sous-ensembles de ces banques. De façon concommitante, au fur et à mesure que les capacités des ordinateurs progressaient, les possibilités d accès aux données et aux programmes se voyaient multipliées du fait du développement d Internet, en particulier grâce au World-Wide Web. Comme ce protocole permettait de construire des interfaces à des programmes situés sur des serveurs distants, il a suffi de deux années après son introduction pour qu il soit possible d utiliser pratiquement n importe quelle banque ou logiciel d analyse des séquences sur un serveur Web dédié. Cette possibilité supprima un des principaux facteurs limitant l utilisation de ces programmes : la nécessité d effectuer une installation locale ou d avoir un compte sur un centre serveur. Avec la disponibilité d un nombre considérable de séquences appartenant à des taxons parfois fort lointains du point de vue phylogénétique, une branche de la bioinformatique a alors connu un développement particulier : la génomique comparative. En effectuant des comparaisons de séquences (par exemple au moyen d alignements) entre plusieurs organismes, il est possible d enrichir les connaissances que l on a sur un gène ou un groupe de gènes. C est en utilisant cette approche que sont le plus souvent effectuées des assignations de fonction ainsi que de nombreuses études ayant trait à la phylogénie moléculaire. Du fait de l importance de cette approche pour tout ce qui touche à l analyse des génomes microbiens, la revue Microbial and Comparative Genomics a d ailleurs été récemment créée. Cependant, il ne faudrait pas penser que la bioinformatique s est mise à exister depuis cinq ou six ans seulement. Mon recrutement au CNRS, en 1992, s est fait sur un poste flêché ayant trait au développement de banques de données en biologie moléculaire. Par ailleurs, l équipe «Biométrie Moléculaire, Évolution et Structure de Génomes» du Laboratoire de Biométrie et Biologie Évolutive est impliquée dans ce - 1 -
6 INTRODUCTION domaine depuis plus de vingt ans! Dès mon intégration dans cette équipe, je me suis intéressé au développement d outils pour l analyse des génomes bactériens. Plus particulièrement j ai participé à la construction de plusieurs banques de données de séquences et à la conception de logiciels permettant d accéder à ces banques et à en traiter les données. Mon travail ne s est cependant pas limité à une activité de génie logiciel puisque j ai été le premier utilisateur de ces outils afin d obtenir des résultats biologiques nouveaux. Ce mémoire se présente donc comme un bilan de mes travaux récents (depuis 1995) dans ce domaine. Ne sont donc pas abordés les développements plus anciens que j ai pu effectuer dans la continuité de ma thèse sur la base de connaissance ColiGene ( ), ou pendant mon année de mise à disposition au Japon sur la banque de séquences NRSub ( ). Le plan de cet ouvrage est le suivant : le premier chapitre est consacré à un «état de l art» dans le domaine des banques de séquences. Il décrit tout d abord les principales collections généralistes de séquences nucléotidiques et protéiques puis s attache à montrer quelles sont leurs limites. Ensuite sont décrites quelques unes des banques spécialisées qui ont été développées dans le but de répondre à ces limitations. Ce chapitre se termine sur un aperçu des différents systèmes d interrogation à la disposition des utilisateurs pour accéder aux banques. Le deuxième chapitre expose les différents développements informatiques auxquels j ai participé. Ces développements ayant été effectués en collaboration (ou à la suite de fortes interactions) avec d autres chercheurs de mon équipe ou d équipes extérieures. Ainsi le travail que j ai effectué sur le serveur du Pôle Bio-Informatique Lyonnais est typiquement un exemple de tâche impliquant la collaboration de plusieurs autres personnes. Parmi les autres outils à la construction desquels j ai participé, figurent la banque de séquences de génomes complets microbiens EMGLib et la banque de gènes homologues de procaryotes HOBACGEN. Le troisième chapitre présente les différents résultats biologiques que j ai pu obtenir avec les différents outils que j ai développés. Tout d abord est présenté un résultat sur la localisation subcellulaire des protéines de bactéries Gram négatives, résultat obtenu au moyen d une méthode d analyse multivariée d introduction relativement récente : l analyse discriminante des correspondances. Ensuite, est décrite une étude sur l asymétrie de localisation des gènes entre le brin direct et le brin retardé du chromosome bactérien. Enfin, je montre en quoi l existence de variations dans la composition en base des chromosomes bactériens peut conduire à une surestimation du nombre de gènes prédits comme ayant été obtenus par transfert horizontal. Pour conclure, plusieurs projets de recherche en continuité avec les travaux exposés dans cet ouvrage sont présentés. Un premier groupe de projets concerne la construction de nouvelles banques utilisant le modèle de données et le système d interrogation d HOBACGEN. Une autre voie en cours d exploration est celle de la recherche de gènes obtenus par transfert horizontal chez plusieurs groupes d espèces bactériennes : des organismes «modèles», comme Escherichia coli, mais également des pathogènes humains ou végétaux. Le dernier projet concerne l étude de la génomique des bactéries endocytobiotiques des arthropodes, qu il s agisse de parasites du genre Buchnera ou de symbiotes comme Wolbachia
7 BANQUES DE DONNÉES I. BANQUES DE DONNÉES Depuis que les biologistes travaillent avec des séquences en grande quantité c est-àdire depuis le développement et la généralisation de l utilisation des méthodes rapides de séquençage la nécessité d organiser et d accéder aisément à ces données s est fait ressentir. Les premières banques de données en biologie moléculaire concernèrent les informations structurales sur les protéines, puis, très rapidement, les séquences protéiques et nucléotidiques. Il existe des banques généralistes, dans lesquelles sont stockées les séquences provenant de tous les organismes, et des banques spécialisées qui se consacrent plus particulièrement à un organisme ou à une thématique donnée. L existence de ces banques a nécessité le développement de systèmes d interrogation permettant d accéder aux données qu elles contiennent. Ce chapitre se propose donc de faire un tour d horizon de ces différents systèmes. 1. LES BANQUES GÉNÉRALISTES Cette section présente les principales collections généralistes de séquences nucléotidiques et protéiques ainsi que les centres de saisie qui leur sont associés. Nous verrons en particulier comment les données sont collectées et sous quel format elles sont stockées et rendues publiques. Par la suite, j exposerai les principales limitations de ces systèmes, limitations qui sont en partie responsables du développement des banques spécialisées. C est d ailleurs du fait de ces limitations que plusieurs banques spécialisées ont été développées dans notre équipe, certaines étant décrites plus en détail dans le deuxième chapitre BANQUES DE SÉQUENCES NUCLÉOTIDIQUES Il existe trois banques généralistes de séquences nucléotidiques publiquement accessibles de par le monde : l EMBL (Baker et al., 2000) en Europe, GenBank (Benson et al., 2000) aux États-Unis, et la DDBJ (Tateno et al., 2000) au Japon. La banque EMBL a été créée en 1980 à Heidelberg, et elle est maintenue depuis 1994 par l EBI (European Bioinformatic Institute), à Cambridge. GenBank a été mise en place en 1979 au LANL (Los Alamos National Laboratory), à Los Alamos. Depuis 1992 elle est maintenue au NCBI (National Center for Biotechnology Information), à Bethesda. Enfin, la DDBJ a débuté son activité en Cette banque a été créée et est toujours maintenue au NIG (National Institute of Genetics), à Mishima. Ces trois centres étant également en charge de la saisie et de la distribution des données DONNÉES Les données proviennent dans 95 % des cas de soumissions directes effectuées par les auteurs, soumissions réalisées désormais quasi-exclusivement par l intermédiaire du réseau Internet. En effet, la plupart des revues de biologie moléculaire n acceptent de - 3 -
8 CHAPITRE I publier des séquences que si celles-ci sont dotées d un numéro d accession fourni par les banques. Il convient donc, dès l obtention d une nouvelle séquence, de soumettre celle-ci au centre de saisie le plus proche géographiquement. Les 5 % de séquences restants sont extraits de la littérature scientifique (notamment les documents de brevets). Il est à noter que la collecte des séquences via les publications représente un travail long, difficile, beaucoup moins précis que les soumissions directes. Les séquences recueillies par les trois centres sont échangées quotidiennement, ceci de façon à obtenir un ensemble de données aussi complet et cohérent que possible. La conséquence de ceci est qu en pratique, ces trois banques n en font qu une car leur contenu est identique à plus de 99,9 %. Ceci amène régulièrement les responsables des centres de saisie à se poser la question de l utilité de la maintenance de trois banques différentes. Il existe ainsi depuis longtemps un projet de fusion d EMBL, de GenBank et de la DDBJ en un seul système. Depuis leur création au début des années 80, la taille de ces banques n a cessé de croître. Si l on regarde l allure de cette croissance, on constate qu elle peut être approximativement assimilée à une exponentielle pour les séquences nucléotidiques (Figure I.1). Par contre, le nombre de séquences protéiques croît beaucoup moins vite. L accélération constatée en 1995 correspond au développement des techniques de PCR (Polymerase Chain Reaction) et à l arrivée massive des EST (Expressed Sequence Tags) et des STS (Sequence-Tagged Sites), largement utilisés pour l étude des génomes eucaryotes (Venter, 1991). log(taille) 10 9,5 9 8,5 8 7,5 7 6,5 6 5,5 5 09/83 07/85 05/87 03/89 01/91 11/92 09/94 07/96 05/98 Date Figure I.1. Croissance en taille des banques nucléotidiques GenBank ( ) et EMBL ( ), et des banques protéiques PIR ( ) et SWISS-PROT ( ). Au moment où sont écrites ces lignes, les génomes complets de 22 bactéries, six archées, et trois eucaryotes (Saccharomyces cerevisiae, C. elegans et D. melanogaster) sont disponibles. Par ailleurs, c est d ores et déjà près de 75 % du génome humain qui est accessible. La base de données GOLD (Genome On Line Database) permet de suivre la progression des différents projets de séquençage en cours 1. C est ainsi qu il existe plus de 100 projets pour des organismes procaryotes (dont une majorité de bactéries pathogènes) et 31 pour des organismes eucaryotes
9 BANQUES DE DONNÉES FORMAT DE STOCKAGE EMBL, GenBank et la DDBJ sont distribuées par les centres sous la forme d un ensemble de fichiers plats (environ une centaine) regroupant les séquences en fonction de critères taxonomiques (procaryotes, virus, primates, etc.) ou de leur origine (brevets, EST et STS). À l intérieur de ces fichiers, chaque séquence est contenue dans une structure appelée «entrée», une entrée comprenant une quantité variable d informations liée à la séquence considérée (comme sa structure, son rôle biologique, ainsi que la mention de l organisme dont elle est issue). Les informations en question sont introduites au niveau de «champs» bien définis. Le format de stockage utilisé par EMBL est différent de celui utilisé conjointement par GenBank et la DDBJ. Cette différence ne porte que sur la façon de représenter les données et la philosophie générale des deux systèmes est la même. Le format utilisé par EMBL étant cependant plus simple à utiliser si l on se place dans la perspective d une structuration des données au moyen d un SGBD (Système de Gestion de Bases de Données). Dans ce format, les champs sont identifiés à l aide d un code à deux lettres localisé dans les deux premières colonnes du fichier (Figure I.2). Dans le cas de GenBank et de la DDBJ, ces champs sont indiqués par des identificateurs organisés sur deux niveaux, la séparation entre ces différents niveaux étant repèrable par une indentation différente. Des informations d ordre général sont disponibles au niveau de sept champs. Le champ ID (LOCUS, dans le cas de GenBank et de la DDBJ) correspond au nom de l entrée, son statut, la nature de la molécule séquencée et sa longueur. AC (ACCESSION) contient un ou plusieurs numéros d accession, uniques pour l ensemble des banques. SV (VERSION) reprend les numéros d accession du champ AC et en plus leur ajoute un numéro de version. À chaque modification de l entrée, ce numéro de version est incrémenté. DE (DEFINITION) contient ce que l on appelle la définition de la séquence. Il s agit de quelques lignes, fournies par les auteurs, décrivant sommairement le contenu de l entrée (noms des gènes, fonction des protéines pour lesquelles ils codent, etc.) DT est spécifique au format EMBL et contient la date de création de l entrée (première ligne) ainsi que la date de dernière modification (deuxième ligne). Une liste de mots-clés figure au niveau du champ KW (KEYWORDS). CC (COMMENT) permet l introduction de références croisées avec d autres banques ainsi que des commentaires. Ces commentaires peuvent être introduits par les auteurs, ou bien par les personnes en charge de la saisie des séquences dans les centres. La classification taxonomique de l organisme dont est issue la séquence est introduite au niveau de trois champs. OS (SOURCE) contient le nom de l espèce, OC (ORGANISM) sa position dans l arbre, et OG permet de préciser si la séquence provient d un organelle (mitochondrie ou chloroplaste) de l espèce considérée. La classification qui figure dans ces champs est fort ancienne et comporte de nombreuses inexactitudes, ceci alors que le NCBI propose sur son site Web un arbre des espèces beaucoup plus récent et complet 2. Un projet de migration des différentes banques généralistes vers cette taxonomie étant d ailleurs prévu. Les références bibliographiques nécessitent six champs. RN (REFERENCE) contient le numéro de la référence, RP spécifie quelle est la région de la séquence concernée par cette référence, RX (MEDLINE) renvoie au numéro d accession de cette référence dans la banque de données bibliographique Medline, RA (AUTHORS) contient les noms des auteurs, RT (TITLE) donne le titre de l article, et enfin, RL (JOURNAL) donne la référence proprement dite (nom du journal, volume, pagination et année de publication)
10 CHAPITRE I ID ECINTER standard; DNA; PRO; 2509 BP. XX AC M63497; X53729; XX SV M XX DT 28-MAR-1991 (Rel. 27, Created) DT 05-JUL-1999 (Rel. 60, Last updated, Version 7) XX DE E.coli intergenic region between iclr and acek genes. XX KW acek gene; iclr gene; isocitrate dehydrogenase kinase/phosphatase. XX OS Escherichia coli OC Bacteria; Proteobacteria; gamma subdivision; Enterobacteriaceae; OC Escherichia. XX RN [1] RP RX MEDLINE; RA Galinier A., Bleicher F., Negre D., Perriere G., Duclos B., Cozzone A.J., RA Cortay J.-C.; RT "Primary structure of the intergenic region between acek and iclr in the RT Escherichia coli chromosome"; RL Gene 97: (1991). XX CC SWISS-PROT; P11071; ACEK_ECOLI CC SWISS-PROT; P16528; ICLR_ECOLI CC SWISS-PROT; P23325; YJAC_ECOLI [ ] XX FH Key Location/Qualifiers FH FT source FT /db_xref="taxon:562" FT /organism="escherichia coli" FT /strain="k-12" [ ] FT CDS FT /codon_start=1 FT /db_xref="swiss-prot:p23325" FT /label=orf FT /transl_table=11 FT /protein_id="aaa " FT /translation="meilpqrsdtldkqeldlksdrkekefpriklngqcyfpgrpqnr FT IVCRHIAAQYINDIYQNVDYKPHQDDYSSAEKFLTHFNKKCKNQTLALVSSRPEGRCVA FT ACGDFGLVMKAYFDKMESNGISVMAAILLVDNHALTVRLRIKNTTEGCTHYVVSVYDPN [ ] FT GLFSAIQHKQQNVVETVYLALSDHARLFGFTAEDIMDFWQHKAPQKYSAFELAFEFGHR FT [ ] XX SQ [ ] // VIAELILNTLNKMAESFTQKSISPYRTLNLCLRRYA" Sequence 2509 BP; 825 A; 494 C; 459 G; 731 T; 0 other; aagcggcgaa ggaagtgacg ctggcgtacg gtggaatgcg ctgacttttt ctggcgggca 60 gaggcaattt ctgcccatca tacctgagtg gcaatagaat aagggtgtct gttaatcgca 120 ttgacgccaa aataacttaa tcgcattgac gccaaaataa cttaatgtca tacacttcac 180 taaataagat ggctgaaagc tttactcaaa aaagcatctc cccataccgt acgctaaatc 2460 tttgcctgcg ccgatacgca taaacatctt ccacatgccc ttcacgtat 2509 Figure I.2. Exemple d entrée EMBL. Le champ FT (FEATURES) contient des informations très variées sur la position et la nature des segments de séquence biologiquement significatifs (régions codantes, signaux de régulation, conflits entre séquences, etc.) Ce champ est lui-même subdivisé par ce que l on appelle des qualifiers ou descripteurs. Ces descripteurs sont nombreux et je n en citerai donc que quelques uns : /gene contenant le nom du gène, /protein_id donnant le numéro d accession de la protéine encodée par le gène, /db_xref pointant vers une référence croisée dans une autre banque, et /translation contenant la traduction en acides aminés de la partie codante. La structure des features a été standardisée il y a quelques années et est désormais identique pour les trois banques. Enfin, la séquence elle-même est signalée par le champ SQ (ORIGIN), tandis que la fin de l entrée est indiquée par un // DISTRIBUTION ET MISE À JOUR De nouvelles versions de ces banques sont proposées avec une périodicité de deux mois pour GenBank et trois mois pour EMBL. Par ailleurs, les trois centres de saisie - 6 -
11 BANQUES DE DONNÉES procèdent à des mises à jour quotidiennes de leurs banques respectives. La récupération des données se fait désormais uniquement par l intermédiaire de transferts FTP (File Transfer Protocol), ce qui pose de sérieux problèmes techniques du fait de la taille conséquente atteinte par les fichiers et de l encombrement du réseau Internet BANQUES DE SÉQUENCES PROTÉIQUES Tout comme leurs homologues consacrées aux séquences nucléotidiques, les banques dédiées aux protéines ont une organisation centrée autour de deux types d informations : les annotations biologiques et biochimiques d une part (avec les références bibliographiques et les données taxonomiques associées), et les séquences d autre part TREMBL ET GENPEPT Il existe une version «protéomique» de deux des grandes banques généralistes de séquences nucléotidiques EMBL et GenBank. Ainsi TrEMBL, distribuée par l EBI, contient la traduction de toutes les parties codantes annotées figurant dans EMBL, avec exclusion des protéines figurant dans SWISS-PROT. Certaines protéines présentant une variabilité très importante (comme les immunoglobulines) sont également exclues de TrEMBL. De son côté GenPept, distribuée par le NCBI, correspond à la traduction de l ensemble des parties codantes de GenBank. Ces deux systèmes ne peuvent cependant être considérés comme de véritables banques protéiques, du fait que les annotations qu ils contiennent sont uniquement celles provenant des séquences nucléotidiques. Les véritables banques de protéines contiennent de nombreuses informations spécifiques, comme nous allons le voir dans les sections suivantes SWISS-PROT La principale banque de protéines à l heure actuelle est sans conteste SWISS-PROT (Bairoch et Apweiler, 2000). Cette banque a été créée en 1986 par Amos Bairoch à Genève, et elle est maintenue et distribuée conjointement par le SBI (Swiss Institute of Bioinformatics) et l EBI. Le format de données adopté par SWISS-PROT suit de très près celui en vigueur à l EMBL (Figure I.3). Quelques champs supplémentaires sont introduits, comme GN ou DR, contenant respectivement le nom du gène codant pour la protéine considérée et des références croisées avec d autres banques. Par ailleurs, le champ DE est rempli non pas en fonction d informations fournies par les auteurs des séquences, mais bien par les annotateurs eux-mêmes, ceci permettant de donner une cohérence beaucoup plus grande à son contenu. Les séquences présentes dans cette banque proviennent de quatre origines : la traduction des gènes annotés dans EMBL, certaines protéines issues d autres banques protéiques, la consultation de publications scientifiques et quelques soumissions directes par les auteurs. Un certain nombre de raisons font que SWISS-PROT est devenue la banque de référence pour les séquences protéiques. La première d entre elles est une redondance minimale, les différentes versions d une même entrée étant fusionnées, avec annotation des positions conflictuelles. Par ailleurs, lorsque la séquence d une protéine est identique dans plusieurs espèces, une seule entrée est créée. Un autre atout de SWISS- PROT est également l introduction d un nombre très important de références croisées avec d autres banques de données (plus d une cinquantaine). Cette particularité se révélant particulièrement utile avec le développement d Internet et les possibilités d interconnections des banques qu offre ce réseau
12 CHAPITRE I ID ARP_ECOLI STANDARD; PRT; 728 AA. AC P23325; P76781; DT 01-NOV-1991 (Rel. 20, Created) DT 01-OCT-1993 (Rel. 27, Last sequence update) DT 01-NOV-1997 (Rel. 35, Last annotation update) DE ANKYRIN-LIKE REGULATORY PROTEIN. GN ARP. OS Escherichia coli. OC Bacteria; Proteobacteria; gamma subdivision; Enterobacteriaceae; OC Escherichia. [ ] RN [2] RP SEQUENCE OF FROM N.A. RX MEDLINE; RA Galinier A., Bleicher F., Negre D., Perriere G., Duclos B., Cozzone A.J., RA Cortay J.-C.; RT "Primary structure of the intergenic region between acek and iclr in RT the Escherichia coli chromosome."; RL Gene 97: (1991). [ ] DR EMBL; U00006; AAC ; -. DR EMBL; AE000474; AAC ; -. DR EMBL; M63497; AAA ; -. DR PIR; JQ0870; JQ0870. DR ECOGENE; EG11208; arp. DR PRODOM; P23325; P DR SWISS-2DPAGE; P23325; P KW Repeat; ANK repeat. FT DOMAIN X ANK MOTIF REPEATS. FT REPEAT ANK MOTIF 1. FT REPEAT ANK MOTIF 2. FT REPEAT ANK MOTIF 3. FT REPEAT ANK MOTIF 4. FT REPEAT ANK MOTIF 5. FT REPEAT ANK MOTIF 6. FT REPEAT ANK MOTIF 7. FT CONFLICT N -> D (IN REF. 2). FT CONFLICT GFTDNPRYIAEKNYMEALLKKASPHTVR -> FT TQKSISPYRTLNLCLRRYA (IN REF. 2). SQ SEQUENCE 728 AA; MW; E CRC64; MITRIPRSSF SANINNTAQT NEHQTLSELF YKELEDKFSG KELATPLLKS FSENCRQNGR HIFSNKDFVI KFSTSVLQAD KKEITIINKN ENTTLTQTIA PIFEKYLMEI LPQRSDTLDK QELNLKSDRK EKEFPRIKLN GQCYFPGRPQ NRIVCRHIAA QYINDIYQNV DYKPHQDDYS [ ] DFWQHKAPQK YSAFELAFEF GHRVIAELIL NTLNKMAESF GFTDNPRYIA EKNYMEALLK KASPHTVR // Figure I.3. Exemple d entrée SWISS-PROT. Une autre des caractéristiques de SWISS-PROT est la qualité particulièrement élevée de ses annotations. Les différentes catégories d informations figurant dans la banque comprennent notamment la ou les fonctions des protéines, les modifications post-traductionnelles, les sites et domaines structuraux ou fonctionnels identifiés, les structures secondaires et quaternaires, les similarités avec d autres protéines, les positions conflictuelles pour chaque entrée, etc. Toutes ces annotations proviennent à la fois d une consultation régulière de la bibliographie et de l apport d informations par des «experts» sur certaines familles de protéines. Un certain nombre d organismes modèles, objets de projets de séquençage, ont par ailleurs été sélectionnés par SWISS- PROT de façon à améliorer encore le niveau d annotations de leurs protéines, et à établir des références croisées avec des banques spécialisées. Enfin, il faut savoir que SWISS-PROT se situe au cœur d un ensemble de banques de données développées par le SIB. Ces banques lui sont complémentaires et couvrent de nombreux aspects de la biologie et de la biochimie des protéines. On peut ainsi citer PROSITE (Hofman et al., 1999), dédiée aux motifs consensus caractéristiques d une famille de protéines ; ENZYME (Bairoch, 2000), sur la nomenclature des enzymes ; SWISS-2DPAGE (Hoogland et al., 2000) sur les données de migration électrophorétiques ; et SWISS-3DIMAGE (Petsch et al., 1995) sur les images de structures tridimensionnelles. Cette intégration est réalisée de façon exemplaire au niveau d ExPASy 3 le serveur Web du SIB
13 BANQUES DE DONNÉES SWISS-PROT constitue donc une exception dans le monde des banques de séquences généralistes dans le sens qu elle s attache à répondre aux critiques habituellement formulées à l égard de ces systèmes. Avec le flot continu de séquences qu apportent les projets génomes, la richesse des annotations dans SWISS-PROT est, de façon inévitable, obtenue au détriment de l exhaustivité des séquences. Cependant, si l on considère cette banque en prenant en compte son annexe TrEMBL, ce problème disparaît PIR Les origines de la banque PIR (Protein Information Resource) sont anciennes puisque la toute première version remonte au milieu des années 60 (Dayhoff et al., 1965). Depuis 1988, cette banque de données est maintenue conjointement par la NBRF (National Biomedical Research Foundation) aux États-Unis, le MIPS (Martinsried Institute for Protein Sequences) en Allemagne, et la JIPID (Japan International Protein Information Database) au Japon. Le but de cette collection est de fournir des informations exhaustives et non redondantes organisées selon des critères taxonomiques et de similarité (Barker et al., 2000). Si l exhaustivité semble effectivement atteinte, il reste encore un taux de redondance non négligeable. Par ailleurs, la qualité des annotations est bien moindre que celle de SWISS-PROT. La banque PIR comprend trois composantes : les références bibliographiques, les séquences protéiques telles qu elles ont été publiées, et des séquences canoniques construites à partir des différentes versions d une même entrée. Les données proviennent de trois sources : les publications scientifiques, les soumissions des auteurs, et la traduction des parties codantes annotées présentes dans les banques nucléotidiques. Quelques références croisées ont été mises en place avec les banques de séquences nucléotidiques et quelques banques spécialisées, mais en nombre nettement moins important que dans SWISS-PROT. Alors que dans SWISS-PROT la classification des protéines en familles est réalisée essentiellement en utilisant les motifs PROSITE, PIR utilise une approche bien différente pour construire ses superfamilles. Cette approche étant basée sur des similarités de séquence mais aussi de fonction (Barker et al., 1996) AUTRES BANQUES D autres banques ayant connu un succès bien moindre que SWISS-PROT et PIR ont été également développées. On peut ainsi citer OWL (Bleasby et al., 1994) une banque composite et non redondante construite à partir de quatre sources de données : la traduction de parties codantes de GenBank, SWISS-PROT, PIR et NRL_3D (Pattabiraman et al., 1990). SWISS-PROT était la source d information prioritaire, représentant près de la moitié des entrées de OWL, les trois autres collections permettant d introduire des séquences manquantes. Des critères très stricts de non redondance faisaient d OWL une banque relativement petite en volume, mais théoriquement exhaustive en quantité de données. Cependant, cette banque n a plus connu de nouvelle version depuis août 1998 et sa maintenance semble désormais abandonnée. Plus récemment, on a vu l apparition de KIND (Kallberg et Persson, 1999), une banque du même type que OWL mais construite à partir de TrEMBL, GenPept, SWISS-PROT et PIR. Là encore, la fréquence d apparition de nouvelles versions semble assez limitée
14 CHAPITRE I 1.3. INTERÊT ET LIMITES DES BANQUES GÉNÉRALISTES Les banques de données généralistes en biologie moléculaire constituent un outil absolument indispensable à l activité quotidienne du chercheur amené à travailler avec des séquences. Elles représentent une source précieuse d informations, dont l exhaustivité permet théoriquement d effectuer des recherches efficaces sur les connaissances du moment au sujet de tel gène ou de telle protéine. De plus, elles centralisent en un même lieu et sous un même format l ensemble des séquences connues. Ainsi les critiques que je vais maintenant formuler ne doivent pas faire oublier l absolue nécessité de l existence de ces banques. Les défauts de ces types de collections sont en effet multiples, parmi lesquels de trop nombreuses erreurs, notamment au niveau des séquences mêmes (erreurs de séquençage, de saisie, présence dans les séquences de vecteurs de clonage, etc.) Erreurs qui déprécient sensiblement les bénéfices apportés par ces outils. Cette qualité très inégale, voire même médiocre, des informations stockées peut être expliquée par plusieurs raisons : La libre soumission des séquences et de leurs caractéristiques par les auteurs entraîne imprécisions, omissions, voire erreurs. La responsabilité des biologistes ayant déterminé et soumis les séquences n est pas systématiquement mise en cause ici, mais il faut reconnaître que les régions attenantes au(x) gène(s) considéré(s) dans le cadre d une étude donnée sont bien souvent approximativement séquencées et pauvrement annotées. Le format de stockage des données est très strict en théorie, mais il l est malheureusement moins dans son application. Ce problème se révèle particulièrement aigu au niveau des features où les différents types d informations qu il est possible d annoter sont souvent utilisés à mauvais escient, ceci sans même évoquer les cas trop fréquents où des informations importantes sont placées sans aucune structuration au niveau des commentaires. Il existe une forte incohérence des informations, particulièrement au niveau des mots-clés. Ceux-ci devraient théoriquement permettre une extraction aisée et fiable d informations, mais l existence de multiples formulations pour désigner un même concept rend cet outil à peu près inutilisable. Malgré tout, des outils plus performants mis à la disposition des biologistes pour soumettre les séquences, ainsi que des contrôles de cohérence accrus lors de l intégration de nouvelles informations dans les collections devraient permettre une amélioration sensible de la qualité des données. Cependant, ces progrès n auront probablement aucun effet sur le deuxième problème majeur des banques généralistes : la redondance des informations. En effet, pour une des raisons évoquées précédemment la libre soumission de nombreuses séquences se trouvent partiellement ou intégralement dupliquées dans ces collections. Ces doublons présentent très fréquemment des variations dans leurs séquences et/ou leurs annotations. D une certaine façon, ce constat constitue un point positif en autorisant des comparaisons entre différents points de vue sur une même information. En effet, il peut exister des variations naturelles entre des souches ou des individus différents. Cette redondance pose tout de même deux problèmes : d une part, les différences observées au niveau des séquences et des annotations représentent des difficultés extrêmement complexes à résoudre lorsqu il s agit d étudier un fragment de séquence particulier car il est impossible de décider par un processus automatique si elles sont issues d un polymorphisme allélique naturel, d erreurs de séquençage, ou encore de
15 BANQUES DE DONNÉES duplications de gènes (Perrière et al., 1994). D autre part, il est clair que toute analyse globale sera fortement biaisée par cette duplication d informations. Il est donc nécessaire, avant d entreprendre toute expérience sensible à ce phénomène de redondance, d éliminer proprement ces duplications, ce qui n est pas chose aisée. Ce problème de la redondance a encore été aggravé avec l arrivée des génomes complets. En effet, les séquences de ces génomes ne figurent pas, dans les versions standard des collections généralistes, sous la forme de contigs d un seul tenant, mais sous la forme de fragments chevauchants d une longueur de 300 kb maximum. Le plus fréquemment, les génomes (ou les chromosomes) sont découpés en morceaux de 100 kb avec un chevauchement de 10 kb. Cette décision de découper les très grandes séquences en fragments est liée à la volonté de conserver la compatibilité des banques avec le système GCG (Genome Computer Group), le package le plus largement employé en analyse des séquences aux États-Unis (Devereux et al., 1984). En effet, du fait de ses origines remontant aux années 80, les programmes faisant partie de ce système sont incapables de gérer des séquences dont la longueur est supérieure à 300 kb. 2. BANQUES SPÉCIALISÉES L existence de ces nombreux problèmes inhérents aux banques généralistes a conduit tout naturellement à considérer les solutions permettant de pallier ces inconvénients. Parmi celles-ci, les collections spécialisées représentent probablement le meilleur moyen pour répondre aux critiques précédemment formulées. Il faut cependant garder à l esprit qu une des raisons de la création de telles banques tient également à la nécessité d introduire des informations spécifiques, informations qui n auraient rien à faire dans les collections généralistes BANQUES THÉMATIQUES Les banques de ce type se consacrent à une thématique biologique bien précise. Elles intègrent donc des séquences et des données qui sont spécifiques à ce domaine. Ne seront abordées dans cette section que des banques comprenant des séquences nucléotidiques ou protéiques dans leur structure. Il existe en effet de multiples collections abordant des aspects de la biologie moléculaire non directement liés aux séquences (e.g., sur le métabolisme ou les réseaux de régulation), et la description de tels systèmes ne rentre pas dans le cadre de cet ouvrage STRUCTURES MOLÉCULAIRES La principale banque de structures tridimensionnelles est la PDB (Protein Data Bank) (Berman et al., 2000). Cette banque, créée aux États-Unis en 1977, est une collection de structures de macromolécules biologiques. Les protéines constituent l essentiel des entrées de la PDB, mais on y trouve aussi des structures de molécules de RNA et de DNA, de complexes protéine-acides nucléiques, de virus et de polyosides. Ces structures ayant quasiment toutes été déterminées expérimentalement par cristallographie aux rayons X ou par RMN, seul un petit nombre d entrées de la PDB (2 %) provenant de résultats de modélisation moléculaire. Les entrées de la banque comprennent des informations sur les structures primaires et secondaires des molécules considérées, les coordonnées des atomes ainsi que des références bibliographiques
16 CHAPITRE I Bien que le nombre de structures de macromolécules biologiques connues à l heure actuelle soit très inférieur à celui des séquences ( structures dans la PDB contre respectivement et protéines dans SWISS-PROT et PIR en mars 2000), celui-ci croît actuellement à une vitesse comparable à celle observée pour les séquences protéiques il y a quelques années (Figure I.4). log(entrées) 4,25 4 3,75 3,5 3,25 3 2,75 2,5 2,25 2 1,75 09/78 04/81 12/83 07/86 02/89 09/91 04/94 12/96 07/99 Date Figure I.4. Croissance du nombre d entrées de la PDB depuis sa création. MMDB (Molecular Modelling Data Base) est une banque construite par le NCBI à partir des entrées de la PDB avec une intégration au sein d Entrez (Wang et al., 2000). L utilisation de ce puissant système d interrogation permet le lien avec les références des articles dans lesquels les structures ont été publiées. Entrez permet également de récupérer toutes les protéines de GenPept présentant une similarité de séquence avec une entrée quelconque de MMDB. Mais de plus, grâce au couplage avec le programme VAST (Gibrat et al., 1996), il est possible de récupérer toutes les entrées présentant une similarité de structure. En effet, il existe des protéines qui peuvent avoir un repliement semblable sans pour autant avoir une similarité de séquence très marquée. SCOP (Structural Classification of Proteins) regroupe les différents types de structures de protéines en familles (Lo Conte et al., 2000). Cette classification s organise sur plusieurs niveaux hiérarchiques : superfamilles, familles, et repliements communs. Les deux premiers niveaux sont bâtis sur les relations évolutives (inférées par la similarité) existant entre les protéines, tandis que le troisième utilise des relations au niveau de la structure spatiale proprement dite. Dans ce cas, les protéines sont regroupées si elles possèdent les mêmes structures secondaires principales avec un arrangement et des connections topologiques identiques. Cinq grandes classes de repliements sont définies dans SCOP en fonction de leurs proportions d hélices α et de feuillets β ainsi que de l agencement de ces structures. HSSP (Homology-derived Secondary Structure of Proteins) contient une classification en familles des protéines de SWISS-PROT établie par des recherches de similarités avec les protéines figurant dans la PDB (Holm et Sander, 1999). Chaque famille est donc centrée sur une protéine de structure 3D connue. De cette façon, il est possible d inférer les structures secondaires et tertaires potentielles des membres de cette famille. À l heure actuelle, près de 36 % des protéines de SWISS-PROT sont classées dans HSSP
17 BANQUES DE DONNÉES Enfin, BMRB (BioMagResBank) est une base de données relationnelle contenant des déplacements chimiques de RMN dérivés de peptides et de quelques protéines ainsi que des informations de séquence, de bibliographie, et sur les conditions expérimentales d obtention de la structure (Seavey et al., 1991) SÉQUENCES ET STRUCTURES DE RNA Il existe deux compilations de séquences de rrna, une en Europe et une aux États- Unis. Le contenu de ces deux banques est assez proche puisque toutes deux intègrent non seulement les séquences des rrna de la grande ou de la petite sous-unité du ribosome, mais aussi des alignements et des arbres phylogénétiques. La banque américaine est la RDP (Ribosomal Database Project) (Maidak et al., 2000), tandis que son homologue européenne se divise en deux parties : LSU rrna (Large Subunit rrna database) (De Rijk et al., 2000), consacrée à la grande sous-unité, et SSU rrna (Small Subunit rrna database) (Van de Peer et al., 2000), consacrée à la petite sousunité. Dans ces deux banques, les alignements disponibles présentent la caractéristique très intéressante d être corrigés manuellement, ceci afin de permettre l obtention d arbres phylogénétiques plus fiables. En effet, la qualité d une phylogénie est directement liée à la qualité de l alignement utilisé, et il est bien connu que des alignements sur des séquences aussi longues que des rrna doivent être retravaillés avant d utiliser une méthode de reconstruction phylogénétique. Pour ce qui est des trna, il existe une collection regroupant les séquences des gènes et des molécules transcrites (puisque les trna matures contiennent de nombreuses bases modifiées) (Sprinzl et al., 1998). Outre les séquences, cette banque contient également des structures secondaires ainsi que des alignements basés en partie sur ces structures FAMILLES DE GÈNES Avec le développement de la génomique comparative, de plus en plus de banques de séquences proposent une classification des gènes protéiques sous la forme de familles. Le principe de base pour la construction de ces banques est toujours le même. Dans un premier temps une recherche de similarité entre toutes les protéines d un ensemble donné est effectuée au moyen d un logiciel comme BLAST (Basic Local Alignment Search Tool) (Altschul et al., 1990, 1997) ou FASTA (Pearson et Lipman, 1988), ces protéines étant ensuite regroupées en familles en utilisant des critères de similarité. Ce regroupement peut s effectuer en utilisant des associations qui vont du «simple lien» au «lien complet», avec de nombreuses nuances entre ces deux extrêmes. En simple lien, si une protéine A passe le critère de similarité avec une protéine B, et si la protéine B passe le critère de similarité avec une protéine C, alors A, B et C sont classées ensemble même si la paire A/C ne répond pas à ce critère. En lien complet, toutes les paires possibles à l intérieur d une famille doivent passer le critère de similarité. Parmi les différentes banques de ce type, une des premières est ProtFam 4, distribuée par le MIPS (Mewes et al., 2000). ProtFam est construite à partir des séquences de PIR et elle intègre quatre niveaux de similarités entre les protéines. Au plus haut niveau les protéines sont groupées en utilisant la classification en superfamilles de PIR. Ces superfamilles sont ensuite subdivisées en familles, sous-familles et entrées en utilisant des niveaux de similarité de plus en plus forts (égaux respectivement à 50 %, 80 % et 95 %). Cette banque intègre également des alignements et des dendrogrammes
18 CHAPITRE I construits à partir des séquences complètes mais aussi à partir des domaines de la banque Pfam (Bateman et al., 2000). Qui plus est, l utilisateur a la possibilité de soumettre ses propres séquences sur le serveur de ProtFam, ceci afin de déterminer dans quelle famille celles-ci se placent. 7096(1) (1) 1 3(1) (2) (37) (3) (2) (73) Figure I.5. Relations d homologie existant entre plusieurs familles de ProtoMap. Chaque cercle représente une famille et son diamètre est proportionnel au nombre (indiqué entre parenthèses) de gènes qui en font partie. La taille des traits reliant deux familles est proportionnelle au nombre de liens existant entre elles. Ainsi la famille 44 contient deux gènes qui sont liés à un gène de la famille 2399 comme l indiquent les deux nombres figurant sur le trait. ProtoMap 5 est l équivalent de ProtFam pour la banque SWISS-PROT (Yona et al., 2000). Au lieu d utiliser le score d un seul type de programme pour construire les familles, les concepteurs de cette banque ont préféré employer la combinaison de trois mesures : Smith et Waterman (1981), BLAST et FASTA. Il est possible de visualiser les alignements ou d explorer la classification des protéines au sein d une famille en utilisant une représentation sous forme d arbres, le serveur dédié à la banque intègrant un ensemble d applets Java dotées d interfaces graphiques. Là encore, il est possible de soumettre des séquences personnelles afin de déterminer à quelle famille elles se rattachent. ProtoMap n intègre pas de données sur les domaines protéiques, par contre cette banque permet de visualiser les relations d homologies qui peuvent exister avec des protéines appartenant à des familles différentes (Figure I.5). La banque SYSTERS 6, quant à elle, est construite à partir de la fusion nonredondante de protéines de PIR et de SWISS-PROT (Krause et al., 2000). Cette banque intègre une classification en superfamilles, familles et sous-familles. La classification en familles se fait en affinant les alignements locaux produits par BLAST au moyen du programme LALIGN (Huang et Miller, 1991), plus sensible. Trois types de familles sont définies en fonction de la nature du lien qui relie les protéines entre
19 BANQUES DE DONNÉES elles (du simple lien au lien complet). Les familles les plus voisines entre elles sont ensuite regroupées en superfamilles. Tout comme les deux banques précédentes, SYSTERS comprend également des alignements multiples et des arbres, les alignements intégrant la localisation des domaines protéiques de Pfam. ProClass 7 utilise la classification en superfamilles de PIR en lui ajoutant les définitions fournies par les motifs PROSITE (Huang et al., 2000). Ce système intègre un moteur de recherche et de classification, de nom GeneFIND (Wu et al., 1998), permettant, là encore, de rattacher une protéine fournie par l utilisateur à une famille donnée. Par contre ProClass ne contient pas d alignements ni d arbres et se contente de proposer des liens vers des banques incluant ce genre de données. Enfin, il existe des banques qui ne se consacrent qu à un groupe d organismes, comme HOVERGEN (Homologous Vertebrate Genes database) (Duret et al., 1999), HOBACGEN (Homologous Bacterial Genes database) (Perrière et al., 2000) ou COG (Tatusov et al., 2000). HOVERGEN contient les séquences de tous les gènes de vertébrés figurant dans GenBank, avec une classification réalisée à partir d une recherche de similarité effectuée sur la traduction de ces gènes. De la même façon, HOBACGEN se consacre aux gènes de procaryotes en partant des séquences de SWISS-PROT/TrEMBL. Je reviendrai en détail sur la structure et le contenu de cette banque dans le deuxième chapitre de ce mémoire. Quant à COG, cette banque ne s intéresse qu aux protéines figurant dans les organismes pour lesquels le génome complet est disponible DOMAINES PROTÉIQUES Parallèlement aux familles de gènes, de nombreuses banques s occupent de collecter des données sur la structure en domaines des séquences protéiques. Leur nombre est si important que certains envisagent même de regrouper toutes ces collections en une seule, tant leur contenu est semblable! La raison du succès de ces systèmes est le fait qu il est maintenant bien établi qu une majorité de protéines, y compris chez les bactéries, possède une structure modulaire (Patthy, 1991, 1994). Le nombre moyen de domaines présents dans une protéine ayant été estimé à trois par Corpet et al. (2000). Parmi toutes ces banques, la plus communément utilisée est sans doute ProDom (Corpet et al., 2000). Cette banque contient un ensemble de domaines généré automatiquement à partir des séquences de SWISS-PROT/TrEMBL. La procédure de construction des domaines est basée sur une utilisation récursive du program PSI- BLAST (Gouzy et al., 1999). Certains domaines sont cependant validés au moyen d une expertise manuelle. De plus, les familles de Pfam-A sont également utilisées pour augmenter le nombre des domaines examinés par des experts. Le serveur Web consacré à ProDom permet d accéder à une représentation graphique de l arrangement des domaines protéiques 8. Chaque protéine est représentée par une succession de boîtes utilisant un code conjuguant motifs et couleurs différents permettant d identifier les domaines (Figure I.6). Pfam 9 est construite à partir d une version de SWISS-PROT/TrEMBL dans laquelle toutes les redondances ont été éliminées. Cette banque comprend en fait deux parties : Pfam-A et Pfam-B. La différence entre ces deux divisions tient au fait que les domaines de Pfam-A sont expertisés par des curateurs tandis que ceux faisant partie de Pfam-B sont générés par un programme partant des domaines ProDom. L algorithme
20 CHAPITRE I utilisé pour construire Pfam-B est basé sur l utilisation de chaînes de Markov cachées et ce programme utilise toutes les familles de ProDom non construites à partir de Pfam-A. L interface Web de Pfam comprend une applet Java permettant de visualiser non seulement les alignements mais aussi les dendrogrammes construits sur les domaines. Figure I.6. Visualisation au moyen de l interface Web de la banque d un ensemble de protéines possédant en commun au moins un domaine ProDom. SBASE 10 (Murvai et al., 2000) est construite en utilisant les séquences de SWISS- PROT et de PIR ainsi que les traductions de parties codantes de GenBank et EMBL. SBASE utilise un concept un peu différent de ceux employés dans les autres banques du même type. Plutôt que de se baser sur des profils, des chaînes de Markov cachées ou des séquences consensus, ce système utilise ce que ses concepteurs appellent des «groupes de similarité». Ces groupes sont constitués par des ensembles de domaines présentant des scores BLAST plus ou moins élevés. Il existe ainsi des ensembles à forte ou faible similarité lorsque l on considère la moyenne des scores BLAST entre les différents représentants d un groupe donné. Là encore, la banque est divisée en deux parties : SBASE-A qui contient les domaines bien établis du point de vue structural et fonctionnel, et SBASE-B, qui contient les domaines moins bien caractérisés ou définis uniquement par leur composition (e.g., les régions riches en glycine). De nombreuses autres banques de domaines ont été construites. On peut ainsi citer Domo (Gracy et Argos, 1998), qui utilise une procédure de création automatique proche de celle employée par ProDom. La banque Blocks (Henikoff et al., 2000), quant à elle, est construite à partir des familles protéiques définies à partir de signatures PROSITE. Ces données sont ensuite complémentées avec celles de PRINTS (Attwood et al., 2000), ProDom, Pfam-A, et Domo. Enfin, COLIPAGE (Labedan et Riley, 1999) ne se consacre qu aux modules paralogues d une seule bactérie : E. coli, bien que ses
21 BANQUES DE DONNÉES concepteurs aient l intention de l étendre à toutes les bactéries dont le génome complet est disponible SIGNATURES PEPTIDIQUES Très proches des banques de domaines, figurent les collections qui compilent des «signatures», caractéristiques de certaines protéines. L utilisation de ces signatures oligopeptidiques est particulièrement utile pour essayer de classer des protéines qui ne présentent pas de similarités très significatives avec d autres séquences. La différence essentielle avec les banques de domaines vient que les motifs qui y figurent sont souvent de plus petite taille. PROSITE 11 a été la première collection de ce type à être développée, ceci dès Les entrées figurant dans cette banque peuvent être divisées en deux catégories : les motifs proprement dits et les profils. Les motifs sont des régions bien conservées, cette conservation étant généralement liée à des contraintes fonctionnelles très fortes (e.g., au niveau des sites actifs des enzymes). Les profils sont utilisés dans le cas de sites où le degré de conservation est faible et limité à quelques positions. A C D E F G H I K L M N P Q R S T V W Y T T G K R G W F P G N Y V E P Table I.1. Exemple de profil tiré de la banque PROSITE. En ligne figurent les scores de chacun des 20 acides aminés pour chacune des 15 positions du motif, la dernière ligne donnant le consensus parfait. Un profil consiste en une matrice pondérée avec les acides aminés en ligne et les positions en colonne (Table I.1). En utilisant une matrice de ce type il est possible de calculer le score d une séquence peptidique quelconque, et donc de déterminer si elle peut être considérée ou non comme une occurrence de la signature. La procédure de création des profils PROSITE utilise en entrée des alignements multiples ainsi qu une table de comparaison de symboles afin de convertir les fréquences des acides aminés en pondérations (Gribskov et al., 1990 ; Luethy et al., 1994)
22 CHAPITRE I PRINTS 12 est la seconde principale collection de motifs disponible. Les premières versions de PRINTS étaient construites à partir de la banque OWL. Du fait de l arrêt de cette dernière, cette banque est maintenant développée à partir de SWISS- PROT/TrEMBL. L idée de base de ce système est de partir des régions les plus conservées obtenues dans des alignements de séquences ; les zones avoisinantes pouvant éventuellement être utilisées pour affiner la classification. Du fait que le processus d extraction de ces régions conservées est particulièrement complexe et implique de nombreuses opérations manuelles, les concepteurs de PRINTS éprouvent d importantes difficultés à sortir de nouvelles versions de leur banque. Celle-ci n est donc plus du tout synchrone avec les versions récentes de SWISS-PROT BANQUES GÉNOMIQUES Les problèmes des banques généralistes ainsi que leur structure même font qu il est très difficile (voire même impossible) d extraire efficacement et avec précision l ensemble des informations concernant un organisme donné. Pour cette raison, un grand nombre de collections consacrées exclusivement à un (voire à plusieurs) génome(s) particulier(s) ont vu le jour ces dernières années. Les systèmes les plus nombreux sont ceux consacrés aux organismes unicellulaires comme les bactéries, les archées ou la levure mais il existe également quelques banques dédiées à certains eucaryotes PROCARYOTES Avant que les premiers génomes complets ne soient disponibles, beaucoup de banques se consacraient à un organisme unicellulaire particulier (procaryote ou eucaryote). La caractéristique commune à ces différents systèmes était que leurs concepteurs partaient des collections généralistes pour en éliminer les redondances. Par ailleurs, les séquences chevauchantes étaient fusionnées sous la forme de contigs ce qui impliquait d effectuer des choix quand les zones chevauchantes différaient dans leur séquence. Dans certains cas, les erreurs présentes dans les annotations (voire les séquences ellesmêmes) étaient corrigées. La construction de tels systèmes nécessitait donc une somme de travail très importante, ceci d autant plus que l organisme en question possèdait un génome de grande taille. Avec l arrivée de plus en plus fréquente de génomes complets publiés d un seul coup, la construction de banques de ce type n a plus guère de raisons d être. Certains systèmes subsistent néanmoins, du fait d apports notables au niveau de la qualité des annotations et par des corrections de la séquence. Alors que le génome d E. coli était en cours de séquençage, on ne comptait pas moins de trois compilations de séquences consacrées à cet organisme : Colibri (Médigue et al., 1993), ECDC (E. coli Database Collection) (Wahl et Kroeger, 1995) et EcoGene (Rudd, 1993). De toutes ces banques, seule cette dernière est encore maintenue par son concepteur (Rudd, 2000), le système Colibri étant utilisé pour gérer et visualiser les données d EcoGene (Figure I.7). EcoGene consiste en une compilation des gènes et des séquences protéiques présentant de nombreuses corrections par rapport aux séquences figurant dans GenBank. Ainsi, les sites de démarrage de la traduction de 706 gènes ont été corrigés. De même 61 décalages de phase, probablement provoqués par des erreurs de séquençage ont été détectés. Au niveau des ajouts figurent des références croisées avec près de références bibliographiques liées aux gènes d E. coli
23 BANQUES DE DONNÉES rrle rrfe yjaa meta aceb acea acek (a) yjab arpa : 2187 bp (728 aa) Go to options ( Navigate, Export ) Synonym b4017, yjac Type CDS iclr Mnemonic Ankyrin-like regulatory protein Accession number EG11208 Top Cross-references meth Description Regulator of acetyl-coenzyme A synthetase gene expression BLASTP reports against Nrprot (run date: Feb 25, 2000) Location Coordinates SWISS-PROT P23325 Position kb From Codon usage 3 Molecular weight Isoelectric point Blattner b4017 Calculated map To " Direction GenBank g (b) yjbb Help pepe arpa yjbc yjbd Figure I.7. Visualisation d un fragment de la carte génétique du chromosome d E. coli (a) ainsi que des annotations associées au gène arpa (b) au moyen de l interface Web de Colibri. Dans le cas de Bacillus subtilis, deux banques ont été conçues vers le milieu des années 90 : NRSub (Non-Redundant B. subtilis) (Perrière et al., 1994) et SubtiList (Moszer et al., 1995). Par la suite, les séquences figurant dans NRSub ont été récupérées de SubtiList et la seule différence existant entre ces deux banques résidait dans l emploi de systèmes d interrogation à la philosophie très différente (Perrière et al., 1996b) ; NRSub permettant d effectuer des requêtes complexes au moyen du système ACNUC et SubtiList autorisant une navigation graphique dans la carte physique du chromosome par l intermédiaire du SGBD 4 ème Dimension. Avec la disponibilité d un nombre de plus en plus important de génomes, on constate le remplacement progressif des collections consacrées à un seul organisme par des serveurs Web permettant d accéder à l ensemble des génomes complets disponibles. Cependant, rares sont les banques qui sont distribuées pour pouvoir être installées localement. En effet, ces collections sont le plus souvent associées à un SGBD propriétaire installé sur le serveur. Une exception est la banque EMGLib (Enhanced Microbial Genomes Library) (Perrière et al., 2000) qui est décrite dans le deuxième chapitre de cet ouvrage SACCHAROMYCES CEREVISIAE Concernant le génome de la levure, plusieurs collections de données spécialisées ont été mises en place bien avant que le génome complet ne soit connu. Ainsi le MIPS, responsable de la gestion et de l analyse des séquences dans le cadre du projet de séquençage de la levure distribue la banque MYGD (MIPS Yeast Genome Database) (Mewes et al., 2000). Cette banque intègre les séquences des gènes, des données biochimiques ainsi que de nombreux résultats issus de la littérature et du projet européen d analyse fonctionnelle de cet organisme (Oliver, 1996). Par ailleurs, MYGD intègre des références croisées aux superfamilles de PIR ainsi qu aux familles de ProtFam et de COG
24 CHAPITRE I La banque SGD (Saccharomyces Genome Database) (Ball et al., 2000) constitue une autre source exhaustive d informations sur le génome de S. cerevisiae : séquences complètes des chromosomes, annotations des gènes et des protéines, cartes physiques et génétiques. De son côté, YPD (Yeast Proteome Database) (Costanzo et al., 2000) consacrée aux protéines, contient surtout des données biochimiques et fonctionnelles obtenues par une analyse systématique de la littérature. Cette banque comporte également de nombreux liens vers SGD DROSOPHILA MELANOGASTER La banque principale consacrée à la drosophile est FlyBase 13. À l origine, cette collection se consacrait essentiellement à la bibliographie sur les gènes et le génome de D. melanogaster. Depuis quelques années, FlyBase est en pleine réorganisation avec l intégration des données fournies par deux organismes : le BDGP (Berkeley Drosophila Genome Project) et l EDGP (European Drosophila Genome Project) (FlyBase Consortium, 1999). FlyBase contient des informations très variées sur les noms, localisations, fonctions et formes alléliques des gènes connus ainsi que sur des données moléculaires sur l expression des gènes. Cette banque permet également d accéder à la liste complète des clones de cosmides, YAC (Yeast Artificial Chromosome) et P1 disponibles, et à un nombre très important de références bibliographiques. Figure I.8. Captures d écran de l interface de navigation cartographique de FlyBase. Sur cet exemple figurent : en (a) la vue générale permettant de sélectionner un chromosome ainsi que les données à visualiser (gènes, clones, déficiences génétiques) ; en (b) une vue détaillée montrant les différents gènes localisés sur une région du chromosome X
25 BANQUES DE DONNÉES La structure utilisée est celle d une base de données relationnelle interfacée avec le Web. Un des modes d accès principaux à FlyBase est celui basé sur les localisations chromosomiques. Pour ce faire, une interface cartographique dévelopée en Java est utilisée (Figure I.8). Cette interface permet de représenter les données allant du niveau cytogénétique aux séquences. Il est à noter que cette interface fut, en 1995, l un des tout premiers développements bioinformatiques utilisant le langage Java. Du fait de la variabilité des vitesses de connection sous Internet, un certain nombre de miroirs de FlyBase ont été installés aux États-Unis, en France, en Israël, au Japon en en Australie HOMO SAPIENS La principale banque de données sur la cartographie du génome humain est la GDB 14 (Genome Data Base) (Letovsky, 1999). Cette banque contient des données cartographiques de plusieurs niveaux (cytogénétique, génétique, physique), avec les objets qui se trouvent sur les cartes : gènes, clones, marqueurs (STS, polymorphisme), etc. En elle-même la GDB ne contient pas de données de séquences mais elle incorpore des références croisées vers les collections généralistes. La structure de la banque repose sur un SGBD relationnel avec une couche supérieure orientée-objet utilisant le langage OPM (Object Protocol Model). Tout comme pour FlyBase, c est une interface écrite en Java qui permet de visualiser et de naviguer dans la structure des cartes. Cette interface permet de synchroniser le défilement de cartes obtenues à différentes échelles. Ce système permet aux curateurs de la GDB de modifier directement certaines données de la banque. Mise à part la GDB, il existe de nombreuses autres banques consacrées à divers aspects de la génétique humaine. On peut citer OMIM (Online Mendelian Inheritance in Man) (Scott et al., 1999), dédiée à la bibliographie et aux observations sur les différentes anomalies génétiques affectant l Homme, ainsi que les différentes banques produites en France par le CEPH (Centre d Étude du Polymorphisme Humain) et le Généthon AUTRES Il existe aujourd hui un très grand nombre d autres collections génomiques non décrites ici, liées aux projets correspondants de cartographie et de séquençage systématique. On peut ainsi citer les ressources informatiques autour du génome de la souris comme MGD (Mouse Genome Database) (Blake et al., 2000) ou EMG (Encyclopedia of the Mouse Genome) ; les différentes banques dédiées à la cartographie de plantes comme AtDB (Arabidopsis thaliana Data Base) (Rhee et al., 2000), INE (INtegrated rice genome Explorer) (Sakata et al., 2000) ou ZmDB (Zea mays Data Base) (Gai et al., 2000). Une liste exhaustive serait difficile à dresser et ne présenterait que peu d interêt, en raison d une évolution trop rapide dans ce domaine. Il est préférable pour cela de s adresser aux serveurs Web spécialisés qui ont pris l habitude de recenser l ensemble des collections disponibles. 3. SYSTÈMES D INTERROGATION Depuis qu il existe des banques de séquences, de nombreux systèmes permettant d accéder aux données et de récupérer des entrées correspondant à certains critères ont été développés. Seuls quatre d entre eux, largement utilisés, sont présentés dans cette
26 CHAPITRE I section ; ces quatre systèmes présentant entre autre la caractéristique de pouvoir être consultés en ligne au moyen de serveurs Web ou de logiciels 3.1. LA PRÉDOMINANCE D INTERNET L extraordinaire développement d Internet depuis 1994 a entraîné des modifications radicales dans les possibilités offertes aux utilisateurs des outils bioinformatiques, en particulier pour la consultation des banques de séquences. En fait, si ce domaine a connu des développements si importants au cours de ces cinq dernières années, c est aussi en partie grâce au développement d Internet. Cette explosion est elle-même liée à l apparition du World-Wide Web (plus souvent abrégé en WWW ou Web). Le Web fait appel au protocole de transfert par hypertexte HTTP (HyperText Transfer Protocol) créé au CERN en Les pages d informations accessibles par le Web sont décrites à l aide du langage HTML (HyperText Markup Language), qui permet de structurer les documents et de définir des pointeurs vers d autres serveurs. À travers des liens hypertexte, le Web donne accès à des documents de tout ordre (textes, images, sons) disponibles dans un grand nombre de formats. Depuis la création d HTTP, le nombre de serveurs Web n a pas cessé de croître (Figure I.9) log(sites) /93 06/94 03/95 12/95 09/96 06/97 03/98 12/98 09/99 Date Figure I.9. Croissance du nombre de serveurs Web depuis la création du protocole HTTP par le CERN en HTML permet également de définir des interfaces simples pour des programmes qui sont lancés sur le serveur. Ces programmes prennent le nom de scripts CGI (Common Gateway Interface), et ils peuvent être écrits en n importe quel langage pourvu que celui-ci accepte les entrées et les sorties sur le flot de données standard. Du fait de cette possibilité d interfaçage, la quasi-totalité des programmes d analyse des séquences ou des banques de données ont pu être mis sur le Web, ceci en l espace de trois ans seulement. Grâce au Web, les banques restent gérées localement sur un seul site et peuvent être consultées à distance par toute personne connectée au réseau ; ceci prévenant les difficultés posées par la duplication des informations en des lieux géographiquement distincts. La contrepartie de cet avantage est que, du fait de la saturation du réseau
27 BANQUES DE DONNÉES Internet, les vitesses de transfert ont tendance à décroître dramatiquement. Ce problème n est pas très important lorsqu un utilisateur désire travailler sur un petit nombre de séquences. Par contre, s il désire effectuer une étude statistique portant sur plusieurs milliers de séquences, les temps de transfert peuvent se compter en heures voire même en jours. Un deuxième problème, posé par l utilisation du Web est la pauvreté graphique du langage HTML. Les seuls éléments disponibles sont en effet des boutons, des sélecteurs, des éditeurs lignes et des champs de texte. Ceci est largement suffisant pour créer des interfaces permettant de définir les paramètres du programme à lancer, par contre la visualisation des résultats est parfois problématique, en particulier lorsque ceux-ci requièrent des interactions avec l utilisateur. En effet, ce langage ne propose aucun outil permettant de travailler sur des sorties graphiques. À la rigueur, il est possible de créer des images bitmap visualisables dans un navigateur quelconque, mais de telles images sont statiques et ne permettent aucune interaction homme-machine ACNUC Les premières versions du système ACNUC ont été éveloppées au Laboratoire de Biométrie et Biologie Évolutive il y a déjà vingt ans (Gouy et al., 1985). Ce système permet de structurer les informations de toute banque de séquences utilisant les formats GenBank, EMBL, SWISS-PROT et PIR. Sous ACNUC, seuls les éléments d information structurés vont pouvoir servir de critères de sélection et pourront être employés pour appeler des séquences lors de la consultation de la banque. Les éléments qui suivent ce principe sont : le nom de la séquence, la longueur en nucléotides, la date d entrée dans la banque, le numéro d accession, les mots-clés, l espèce de provenance ainsi que tous les niveaux de classification taxonomique des organismes, les noms d auteurs, le journal et l année de publication, les références bibliographiques, et enfin les noms des éléments figurant dans les features. ACNUC, dès sa conception a été orienté vers l étude des parties codantes des molécules d acides nucléiques. Une telle organisation permet de définir des séquences en régions fonctionnelles correspondant à des sous-séquences ayant une signification biologique. Ces sous-séquences correspondent aux parties codantes, aux molécules de trna, de rrna, de snrna, et de divers autres types de RNA. ACNUC permet donc de manipuler plus aisément les fragments de séquences définis par rapport à ce type d informations, tout en autorisant l extraction de régions fonctionnelles non définies comme étant des sous-séquences. Il est possible, au moment de la génération des index ACNUC, d utiliser d autres éléments décrits dans les features pour créer des sousséquences. C est ainsi qu HOVERGEN définit comme étant des sous-séquences les introns ou les régions non-codantes situées en 5 ou en 3 des gènes protéiques. Une base ACNUC est constituée par un ensemble de douze index (ACCESS, AUTHOR, BIBLIO, EXTRACT, KEYWORDS, LOCUS, LONGL, SHORTL, SMJYT, SPECIES, SUBSEQ, TEXT) associés aux fichiers plats (en nombre variable suivant la banque considérée) contenant les entrées proprement dites. Ces index sont des fichiers de type binaire à accès direct et ils sont créés à l aide du programme Gener. Leur taille est petite, proportionnellement à celle des fichiers contenant les séquences (7 % dans le cas de GenBank 116). Query, est le programme permettant d accéder aux données d une banque via ces index. Ce logiciel est écrit en C ANSI, ce choix assurant sa portabilité sur une grande variété de machines (stations de travail sous Unix, micro-ordinateurs sous Windows ou MacOS)
28 CHAPITRE I Figure I.10. Capture d écran de la fenêtre principale de l interface de Query telle qu elle apparaît sur une station de travail Sun. L apparence de cette interface peut varier en fonction de la plate-forme utilisée. Depuis 1994, Query intègre une interface graphique écrite à l aide de la bibliothèque Vibrant, développée par le NCBI (Perrière et al., 1994) ; cette interface facilitant l écriture des requêtes et la visualisation des séquences sélectionnées (Figure I.10). Query permet d effectuer des requêtes en utilisant les différents points d entrée listés ci-dessus. Des requêtes complexes, combinant de multiples critères de sélection sont possibles, chaque critère étant lié au(x) suivant(s) par l intermédiaire d opérateurs logiques (ET, OU, NON). Par ailleurs cette interface intègre un système de navigation dans l arbre des espèces ainsi que dans celui des mots-clés. Une notion importante sous Query est celle des listes de séquences ou de mots-clés. Le résultat d une requête est stocké dans une liste, elle-même éventuellement réutilisable pour effectuer de nouvelles recherches. Query permet de sauvegarder aussi bien les listes que les séquences elles-mêmes. Plusieurs formats de sauvegarde sont disponibles pour les séquences : FASTA, le format de la banque et deux formats spécifiques aux programmes développés dans notre équipe (Analseq et Extract). La principale limitation d ACNUC est le fait que ce système ne permet d interroger qu une seule banque à la fois. Par contre, du fait que les bibliothèques de Query sont écrites en C standard, il est très facile d interfacer des banques ACNUC avec des programmes développés par des utilisateurs, ceci pour effectuer des traitements complexes qui ne sont pas autorisés (ou qu il est difficile de réaliser) avec l interface SRS Le système SRS (Sequence Retrieval System) permet d interroger à l aide d une même interface pratiquement n importe quelle collection de séquences disponible sous la forme de fichiers texte (Etzold et al., 1996). À l heure actuelle, c est environ 250 banques de données différentes dispersées sur près de 35 sites Web publics qui sont
29 BANQUES DE DONNÉES interrogeables sous SRS. La possibilité pour ce système d accéder à un aussi grand nombre de banques différentes vient du fait que ses concepteurs ont utilisé un langage de description et d exploration des données qui permet d indexer toute collection structurée. Ce langage de nom ICARUS (Interpreter of Commands and Recursive Syntax) est d abord utilisé pour décrire l ordonnancement des données à l intérieur d une banque quelconque. Une fois cette étape franchie, un interpréteur qui parcourt les fichiers de la dite banque va créer les index qui seront ensuite utilisés par le programme d interrogation associé à SRS. Ces index ont une taille un peu supérieure à ceux d ACNUC, correspondant à environ % de celle de banque associée. Une autre des caractéristiques de SRS est le fait qu ICARUS autorise la création automatique d un réseau de références croisées, permettant la navigation inter-banques (Figure I.11). Cette fonctionnalité fait qu il est possible de relier entre elles des collections ne présentant pas directement des références croisées. Par exemple, il est possible de passer d entrées provenant de PROSITE à des entrées GenBank en utilisant SWISS-PROT comme intermédiaire. L accès aux banques structurées sous SRS peut tout d abord se faire au moyen du programme Getz, portable sur la plupart des machines Unix. Ce programme est écrit en C et s utilise en mode ligne. Cependant, la façon la plus courante d accéder aux banques SRS est sans nul doute au travers de son interface Web : SRSWWW. Un des points forts de cette interface est le fait que les sessions sont spécifiques à l utilisateur et qu elles peuvent être conservées pendant un certain temps sur le serveur. De ce fait, un utilisateur peut commencer à effectuer des requêtes, puis sauvegarder dans ses bookmarks, l adresse de sa page de résultats et y revenir ultérieurement. Tout comme avec Query, une fois une requête résolue, il est possible d utiliser le résultat pour effectuer de nouvelles interrogations afin d affiner la sélection. Des options de sauvegarde des séquences dans différents formats sont également disponibles. ALI DSSP PDBFINDER MIMMAP OMIM ENZYME Blocks PROSITEDOC REBASE PROSITE ProDom SWISSNEW SWISSDOM FSSP HSSP GenBank MOLPROBE NRL_3D PDB YPDREF PMD YPD SWISS-PROT TFSITE ECDC EPD EMBL EMNEW ProtFam PIR FlyGene TrEMBL TrEMBLNEW TFACTOR Figure I.11. Réseau des principales banques interrogeables sous SRS. Ce réseau est en perpétuelle évolution du fait de l apparition et de la disparition de nombreuses collections. Les requêtes sous SRS sont également très puisssantes, avec la possibilité d effectuer des recherches sur le contenu de la totalité des champs figurant dans les annotations. De plus, il est possible d interroger non pas une mais plusieurs banques simultanément, dans ce cas seuls les champs communs aux différentes collections
30 CHAPITRE I sélectionnées peuvent être utilisés pour composer une requête. Là encore, l existence d un réseau reliant les banques offre d intéressantes possibilités. Il est ainsi envisageable de récupérer toutes les séquences répondant à un certain critère dans une banque donnée et qui sont également référencées dans un autre banque. Par exemple, il est possible de récupérer les entrées SWISS-PROT contenant le motif PROSITE de fixation du calcium et pour lesquelles il existe une entrée dans la PDB ; ceci répondant à la question biologique : «Donnez moi toutes les séquences possédant un site de fixation du calcium et dont la structure tertiaire est connue». Une autre fonctionnalité spécifique de SRSWWW est la base de données de banques de séquences DATABANKS. Ce système tient la comptabilité des quelques 250 banques installées sur 35 serveurs SRS publics localisés dans 24 pays. Cette base permettant à un utilisateur de savoir immédiatement sur quel serveur est installée la banque qu il souhaite interroger ENTREZ Entrez est un système de recherche et d extraction de données conçu au NCBI et capable d accéder à des informations de séquences nucléotidiques ou protéiques, aux références bibliographiques associées, et aux collections de séquences génomiques et structurales du NCBI (Schuler et al., 1996 ; McEntyre, 1998). Le maître mot d Entrez est l intégration et l interconnection des sources de données entre elles (Figure I.12). Résumés d Articles (PubMed) Génomes Complets Phylogénie (Taxman) Structures 3D (MMDB) Séquences Nucléiques (GenBank) Séquences Protéiques (GenPept) Figure I.12. Interconnection des données sous Entrez. Les hexagones représentent les banques intégrées, les lignes courbes les liens entre des entrées similaires et les lignes droites les liens entre différents types de données. À partir d une simple interrogation sur l un des trois principaux types de données accessibles (DNA, protéines, bibliographie), Entrez permet de naviguer vers toute autre collection, soit par l intermédiaire de liens directs (e.g., les protéines codées par une séquence nucléotidique), soit par l intermédiaire d un système de «voisins». Cette
31 BANQUES DE DONNÉES possibilité constitue la grande originalité d Entrez par rapport aux autres systèmes d interrogation. Deux sortes de critères sont utilisées pour définir le voisinage entre informations : la ressemblance entre les séquences et la présence de mots-clés communs. Grâce à cette fonctionnalité, il est en particulier possible d effectuer des recherches bibliographiques exhaustives sur une thématique précise en progressant de proche en proche à partir d un unique article de départ. En interne, toutes les banques exploitées par Entrez utilisent le format ASN.1 (Abstract Syntax Notation), qui emploie une syntaxe proche de celle utilisée pour les structures en langage C. Ce choix permet de développer facilement des programmes capables de charger, voire même de compiler les données. Concernant les références bibliographiques, celles-ci proviennent de la banque PubMed qui contient les résumés de la plupart des articles figurant dans Medline, plus quelques enregistrements supplémentaires. Des liens vers les éditeurs proposant le texte complet de leurs articles sont également proposés. Entrez peut être utilisé soit par le biais d un logiciel client, soit par l intermédiaire du serveur Web du NCBI 15. Le logiciel client est écrit en langage C et dispose d une interface construite en utilisant la bibliothèque graphique Vibrant, également distribuée par le NCBI. Ce programme tourne sur la quasi-totalité des plates-formes disponibles, des stations de travail Unix aux micro-ordinateurs. L avantage procuré par l utilisation d un tel outil est bien sûr le fait que les limitations propres aux interfaces écrites en langage HTML sont contournées ACEDB Dans le cadre du projet de cartogaphie (puis de séquençage) du nématode C. elegans, fut conçu un système intégré permettant de gérer les différentes données associées à cet organisme : ACeDB (A C. elegans DataBase) (Thierry-Mieg et al., 1999). Estimant que les systèmes disponibles au moment du lancement de ce projet ne répondaient pas aux besoins, Durbin et Thierry-Mieg (1994) decidèrent de construire de toutes pièces un SGBD ainsi qu une interface graphique permettant d accéder aux données. Ce système tourne sur la quasi-totalité des stations de travail Unix utilisant l environnement XWindows ainsi que sur des PC sous Linux ; un portage sous Windows étant annoncé par les concepteurs. ACeDB possède un noyau de nom Ace ayant une structure de SGBD orienté-objet lui permettant de manipuler des données hétérogènes. Ace a été entièrement écrit en C ANSI, et ce système a été conçu dès le départ dans une optique d évolutivité et d adaptabilité. C est ainsi qu au fil des ans ACeDB a été employé dans le cadre de projets génomes aussi divers que celui des mycobactéries avec MycDB (Mycobacterium Data Base) (Bergh et Cole, 1994), celui d A. thaliana avec AAtDB (An A. thaliana Data Base) (Cherry et al., 1992), ou celui visant à intégrer les données de cartographie de l Homme avec IGD (Integrated Genome Database) (Ritter et al., 1994). Un langage d interrogation et un certain nombre d outils d analyse des séquences ont été progressivement ajoutés à ce système. Du fait de la mise à disposition du code source, de nombreux développeurs se sont attachés à déboguer et améliorer le programme, au risque parfois de créer de multiples versions incompatibles les unes avec les autres
32 CHAPITRE I Une des raisons du succès de ACeDB tient non pas à sa gestion évoluée des données mais plutôt à son interface graphique. Conscients de ce fait, les concepteurs ont veillé à la moderniser au fur et à mesure que les techniques informatiques évoluaient (en particulier avec le développement d Internet). C est ainsi qu une nouvelle architecture de type client/serveur a récemment été conçue, avec trois types de clients écrits respectivement en C, Java ou Perl. Dans cette structure, le serveur tourne sur une machine de type Unix et l utilisateur emploie le client le plus approprié à ses besoins du moment. Le client C (Xclient) comprenant l interface graphique complète d ACeDB, le client Java (Jade2Ace) une interface simplifiée, et le client Perl (AcePerl) s utilisant pour effectuer des requêtes en mode ligne au moyen de scripts
33 DÉVELOPPEMENTS INFORMATIQUES II. DÉVELOPPEMENTS INFORMATIQUES Depuis mon entrée au CNRS, les développements informatiques, plus particulièrement dans le domaine des banques de données, ont constitué une part importante de mon travail. Alors que pendant longtemps les logiciels n ont pas été considérés comme des résultats scientifiques, le développement important de la bioinformatique au cours de ces dernières années a contribué à faire évoluer cet état de fait. La revue Bioinformatics (anciennement Computer Applications in the Biosciences), pionnière dans ce domaine, a depuis été rejointe par In Silico Biology. Par ailleurs, de nombreuses revues plus généralistes (Genome Research, Gene ou Nucleic Acids Research) publient régulièrement des articles sur des logiciels. Ce chapitre se propose de décrire les différents développements informatiques auxquels j ai participé et qui peuvent être considérés comme des résultats à part entière. 1. LE SERVEUR DU PBIL 1.1. MISE EN PLACE Du fait de l extraordinaire développement d Internet grâce au langage HTML et au protocole HTTP il était clair, dès 1995, que la mise en place de serveurs Web allait constituer un moyen incontournable d accès aux logiciels et aux bases de données produits dans le domaine de la bioinformatique. C est pourquoi, dès cette date, nous avons développé WWW-Query, un service permettant de consulter les banques généralistes ainsi que les banques spécialisées développées dans notre groupe (Perrière et Thioulouse, 1996 ; Perrière et Gouy, 1996). Par la suite, ce système a été étendu au cours de la constitution du PBIL 1 (Pôle Bioinformatique Lyonnais) en Le PBIL est une structure qui a été mise en place par le COMI (Comité des Moyens Informatiques du CNRS), et qui a pour but de favoriser la diffusion et l utilisation des bases de données et des logiciels produits à Lyon dans le domaine de la bioinformatique. Le PBIL implique essentiellement deux groupes, d une part l équipe «Conformation des Protéines» dirigée par Gilbert Deléage à l Institut de Biologie et de Chimie des Protéines et d autre part l équipe «Biométrie Moléculaire, Évolution et Structure des Génomes» dirigée par Manolo Gouy au Laboratoire de Biométrie et Biologie Évolutive. L équipe de G. Deléage s occupant des questions liées au protéome et celle de M. Gouy, des questions liées au génome. N ayant participé qu à certains développements parmi tous ceux consacrés à la composante «génomique» du serveur, je ne ferai donc état que de ceux-ci
34 CHAPITRE II Il est important de noter que si les deux composantes du PBIL sont géographiquement séparées, le service Web proposé permet de passer de la partie génome à la partie protéome de façon complètement transparente pour l utilisateur, grâce à des passerelles informatiques échangeant les données entre les machines ACCÈS AUX BANQUES La première ambition du serveur du PBIL est de proposer un moyen simple et performant de consultation des banques de séquences. Pour ce faire nous avons construit une interface Web permettant d accéder à des banques structurées au moyen d ACNUC. Il est ainsi possible d interroger aussi bien les collections généralistes comme EMBL, GenBank, PIR ou SWISS-PROT/TrEMBL, que les banques spécialisées développées dans notre groupe comme NRSub, EMGLib, HOVERGEN ou HOBACGEN RÉCUPÉRATION DES SÉQUENCES Le système d interrogation utilisé est constitué par un ensemble de scripts CGI, écrits en langage C à partir des bibliothèques d ACNUC. Le script principal permet de construire des requêtes complexes utilisant les mnémoniques ou les numéros d accession, des mots clés, des données taxonomiques, des références bibliographiques, la nature des régions fonctionnelles présentes dans les génomes, etc. (Figure II.1). Jusqu à quatre critères sont utilisables simultanément, ces critères étant reliés au moyen de connecteurs logiques. Species Send Modify Retrieve Help Databank: EMGLib Selection criteria: 1. DEFAULT Species Bacillus subtilis 2. AND Type CDS 3. AND NOT Keyword partial 4. AND Keyword List name: list SUBMIT CLEAR Figure II.1. Interface de composition des requêtes permettant d interroger les banques du PBIL. Dans cet exemple l utilisateur demande a récupérer toutes les parties codantes non partielles de B. subtilis figurant dans EMGLib. Le résultat de chaque requête est inscrit dans une liste qui est stockée sur le serveur jusqu à minuit heure locale. Les listes créées sont elles-mêmes utilisables pour construire d autres requêtes et il est ainsi possible d affiner petit à petit une sélection
35 DÉVELOPPEMENTS INFORMATIQUES L introduction de cette fonctionnalité permet également une utilisation beaucoup plus souple des programmes d analyse des séquences disponibles sur le serveur. En effet, l utilisateur a la possibilité de lancer ces programmes non pas sur une seule séquence, comme cela se fait sur la plupart des serveurs de ce genre, mais sur une liste complète. De nombreuses fonctions permettent de modifier les listes créées. On peut ainsi effectuer des sélections tenant compte de la longueur des séquences, de leur date d insertion dans la banque, de la présence d une chaîne de caractères particulière dans les annotations, etc. Une fois une liste de séquences générée (et éventuellement modifiée), il est possible de télécharger les entrées correspondantes dans divers formats. Ceci peut se faire directement au niveau d une page chargée par le navigateur Web ou par l intermédiaire d un transfert par FTP, si le nombre de séquences est trop élevé RECHERCHE DE SIMILARITÉS ET ALIGNEMENTS Une fonction de recherche de similarités au moyen du programme BLAST2 (Altschul et al., 1997) est également implémentée. Par rapport aux nombreux services équivalents proposés sur d autres sites, la version disponible sur le serveur du PBIL présente certaines originalités. Les résultats d une recherche peuvent être filtrés au moyen de différents outils là encore développés au moyen des bibliothèques d ACNUC. Il est ainsi possible de récupérer les séquences en fonction de leur appartenance à certains groupes taxonomiques, de la présence de certains mots clés ou de leur date d insertion dans la banque. Plusieurs filtrages successifs sont possibles afin de spécifier petit à petit la recherche. L utilisateur peut sélectionner un ensemble de séquences provenant de la sortie de BLAST2 (ainsi que la séquence requête) pour en effectuer l alignement au moyen de versions en ligne des programmes CLUSTAL W (Tompson et al., 1994 ; Higgins et al., 1996) ou MULTALIN (Corpet, 1988). C est à ce niveau qu intervient le protocole de communication que nous avons établi entre les deux sites constituant le serveur du PBIL. En effet, ces programmes d alignement multiples ont été implémentés sur la partie protéome et non sur la partie génome du PBIL. Le programme d alignement par paires LFASTA a également été interfacé avec les collections GenBank, EMBL et HOVERGEN du serveur. Ce programme permet d effectuer des alignements locaux entre des paires de séquences nucléotidiques, que ces séquences proviennent des banques ou qu elles soient rentrées par l utilisateur. Nous verrons infra, que cette implémentation de LFASTA gagne à être utilisée en conjonction avec le programme LALNVIEW, développé dans notre équipe AUTRES FONCTIONNALITÉS D autres types de recherches sont également disponibles. Nous avons ainsi développé un système de navigation dans la classification taxonomique des espèces. Ce système permet, à partir d un nom de taxon rentré par l utilisateur, de visualiser et d explorer les nœuds ascendants (ou descendants) conduisant à (ou partant de) ce taxon, la profondeur descendante étant paramétrable par l utilisateur. Chaque nœud est représenté sous la forme d un lien hypertexte permettant à son tour de remonter ou de descendre dans la structure de l arbre. Les banques disponibles sur le serveur du PBIL pour lesquelles il est possible d explorer la taxonomie sont GenBank, EMBL et HOVERGEN, l arbre des espèces utilisé étant celui du NCBI. Une fonction de recherche par croisement de taxon est utilisable sur les banques de familles de gènes homologues développées dans notre équipe (HOVERGEN et HOBACGEN). Cette fonction est applicable à n importe quel niveau taxonomique
36 CHAPITRE II pour les espèces figurant dans la banque sélectionnée. Par exemple, dans le cas d HOBACGEN, on peut demander de récupérer toutes les familles comprenant au moins une séquence de B. subtilis, une séquence d entérobactérie et une séquence d archée. La sortie du programme est affichée sous la forme d une page HTML contenant l ensemble des numéros de familles, chacun de ces numéros apparaissant comme un lien hypertexte permettant d accéder à la liste des séquences appartenant à la famille ANALYSE MULTIVARIÉE Parmi les méthodes installées sur le serveur, les plus spécifiques sont celles ayant trait à l analyse multivariée. En effet, ces techniques restent encore d un emploi difficile pour les non-biométriciens, et les logiciels donnant accès aux principaux programmes de ce type (comme SAS, SYSTAT ou SPLUS) ne sont pas vraiment adaptés à l étude des séquences biologiques. Les méthodes de ce genre disponibles sur le serveur et utilisables pour analyser des séquences sont l AFC (Analyse Factorielle des Correspondances), l ACO (Analyse en Coordonnées principales) et l ADC (Analyse Discriminante des Correspondances). Les sources de ces méthodes proviennent du package ADE-4, dédié à l analyse des données en écologie (Thioulouse et al., 1997) ; les programmes ayant été adaptés pour pouvoir être interfacés avec le Web (Thioulouse et Chevenet, 1996 ; Perrière et Thioulouse, 1996 ; Perrière et Gouy, 1996). Par ailleurs, du fait que l interprétation des résultats retournés par des méthodes d analyse multivariée est essentiellement graphique, nous avons implémenté un programme de tracé de graphes sur le serveur. Ce programme, basé sur le logiciel du domaine public GNUplot, est limité à la visualisation, sous forme d images bitmap, des plans factoriels d une analyse. Il ne peut donc être utilisé que pour avoir un aperçu rapide des résultats. Les différents exemples de plans factoriels donnés dans cette section ont tous été tracés à l aide de cet outil. Si l utilisateur désire analyser plus en profondeur ses résultats, il lui est possible de récupérer les fichiers de sortie des programmes utilisés. En effet, tous ces fichiers sont stockés sur le serveur du PBIL, ce qui permet de les télécharger facilement ANALYSE FACTORIELLE DES CORRESPONDANCES L AFC est une méthode qui a été fréquemment utilisée en analyse de séquences, en particulier dans notre équipe. Historiquement, la première utilisation de l AFC dans ce domaine a été l étude comparative de l usage des codons entre différentes espèces (Grantham et Gautier, 1980 ; Grantham et al., 1980a, 1980b). Ces travaux ont permis à Grantham et ses collègues de formuler leur fameuse «hypothèse du génome» stipulant que chaque organisme utilise préférentiellement certains codons synonymes. Par la suite, l AFC a été fréquemment utilisée dans la détection des biais dans l usage des codons au sein d un organisme donné, ces biais étant fréquemment liés à l expressivité des gènes (Holm, 1986 ; Sharp et al., 1986 ; Shields et Sharp, 1987 ; Shields et al., 1988 ; Médigue et al., 1991 ; Perrière et al., 1994 ; Stenico et al., 1994 ; McInerney, 1997 ; Lafay et al., 1999). Cette méthode a également été employée pour de nombreuses autres études comme la prédiction de la localisation de séquences codantes (Fichant et Gautier, 1987) ; l étude de l évolution des séquences répétées du génome des primates ou des rongeurs (Quentin, 1988, 1989) ; la détermination les facteurs guidant la composition en acides aminés des protéines d E. coli (Lobry et Gautier, 1994) ; ou la détection d erreurs de séquençage (Fichant et Quentin, 1995)
37 DÉVELOPPEMENTS INFORMATIQUES Figure II.2. Croisement des deux premiers facteurs d une AFC réalisée sur l ensemble des gènes de B. burgdorferi. La dichotomie observée correspond à la localisation des gènes sur l un ou l autre des brins du chromosome. Le programme permettant de réaliser des AFC sur des séquences biologiques disponibles sur le serveur du PBIL utilise uniquement des fréquences absolues de codons ou d acides aminés. Il s agit donc de tableaux d effectifs X = [x ij ] à n lignes et p colonnes, n étant le nombre de gènes ou de protéines et p étant le nombre de codons (61) ou d acides aminés (20). Les notations utilisées sont celles classiquement employées pour l AFC : n p x = x ij x j = x ij x i = x ij i =1 j =1 Ce programme ne travaille pas sur les fréquences relatives par codons synonymes ou les RSCU (Relative Synonymous Codon Usage). En effet, en toute rigueur, l AFC ne doit être utilisée que pour traiter des tableaux d effectifs pour lesquels les «marges» (les valeurs de x i et x j ) ont une signification (Hill, 1974). Ceci étant lié au fait que cette méthode effectue une pondération des lignes et des colonnes en utilisant ces marges. Dans le cas de tableaux de fréquences relatives ou de RSCU, la pondération n a aucun sens puisque les marges des lignes sont toutes égales entre elles. Par ailleurs, l AFC utilisant une mesure de distance entre individus basée sur la métrique du χ 2, cette mesure perd toute signification lorsque l on utilise des tableaux qui ne sont pas des tableaux d effectifs. Les étapes nécessaires pour effectuer une AFC sur des séquences biologiques à partir du serveur du PBIL sont les suivantes : (i) sélection d un ensemble de gènes ou de protéines au moyen du système d interrogation ; (ii) calcul des fréquences de codons ou d acides aminés ; (iii) calcul de l AFC proprement dite. Une fois l AFC calculée, une page contenant des indications sur l analyse effectuée apparaît (valeurs propres, nom des fichiers contenant les résultats, etc.) Par défaut, le programme ne conserve que les quatre premiers facteurs de l analyse, l expérience montrant qu il n est jamais nécessaire d explorer les facteurs plus lointains dans le cas des données n i =1 p j =1-33 -
38 CHAPITRE II de séquences. Un exemple d AFC réalisée sur la composition en codons des gènes de Borrelia burgdorferi est donné sur la Figure II.2. Indépendamment de la fonction calculant une AFC sur la composition en codons ou en acides aminés d un ensemble de gènes protéiques, nous avons également implémenté une version en ligne du programme RecSta (Fichant et Gautier, 1987). Ce programme permet de détecter la présence de parties codantes dans une séquence en utilisant l AFC. Le principe de cette méthode est le suivant : la séquence génomique dans laquelle on désire rechercher des parties codantes est découpée en trois séries de blocs chevauchants d une longueur fixée par l utilisateur, chaque série correspondant à l un des cadres de lecture possible. La longueur des blocs, ainsi que la taille de la zone de chevauchement, doivent être des multiples de trois afin de conserver la phase entre les différents blocs d une même série. Les valeurs standard pour la plupart des analyses sont une longueur de 90 nucléotides pour les blocs et un chevauchement de 30 nucléotides. Figure II.3. Prédiction de la localisation de parties codantes dans la séquence de l opéron phénylalanyl-trna synthétase d E. coli (numéro d accession K02844). En abscisse figure le numéro de bloc et en ordonnée le score de ce bloc sur le premier facteur de l AFC. Cinq parties codantes sont détectées par la méthode et leur localisation est indiquée par les barres horizontales au sommet du graphe. Une fois ce découpage effectué, le programme calcule la fréquence des codons et crée un tableau dans lequel se trouvent en ligne les trois groupes de blocs (correspondant respectivement aux trois cadres de lecture) et en colonnes les différents codons. Une AFC est ensuite calculée sur ce tableau. Si, dans l un des trois cadres de lecture possible se trouve une partie codante, la composition en codons des blocs figurant dans cette phase sera biaisée si on la considère par rapport à la composition en trinucléotides des deux autres phases possibles. De ce fait, les scores factoriels de l AFC pour les blocs situés dans la phase contenant cette partie codante se distingueront nettement de ceux des blocs situés dans les deux autres phases. Par contre, dans le cas où aucune partie codante n est présente, les scores factoriels des blocs dans les trois phases seront indiscernables. La Figure II.3 présente une recherche
39 DÉVELOPPEMENTS INFORMATIQUES effectuée sur une séquence d E. coli contenant cinq gènes protéiques regroupés à l intérieur d un opéron ANALYSE DISCRIMINANTE DES CORRESPONDANCES L ADC est une extension récente de l Analyse Discriminante (AD) lui permettant de travailler sur des tableaux d effectifs. Son modèle théorique a été proposé par Yoccoz (1988), et son implémentation a été réalisée par Thioulouse et al. (1997) dans un des modules d ADE-4. D un autre côté, l ADC peut être également vue comme une extension de l AFC dans le sens qu elle permet de traiter des individus dont on sait a prori qu ils appartiennent à différents groupes. L AD «classique» a été fréquemment utilisée en analyse des séquences, ceci pour différentes tâches. Elle a été employée pour étudier les propriétés de certaines séquences protéiques (Klein et al., 1984, 1986), pour prédire la localisation des jonctions d épissage dans les mrna (Nakata et al., 1985 ; Iida, 1988), pour discriminer les segments de structures secondaires de protéines (Kanehisa, 1988), pour distinguer les protéines intracellulaires et extracellulaires (Nakashima and Nishikawa, 1994), et pour localiser les exons internes dans le DNA humain (Solovyev et al., 1994). Les jeux de données utilisables sur le serveur du PBIL sont du même type que ceux de l AFC, à savoir des tableaux d effectifs à n lignes et p colonnes, de terme général x ij. La différence est que ces tableaux sont partitionnés en q classes. Le but d une méthode comme l ADC est essentiellement prédictif. En effet, le principe en est de déterminer la combinaison linéaire des variables permettant d obtenir la meilleure discrimination entre les classes. Une fois les facteurs calculés, il est possible de projeter des individus supplémentaires dans l analyse, ceci afin de les rattacher à une classe donnée. Avec l ADC il existe en effet une relation directe entre la valeur des scores factoriels des individus et ceux des variables : u i (k) = x x i p j =1 v j (k)x ij où u i (k) et v j (k) sont respectivement les scores pour un individu i et pour une variable j sur le facteur k de l analyse (1 k q 1). Chaque facteur d une ADC permet théoriquement de séparer l une des q classes des q 1 autres. En utilisant les distributions des scores sur un facteur k donné, on défini le seuil s(k) permettant de classer ou non un individu dans cette classe : x j s(k) = + q q q q + q q où, q q et, q q sont les moyennes et les écarts types des scores sur le facteur k pour la classe q d une part, et pour l ensemble des autres classes d autre part. La réalisation d une ADC sur le serveur du PBIL requiert tout d abord d effectuer une AFC sur le jeu de données de départ. Une fois ceci fait, il est nécessaire d initialiser l ADC en utilisant les résultats de l AFC précédente ainsi qu un fichier de catégories indiquant la correspondance entre les lignes du tableau de départ et leur classe d appartenance. L étape suivante est le calcul de l ADC proprement dite ; une fois ce calcul effectué, l utilisateur a la possibilité d effectuer un test de permutations lui permettant de tester la qualité de la discrimination obtenue sur le jeu de données. Ce
40 CHAPITRE II test consiste en une suite de permutations aléatoires des lignes entre les classes, suivie à chaque fois d un recalcul de l ADC. Figure II.4. Croisement des deux premiers facteurs d une ADC réalisée sur la composition en tétranucléotides des génomes de E. coli, Haemophilus influenzae et B. subtilis découpés en fragments de 7 kb. Le premier facteur sépare les séquences de B. subtilis (valeurs positives sur l axe des abscisses) tandis que le deuxième facteur sépare les séquences d E. coli (valeurs positives sur l axe des ordonnées). Une fois les calculs terminés il est possible de visualiser les plans obtenus par les croisements des différents facteurs de l AFC préparatoire ou de l ADC proprement dite (Figure II.4). La visualisation d un plan factoriel ne sera possible que si le nombre de classes définies au départ est supérieur ou égal à trois puisque le nombre de facteurs produits par une ADC est égal à q ANALYSE EN COORDONNÉES PRINCIPALES L ACO est une méthode d ordination utilisant des matrices de distances qui a été développée par Gower (1966). L ACO peut être utilisée, à partir d alignements multiples, pour compléter des analyses phylogénétiques conduites avec des techniques plus classiques (Higgins, 1992 ; Perrière et Gouy, 1996), mais elle ne doit pas ellemême être considérée comme une méthode de reconstruction phylogénétique. Elle est surtout utile lorsque l on désire visualiser les grands groupes taxonomiques dans un échantillon comprenant plusieurs centaines de séquences. La principale contrainte régissant l utilisation de l ACO est que la matrice de distance utilisée doit être positive et semi-définie, c est-à-dire que ses valeurs propres ne doivent pas être négatives. Ceci peut être garanti facilement si la distance utilisée est euclidienne. Lorsque l on considère un alignement multiple, une distance de ce type peut être obtenue avec la matrice reportant, pour chaque paire de séquences, le rapport entre le nombre de différences observées et le nombre total de sites (compte non tenu des positions où il existe des gaps). Cette distance est souvent utilisée en phylogénie où elle représente le pourcentage de divergence sans l introduction de corrections pour
41 DÉVELOPPEMENTS INFORMATIQUES les substitutions multiples. Dans le cas des séquences nucléotidiques il est parfaitement légitime d utiliser ce genre de mesure. Par contre, dans le cas des protéines il est souvent préférable de pondérer les différences en tenant compte des similarités plus ou moins grandes qui existent entre les acides aminés. E 1 K 2 2 R H N Q S T I L V F W Y C M A G P D E K R H N Q S T I L V F W Y C M A G Table II.1. Matrice du nombre de mutations nécessaires pour passer d un acide aminé à un autre. La façon la plus simple de procéder serait d utiliser des matrices de pondération traditionnellement utilisées par les programmes d alignement multiples comme PAM (Dayhoff et al., 1978) ou BLOSUM (Henikoff et Henikoff, 1992). Le problème est que les distances construites à partir de telles matrices ne sont pas euclidiennes. En effet, il est fréquent que la mesure de distance entre un acide aminé et lui-même soit non nulle, ceci du fait des corrections introduites pour prendre en compte l existence de substitutions multiples au cours de l évolution. Une solution est d utiliser la matrice de Fitch (1966), qui mesure le nombre de mutations requises pour passer d un acide aminé à un autre (Table II.1). Cette matrice possède une structure hiérarchique sur trois niveaux, et son utilisation permet d obtenir une distance euclidienne. La principale critique que l on puisse faire à cette approche est bien sûr qu elle introduit de grandes simplifications au niveau des relations évolutives existant entre les acides aminés. Une possibilité alternative serait de modifier les mesures de similarité définies par les matrices PAM ou BLOSUM de façon à rendre euclidiennes les distances obtenues. Il existe en effet des méthodes de transformation basées sur l utilisation de la plus forte valeur propre négative. D autre part, tout comme dans la plupart des méthodes de reconstruction phylogénétiques, la présence de gaps dans l alignement n est absolument pas prise en compte dans le calcul des distances, le programme ignorant les sites où se trouve une insertion ou une délétion. La réalisation d une ACO sur le serveur du PBIL nécessite de disposer d un jeu de séquences alignées au format MASE (Faulkner et Jurka, 1989). Ce format est celui utilisé par les programmes SEAVIEW et PHYLO_WIN développés dans notre laboratoire (Galtier et al., 1996). La première étape de l analyse est le transfert de cet alignement sur le serveur, les distances entre chaque paire de séquences étant ensuite calculées. Dans le cas de séquences protéiques l utilisateur a la possibilité d utiliser le
42 CHAPITRE II nombre de différences ou les distances pondérées de la Table II.1. Une fois ceci fait, l ACO proprement dite est calculée. Là encore, seuls les quatre premiers facteurs de l analyse sont conservés. Figure II.5. Croisement des deux premiers facteurs d une ACO réalisée sur un alignement de 31 séquences protéiques d insuline provenant de la banque HOVERGEN (numéro d accession FAM000008). La séparation des grands groupes taxonomiques est bien retrouvée sur ce plan factoriel. Cette méthode permet également le tracé de plans factoriels. Un exemple de plan réalisé à partir d une ACO calculée sur un alignement de séquences d insuline de vertébrés est donné sur la Figure II APPLICATIONS ASSOCIÉES Nous avons vu que, si l apparition du Web a permis de mettre en ligne de très nombreux programmes et banques de données de séquences, les limitations du langage HTML ne permettent pas de construire des interfaces utilisateurs évoluées (cf. I.3.1). Ceci pose d importants problèmes, en particulier lorsque les résultats retournés par une méthode sont essentiellement graphiques (comme une prédiction de structure 3D de protéine), ou requièrent une forte interaction avec l utilisateur (par exemple un arbre phylogénétique). Plusieurs solutions techniques sont disponibles pour pallier ces limitations : développement de plugs-in pour les navigateurs Web, d applets Java ou utilisation de programmes externes associés. Cette dernière solution étant la plus simple à mettre en œuvre, c est elle qui a été retenue pour certains services proposés par le PBIL. Cette approche permet en effet d employer des logiciels pouvant être utilisés en dehors du contexte spécifique d un serveur Web, ce qui n est pas le cas des plugs-in et des applets. De plus, l utilisation d applets impose le chargement du programme à chaque nouvel accès sur le site, ce qui nécessite parfois un temps très important si l utilisateur se connecte depuis un client géographiquement lointain
43 DÉVELOPPEMENTS INFORMATIQUES L utilisation d applications de ce type pose le problème du fait que leur emploi signifie la perte de la possibilité d utiliser l intégralité des fonctionnalités d un serveur sur n importe quelle architecture. En effet, ces applications tournent sur la machine cliente et non plus sur le serveur, et il est donc nécessaire de concevoir autant de portages que de plates-formes sur lesquelles on désire les voir fonctionner. Échelle de similarité 20 % 30 % 40 % 50 % 60 % 70 % 80 % 90 % 100 % U D Figure II.6. Comparaison des séquences génomiques de DHST chez l Homme (D26535 ) et chez F. rubripes (U40758). L alignement a été effectué au moyen de LFASTA puis il a été visualisé par LALNVIEW. Plusieurs logiciels pouvant être utilisés en conjonction avec les services proposés par le PBIL ont donc été développés dans notre groupe : NJplot (Perrière et Gouy, 1996), SEAVIEW et LALNVIEW (Duret et al., 1996). NJplot permet de tracer des arbres phylogénétiques tandis que SEAVIEW est un éditeur d alignements multiples. Lors de la visualisation d une séquence provenant des banques HOVERGEN ou HOBACGEN ces deux programmes peuvent être utilisés en tant qu applications externes permettant d afficher l arbre ou l alignement associés à la famille à laquelle appartient cette séquence. LALNVIEW, quant à lui, permet de visualiser des similarités locales obtenues en effectuant des alignements par paires. La Figure II.6 donne un exemple d utilisation de LALNVIEW. Sur cette figure est représenté l alignement de la séquence génomique de la dihydrolipoamide succinyltransférase (DHST) humaine avec celle du poisson Fugu rubripes. Au cours de l évolution de ce gène, seules les régions correspondant aux exons sont restées conservées mais pas les introns. Par ailleurs, le gène de la DHST est beaucoup plus compact chez F. rubripes que chez l Homme, ceci du fait de la présence d introns plus courts. De ce fait, il n est pas possible de calculer un alignement global entre ces deux séquences, et donc il est nécessaire d utiliser un programme d alignement local comme LFASTA. LALNVIEW peut alors être employé pour visualiser les régions homologues entre ces deux séquences. Une autre fonctionnalité intéressante de ce programme est la possibilité de représenter graphiquement les différentes régions des deux séquences alignées lorsque celles-ci proviennent d une banque. LALNVIEW est en effet susceptible, lorsqu il fonctionne en association avec les serveurs du PBIL ou ExPASy, de récupérer les informations figurant dans les annotations des entrées afin de les représenter dans son interface graphique au moyen d un code couleur. Grâce à cette fonctionnalité, il est possible de mettre en relation les similarités visualisées et la structure des séquences (positions des introns et des exons, présence d éléments régulateurs, domaines
44 CHAPITRE II protéiques, etc.) LALNVIEW peut être utilisé en association avec le serveur du PBIL 2 pour visualiser des alignements de séquences nucléotidiques réalisés avec LFASTA, et en association avec le serveur ExPASy 3 pour visualiser des alignements de séquences protéiques réalisés avec SIM (Huang et Miller, 1991) BILAN DE L UTILISATION DU SERVEUR Depuis la mise en service de WWW-Query en mai 1995 le nombre de connexions au serveur n a cessé de croître et leur total dépasse , avec un nombre moyen de connexions par jour en mars 2000 (Figure II.7). L ensemble de ces connexions a été établi depuis plus de machines différentes. Également, c est plus de sites extérieurs qui pointent vers les différents services du PBIL Connexions ( ) 0 05/95 02/96 11/96 08/97 05/98 01/99 10/99 Date Figure II.7. Évolution du nombre de connexions sur la composante «génomique» du serveur du PBIL depuis sa mise en service en mai Cette croissance suit une tendance générale, directement proportionnelle au nombre de personnes ayant accès au réseau Internet, mais elle traduit également le fait que le serveur du PBIL est perçu comme une source de données fiable et utile. 2. EMGLIB EMGLib a été développée dans la continuité de la banque NRSub, dédiée au génome de B. subtilis (Perrière et al., 1998) et pour laquelle une attention particulière avait été portée à la qualité des annotations. Après l obtention du génome complet de cette bactérie (Kunst et al., 1997), j ai décidé d étendre la structure de NRSub pour lui permettre de gérer non pas un, mais des génomes bactériens. C est ainsi qu EMGLib a été construite à la suite d une collaboration avec l équipe «Mathématiques et Informatique des Génomes», dirigée par Philippe Bessières au Laboratoire de Génétique Microbienne (Centre INRA de Jouy-en-Josas), et l équipe «Évolution
45 DÉVELOPPEMENTS INFORMATIQUES Moléculaire et Génomique», dirigée par Bernard Labedan à l Institut de Génétique et Microbiologie (Université Paris Sud) MOTIVATION L obtention, il y a cinq ans, de la séquence de la bactérie H. influenzae (Fleischmann et al., 1995) a fait basculer la bioinformatique dans l ère des génomes complets. Depuis cette date, 28 organismes procaryotes ont vu leur génome complètement séquencé, et plus de 100 projets sont en cours voire quasiment terminés. Or, un des gros problèmes liés à l apparition de ces très grandes séquences est le fait que la qualité des annotations est pauvre, si on la compare à celle de fragments de plus petite taille. Certaines informations utiles, pourtant faciles à déterminer, ne figurent que rarement dans les annotations des génomes complets : références croisées avec d autres banques, position sur la carte génétique, biais d usage du code, localisation des gènes sur l un ou l autre des brins du chromosome, etc. Par ailleurs, du fait des limitations de certains logiciels d analyse (cf. I.1.3), ces génomes sont systématiquement intégrés sous la forme de grands fragments chevauchants dans les banques généralistes, ce qui complique singulièrement la tâche de l accès aux données. Ceci alors même que le NCBI distribue des fichiers au format GenBank contenant ces génomes assemblés en un seul contig. Certes, il existe de nombreux sites Web permettant d accéder aux séquences des génomes complets, le problème étant que ces serveurs ne permettent pas de récupérer facilement l intégralité des gènes pour en effectuer l analyse. Qui plus est, la récupération d un génome complet ne facilite en rien les traitements ultérieurs que l on peut effectuer dessus, ceci faute d un système d interrogation approprié. Dans le but de pallier ces problèmes nous avons développé EMGLib. D une part cette banque intègre quelques ajouts au niveau des annotations ; ces ajouts étant le plus souvent en liaison directe avec nos préoccupations en matière de recherche. D autre part, l accès aux données est facilité par l utilisation de systèmes de gestion de bases de données capables de manipuler de grandes séquences et leurs informations associées. C est ainsi que EMGLib peut être consultée au moyen des systèmes ACNUC, Micado et SRS. Par ailleurs, la version sous ACNUC est distribuée par FTP, et sa relative petite taille lui permet d être installée sur des postes de type microordinateurs CONTENU DE LA BANQUE SÉQUENCES La première version d EMGLib a été rendue publique en août 1998 (Perrière et al., 1999). Toutes les séquences figurant dans EMGLib proviennent de la division génome du NCBI 4, à l exception de celle de B. subtilis qui provient de NRSub. La version 2.2 d EMGLib contient 49 entrées correspondant à 28 génomes procaryotes, les 16 chromosomes du génome complet de S. cerevisiae, et les chromosomes II et III de Plasmodium falciparum (Table II.2). Ces 49 entrées totalisent pb, elles contiennent gènes protéiques, trna, 191 rrna, 38 snrna et 27 RNA divers. 4 ftp://ncbi.nlm.nih.gov/genbank/genomes
46 CHAPITRE II Espèce Taille Gènes Référence Aeropyrum pernix K Kawarabayasi et al. (1999) Aquifex aeolicus VF Deckert et al. (1988) Archaeoglobus fulgidus DSM Klenk et al. (1997) Bacillus subtilis Kunst et al. (1997) Borrelia burgdorferi B Fraser et al. (1997) Campylobacter jejuni NCTC Parkhill et al. (2000) Chlamydia muridarum Read et al. (2000) Chlamydia pneumoniae CWL Kalman et al. (1999) Chlamydia trachomatis Stephens et al. (1998) Chlamydophila pneumoniae AR Read et al. (2000) Deinococcus radiodurans R White et al. (1999) Escherichia coli K-12 MG Blattner et al. (1997) Haemophilus influenzae Rd Fleischmann et al. (1995) Helicobacter pylori J Alm et al. (1999) Helicobacter pylori Tomb et al. (1997) Methanobacterium thermoautotrophicum H Smith et al. (1997) Methanococcus jannaschii DSM Bult et al. (1996) Mycobacterium tuberculosis H37Rv Cole et al. (1998) Mycoplasma genitalium G Fraser et al. (1995) Mycoplasma pneumoniae M Himmelreich et al. (1996) Neisseria meningitidis MC Tetellin et al. (2000) Pyrococcus horikoshii OT Kawarabayasi et al. (1998) Pyrococcus abyssi GE Non publié Rickettsia prowazekii Madrid E Andersson et al. (1998) Saccharomyces cerevisiae S288C Goffeau et al. (1996) Synechocystis sp. PCC Kaneko et al. (1996) Thermotoga maritima MSB Nelson et al. (1999) Treponema pallidum Nichols Fraser et al. (1998) Ureaplasma urealyticum Non publié Table II.2. Liste des espèces dont le génome complet figure dans EMGLib. La taille des génomes est donnée en kb. Un certain nombre de modifications sont effectuées sur les identifiants utilisés dans les entrées originales de GenBank, ceci dans le but d homogénéiser la nomenclature employée dans EMGLib. Tout d abord un nouveau nom (champ LOCUS), basé sur le format xxxxxcg ou xxchrnnnn, est donné aux séquences. Le premier format est utilisé pour les espèces ne possédant qu un seul chromosome. Dans ce cas, xxxxx correspond à l abréviation du nom de l espèce considérée sur cinq caractéres maximum. Ainsi BACSUCG est le nom de l entrée correspondant au génome de B. subtilis. Le second format est utilisé pour des génomes (procaryotes ou eucaryotes) contenant plusieurs chromosomes. Dans ce cas, xx correspond au nom de l espèce, codé sur deux caractères, et nnnn au numéro du chromosome en chiffres romains. Par exemple, SCCHRIX est le nom de l entrée correspondant au chromosome IX de S. cerevisiae. Les numéros d accession GenBank (champ ACCESSION) sont également remplacés par des numéros propres à EMGLib. Ceux-ci sont basés sur le format CGnnnn, où nnnn est simplement un numéro correspondant à l ordre d intégration de la séquence dans la banque. Les numéros d accession originaux sont cependant conservés, et placés dans un descripteur /db_xref figurant au début du champ FEATURES USAGE DU CODE Parmi les informations spécifiques qui sont intégrées dans EMGLib figurent, pour chaque partie codante, la valeur du CAI (Codon Adaptation Index), l indice d usage
47 DÉVELOPPEMENTS INFORMATIQUES des codons défini par Sharp et Li (1987). En effet, cet indice est conçu de telle façon qu il peut servir d estimateur de l expressivité des gènes. Qui plus est, la table de référence du CAI peut aisément être déterminée pour un organisme unicellulaire. La formule permettant de calculer cet indice pour un gène i donné est : 59 ln c i = 1 x x ij ln w j i où c i est la valeur du CAI, x i le nombre total de codons du gène, x ij le nombre de codons j, et w j le rapport entre la fréquence du codon j et la fréquence du codon synonyme majeur pour l acide aminé considéré, ce rapport ayant été estimé dans un ensemble de gènes de référence, supposés hautement exprimés. Les codons non dégénérés AUG et UGG, correspondant respectivement à la méthionine et au tryptophane, n interviennent donc pas dans ce calcul. Le choix des codons majeurs étant très variable d un organisme à un autre (Grantham et al., 1980a, 1980b), il est nécessaire d établir une table de valeurs de w j pour chaque espèce considérée. Si ces valeurs ont d ores et déjà été établies chez E. coli (Sharp et Li, 1987), B. subtilis (Shields et Sharp, 1987 ; Sharp et al., 1990 ; Perrière et al., 1994), S. cerevisiae (Sharp et al., 1986) ou H. influenzae (Perrière et Thioulouse, 1996), elles l ont parfois été sur des échantillons de gènes très réduits. C est pourquoi nous avons décidé d établir nos propres tables de références pour les valeurs de CAI introduites dans EMGLib. Chez les organismes unicellulaires, il est depuis longtemps établi que les protéines ribosomales figurent parmi celles qui sont susceptibles d être hautement exprimées (Ikemura, 1981 ; Gouy et Gautier, 1982 ; Holm, 1986 ; Sharp et al., 1986). Afin de localiser un ensemble de gènes suffisamment important pour nous permettre d établir notre table de w j, nous avons donc décidé d utiliser ces protéines ribosomales comme indicatrices. Pour ce faire, nous avons réalisé, pour chaque génome, une AFC calculée sur l ensemble de ses gènes de longueur 150 pb. Sur chacune de ces AFC nous avons alors identifié le ou les facteur(s) séparant les gènes codant pour les protéines ribosomales des autres. Puis nous avons récupéré les gènes présentant les scores les plus élevés sur ce(s) facteur(s), ceci jusqu à l obtention d un ensemble dont le cumul en nombre de codons atteignait Le choix de cette approche, plutôt que de ne considérer que les gènes de protéines ribosomales a été guidé par deux considérations. Tout d abord nous voulions pouvoir établir nos tables sur un nombre de codons suffisamment élevé pour éviter des biais liés à des variations stochastiques, et donc il était nécessaire de considérer un choix plus étendu de gènes. Ensuite, il est possible que la composition en codons des gènes de protéines ribosomales soit biaisée non seulement du fait de leur forte expressivité, mais aussi du fait d une composition en acides aminés particulière des protéines. L intégration dans nos jeux de données de gènes codant pour d autres protéines permettant de rééquilibrer ce biais. La Figure II.8 montre les résultats d AFC réalisées sur neuf génomes bactériens. On constate que le facteur séparant les protéines ribosomales des autres est variable en fonction de l espèce considérée. Qui plus est, cette séparation est parfois le fait non d un seul, mais de la combinaison de plusieurs facteurs. La détection du ou des facteur(s) impliqué(s) nécessite donc à chaque fois une inspection visuelle des plans formés par les premiers axes de l analyse. Ceci invalide certaines approches visant à déterminer de façon automatique les valeurs de w j par extraction des gènes possédant un score extrême sur le premier facteur. j =1-43 -
48 CHAPITRE II F2 F2 F2 F1 F1 F1 Bb Bs Cj F2 F2 F3 F1 F2 F1 Ec Hi Hp F2 F3 F2 F1 F2 F1 Mg Mp Mt Figure II.8. Plans factoriels de neuf AFC réalisées sur des génomes bactériens complets. Sur chaque plan, les facteurs utilisés comprennent celui (ou ceux) permettant de séparer les gènes de protéines ribosomales (l) des autres gènes (+). Une fois la table de w j construite, les valeurs du CAI pour tous les gènes protéiques d une espèce donnée sont calculées. Elles sont ensuite introduites dans les annotations au niveau d un descripteur spécifique, de nom /CAI INFORMATIONS COMPLÉMENTAIRES Parmi les autres informations ajoutées aux annotations de EMGLib figure, uniquement dans le cas des bactéries, l indication de l orientation des gènes protéiques sur le chromosome. Les gènes peuvent en effet se trouver soit sur le brin direct, synthétisé en continu par la DNA polymérase, soit sur le brin retardé, synthétisé sous la forme des
49 DÉVELOPPEMENTS INFORMATIQUES fragments d Okazaki (Okazaki et al., 1968). Cette orientation est ajoutée au niveau d un descripteur spécifique : /strand. Pour introduire cette information, il est nécessaire de connaître la position de l origine (OriC) et du terminus (TerC) de réplication du chromosome. Dans le cas de B. subtilis (Ogasawara et al., 1984) et d E. coli (Marsh et Worcel, 1977), cette localisation a été déterminée expérimentalement. Pour M. tuberculosis, la position de l origine de réplication a été inférée à partir de celle de Mycoplasma smegmatis qui présentait une forte similarité structurelle avec cette bactérie (Salazar et al., 1996). Pour H. influenzae, elle a été prédite par Fleischmann et al. (1995) sur la base de la présence de sites de fixation de la protéine DnaA et de l orientation des opérons de rrna. Pour tous les autres génomes bactériens, nous avons utilisé le programme Oriloc (Frank et Lobry, 2000) permettant de prédire la position de OriC et de TerC. Ce programme constitue une implémentation de la méthode de Lobry (1996a, 1996b) basée sur l existence d asymétries compositionnelles entre les deux brins du chromosome. Les informations figurant dans les annotations originales sont fréquemment corrigées ou complétées. Pour certains gènes, une référence croisée à la séquence protéique correspondante dans SWISS-PROT est introduite dans le descripteur /db_xref. Dans le cas des gènes de B. subtilis, le contenu du descripteur /product a été systématiquement modifié (lorsque cela était nécessaire) en utilisant les données de SWISS-PROT. Quand la protéine codée par le gène est une enzyme, le numéro EC (Enzyme Comission), provenant de la base de données ENZYME est ajouté sous le descripteur /EC_number. Enfin, si la référence croisée avec une séquence SWISS- PROT est disponible, le numéro de la famille d HOBACGEN à laquelle appartient le gène est introduit au niveau d un descripteur /gene_family. Dans le cas d E. coli, les données du transcriptome collectées par Thieffry et al. (1998) sont introduites. Elles comprennent les limites de tous les opérons, les localisations des sites de démarrage de la transcription, les scores des régions -35 et -10, ainsi que la séquence des promoteurs. Les limites d un opéron sont fixées en 5 par la position de la première base du codon d initiation du premier gène, et en 3 par la position de la dernière base du codon de terminaison du dernier gène. Figurent également des indications quant à la façon dont ces données ont été obtenues (évidence expérimentale ou détermination par analyse informatique) ACCÈS AUX DONNÉES La façon la plus simple d accéder à EMGLib se fait par l intermédiaire de trois serveurs Web couvrant des aspects complémentaires. Le premier de ces serveurs est celui du PBIL. Toutes ses fonctionnalités précédemment décrites peuvent être utilisées avec EMGLib. De plus, une page spécifique 5 permet d accéder à des informations sur l historique de la banque, les dernières améliorations qui y ont été apportées et les modifications effectuées au niveau des annotations. Cette page donne également accès à une liste de pointeurs sur des sites en rapport avec le séquençage des génomes bactériens. Le second serveur est celui de Micado 6, outre les séquences de la banque, ce système permet d accéder aux données sur l analyse fonctionnelle et la cartographie du génome de B. subtilis (Biaudet et al., 1996, 1997). L implémentation d EMGLib sous Micado a été réalisée par le groupe de Ph. Bessières. Pour gérer les données
50 CHAPITRE II d EMGLib, ce système utilise, côté serveur, un SGBD relationnel. Pour permettre à l utilisateur d accéder aux données, Micado est doté de deux interfaces de consultation et d interrogation : l une écrite en langage Perl et l autre en Java. Parmi les outils spécifiques apportés par la version Java figurent un système multifenêtres d exploration et de comparaison de cartes réalisées à différentes échelles (génétique et physique). Ce dispositif permet la synchronisation du déroulement entre les fenêtres représentant les données cartographiques des génomes à différentes échelles (Figure II.9). Par ailleurs, le client Java communique avec le SGBD par l intermédiaire d un protocole CORBA. L utilisation de ce nouveau standard devant permettre de nombreuses extensions, en particulier au niveau de l interconnexion avec d autres bases de données. SeqView 0.09a 20/02/2000 File Edit Help Start: Stop: BP/Line: Redraw Reset yxam yxoi yxna yxoc yxol yxok yxoj yxoh yxog yxof yxod yxob gntr gntk gntp gntz ahpc Sequence window yxoa ACATGGACAT TGCTGGAAAC CATTATTTCC GTTGCTGGAC TGGGATTCAT TTTATTGTTA AGTTTAGTTG TATGAAATTA AGAAGGAGCT GTAACACATG TTCAATTCGA TTGGTGTCAT AGGCTTAGGC GTAATGGGAA GCAACATCGC CTTAAACATG GCAAATAAAG GCGAAAACGT CGCTGTCTAT AATTACACCA GAGATTTAAC GGACCAGCTT ATCCAAAAGC TGGATGGACA ATCTCTCAGC CCGTATTACG AGCTTGAGGA CTTTGTTCAA TCGTTAGAAA AACCAAGAAA AATCTTTTTG ATGGTCACAG CGGGAAAACC CGTAGATTCC GTCATCCAAT CATTAAAGCC fbp TTTGCTTGAA GAAGGCGACG TCATCATGGA CGGAGGAAAC TCCCACTATG AAGACACAGA AAGAAGATAT GACGAGCTGA AGGAAAAAGG GATCGGCTAC CTGGGAGTCG GCATTTCCGG CGGTGAAGTC GGTGCGTTAA CAGGGCCTTC CATCATGCCG GGCGGAGATC GCGACGTCTA TGAGAAAGCC GCTCCTATCC TGACGAAAAT CGCAGCCCAA GTCGGAGATG ACCCTTGCTG TGTCTATATC GGACCAAAAG GGGCAGGGCA CTTTACAAAA ATGGTGCACA ACGGCATTGA ATATGCCGAC ATGCAGCTGA TTGCAGAAGC TTATACGTTT CTAAGAGAAA CGCTTCGTCT GCCGCTAGAT Type: GAAATTGCAT CG0031 CTATTTTTGA AACATGGAAT Start: CAAGGTGAGC 0 TGAAAAGCTA Stop: TTTAATAGAG ATTACAGCTG AGATTTTACG CAAAAAAGAC GAAAAAACAG GACAGCCTCT Mouse on: GATCGATGTC ATCCTTGATA AAACCGGCCA AAAAGGCACC GGAAAATGGA CGAGCATGCA GGCGATTGAT AACGGCATCC CGTCCACGAT CATCACAGAG TCCTTGTTCG CCCGCTACTT GTCATCTTTA AAAGAAGAAC GGATGGCAGC TCAAGATGTA DNA TTAGCAGGCC sequence written CGGAAGCCGA Quit Qualifier window AGAAAAACAC TTGGATAAAG ACACTTGGAT TGAATACGTC AGACAGGCTC TTTACATGGG GAAAGTATGC GCCTATGCAC AAGGCTTTGC CCAATATAAG ATGTCATCTG AGCTTTACGG CTGGAATCTG CCGCTCAAAG ACATCGCCTT GATTTTCCGA GGCGGCTGCA TCATCCGCGC /CAI= TGATTTCCTA AACGTGATCA GCGAAGCATT CAGTGAGCAG CCAAATCTGG CTAACCTGCT /EC_number= GATCGCGCCT TATTTCACAG ATAAGCTCCA TGCCTATCAA ACAGGCCTGC GAAAAGTCGT /db_xref=swiss-prot:p12013 TTGCGAGGGT ATCAGCACCG GAATCTCTTT CCCATGCTTA ACTACCGCGC TCTCTTATTA /gene=gntz CGACGGCTAC CGCACAGGGC GTTCCAATGC GAACCTCTTG CAGGCACAGC GCGATTACTT /gene_family=hbg TGGCGCTCAC ACGTACGAAC GGACTGATAT GGACGGCGTC TTCCATACGA ATTGGTCTGA /map=351.6 /product=6-phosphogluconate dehydrogenase /strand=lagging Dismiss Dismiss Figure II.9. Visualisation des données d EMGLib au moyen de l interface Java de Micado. Sur cet exemple figure une partie de la carte physique du génome de B. subtilis avec la séquence nucléotidique correspondante, les deux fenêtres ayant un déroulement synchronisé. Le serveur SRS d INFOBIOGEN permet, quant à lui, d accéder uniquement aux séquences des gènes figurant dans EMGLib. La version de la banque disponible sur ce serveur contient des modifications supplémentaires au niveau des annotations des gènes protéiques. Figurent pour les protéines correspondant à la traduction de ces gènes les valeurs du pi, de la masse moléculaire, de l hydrophobicité moyenne et maximale, ainsi que l indication de la présence de segments transmembranaires
51 DÉVELOPPEMENTS INFORMATIQUES Pour les utilisateurs désireux d installer une version d EMGLib sur leur machine, il est possible de télécharger la banque à partir des serveurs FTP du PBIL 7 ou d INFOBIOGEN 8. Ces sites permettent de récupérer le fichier plat au format GenBank contenant toutes les séquences, les index ACNUC ainsi que les différentes versions exécutables du logiciel Query. Du fait que cette banque possède encore une taille raisonnable (300 Mo), il est possible de l installer sur un poste de type microordinateur PERSPECTIVES Un gros effort reste à faire au niveau de l amélioration des annotations des séquences figurant dans EMGLib. En particulier, il serait nécessaire de corriger les noms et les fonctions associés à de nombreux gènes. En effet, lorsqu un gène nouvellement séquencé est fortement similaire à un gène déjà caractérisé dans une autre espèce, son nom lui est attribué. Mais il est fréquent qu à la lumière de l obtention des génomes complets, on constate que de fausses assignations ont été effectuées. Pour ce faire, il serait possible d utiliser les données présentes dans les banques HOBACGEN et COLIPAGE. En effet, ces deux banques procurent des informations complémentaires sur les homologies existant entre les gènes protéiques bactériens. Alors que HOBACGEN contient des données obtenues sur des séquences entières, COLIPAGE compile des informations sur l organisation des protéines en modules paralogues (Riley et Labedan, 1997 ; Labedan et Riley, 1999). À plus long terme, du fait que le système Micado est susceptible de gérer des sources de données très différentes grâce à ses capacités d extensions liées à l utilisation de CORBA, nous pourrons envisager d intégrer dans EMGLib d autres aspects de la biologie moléculaire des bactéries. Nous avons ainsi prévu d inclure une classification métabolique des gènes ainsi que des données de l analyse fonctionnelle pour une espèce bien étudiée comme B. subtilis. 3. HOBACGEN HOBACGEN a tout d abord été conçue dans la continuité d HOVERGEN. Cependant, du fait des concepts nouveaux qui ont été introduits dans ce système, en particulier au niveau de la construction des familles et de son organisation de type client/serveur, HOBACGEN est devenue le prototype de ce que pourrait être une banque de données généraliste dédiée aux gènes homologues dans toutes les espèces. Ce système a été conçu avec Laurent Duret, chercheur CNRS au Laboratoire de Biométrie et Biologie Évolutive. La construction d HOBACGEN a constitué l essentiel de mon activité de développement informatique au cours de la période MOTIVATION La génomique comparative est une approche couramment utilisée dans l analyse des séquences. Son application la plus commune est la confirmation de la présence de régions codantes. Après prédiction de la présence de parties codantes au moyen de méthodes spécifiques, il est d usage de rechercher dans les banques s il existe des 7 ftp://pbil.univ-lyon1.fr/pub/emglib/ 8 ftp://ftp.infobiogen.fr/pub/db/acnuc/emglib/
52 CHAPITRE II séquences protéiques homologues à la traduction de ces gènes putatifs. Si tel est le cas, on a une confirmation de l existence de ces parties codantes. Une autre utilisation est l analyse fonctionnelle. Par homologie avec d autres séquences on peut détecter les régions fonctionnelles d une protéine ou bien prédire sa fonction. L étude des contraintes structurales peut également être abordée de cette façon. Il existe ainsi des méthodes utilisant l information produite par l alignement d un ensemble de protéines pour affiner les prédictions de structures secondaires (Cuff et Barton, 1999). De nombreuses études liées à la phylogénie moléculaire sont également tributaires de la génomique comparative. Ainsi, il est possible de se servir d approches de ce type pour rechercher la présence de transferts horizontaux entre certaines espèces bactériennes (Nelson et al., 1999) ; pour déterminer quelles sont les voies métaboliques communes ou spécifiques à certains taxons ; ou pour estimer le contenu du génome ancestral (Mushegian et Koonin, 1996). Le problème de la recherche des homologues, étape obligatoire de toute étude de génomique comparative, est qu il s agit d un processus complexe qui requiert d effectuer une série de traitements. Ainsi, il est tout d abord nécessaire de déterminer des similarités entre séquences, puis de calculer des alignements multiples et de construire des arbres phylogénétiques en utilisant les dits alignements ; tout ceci nécessitant l emploi de programmes dédiés, souvent complexes à mettre en œuvre. Dans le courant de ce processus, il faut également pouvoir disposer de données taxonomiques de référence sur les espèces étudiées, ceci afin de comparer la cohérence entre les arbres construits à partir des familles de gènes et cette taxonomie. Enfin, il est nécessaire de pouvoir accéder aux annotations des séquences figurant dans les banques. C est dans ce contexte que les banques de gènes homologues HOVERGEN et HOBACGEN ont été développées. De tels systèmes intègrent en effet toutes les données précédemment décrites : séquences alignements et arbres. Ceci ayant été rendu possible par la définition de procédures automatiques qui sont exposées infra CONSTRUCTION DE LA BANQUE La version 6 d HOBACGEN (décembre 1999) a été construite à partir de toutes les séquences protéiques de bactéries, d archées et de levure disponibles dans SWISS- PROT 38 et TrEMBL 12. Sur un total de protéines utilisées pour construire cette version de la banque, provenaient de bactéries, d archées, et de levure. Le choix de SWISS-PROT pour construire la banque s est fait pour plusieurs raisons. Tout d abord, l ensemble constitué par SWISS-PROT et TrEMBL est quasiment non-redondant et exhaustif. L absence de redondances évite donc d avoir à gérer ce problème, comme c est le cas avec HOVERGEN, construite à partir de GenBank. Ensuite les annotations figurant dans SWISS-PROT sont connues comme étant d une qualité supérieure à ce qui peut se trouver dans les autres systèmes généralistes. Enfin, l intégration systématique de références croisées avec les banques de séquences nucléotidiques permet d accéder à cette information. L essentiel des informations apportées par HOBACGEN se trouve dans la classification des gènes protéiques en familles. Pour construire ces familles toutes les séquences sont comparées les unes avec les autres au moyen du programme de recherche de similarités BLASTP2 (Altschul et al., 1997). Ce programme possède une plus grande sensibilité que son prédécesseur BLASTP (Altschul et al., 1990), de plus, il permet l introduction de gaps et donc la production de véritables alignements. Les recherches de similarités sont effectuées sur des séquences filtrées au préalable par le
53 DÉVELOPPEMENTS INFORMATIQUES programme SEG qui permet de se débarrasser des régions de faible complexité (Wootton et Fedheren, 1996). La matrice utilisée pour les mesures de similarité entre acides aminés est BLOSUM62 (Henikoff et Henikoff, 1992), et le seuil pour la valeur du paramètre E est fixé à Seq. A S3 S1 S2 S4 Seq. B Seq. A S1 S2 Seq. B lg1 lghsp1 lg2 lghsp2 lg3 Figure II.10. Élimination des segments incompatibles dans les sorties de BLASTP2. Pour chaque couple de séquences homologues, les segments non compatibles avec un alignement global sont retirés. Dans cet exemple, les segments S1 et S2 sont compatibles, mais pas les segments S3 et S4. Une fois que BLASTP2 a fini de tourner, il est nécessaire de filtrer les résultats retournés. En effet, ce programme effectue des alignements locaux (par blocs) et il est nécessaire d éliminer les segments homologues qui sont incompatibles avec un alignement global, ceci pour chaque couple de séquences (Figure II.10). Pour les séquences non partielles, une fois que tous les segments incompatibles ont été retirés, on regarde si les régions restantes recouvrent au moins 80 % de la longueur des séquences alignées. Si cette condition est remplie, et si la similarité entre les deux séquences est au moins égale à 50 %, alors elles sont incluses dans une même famille. Une relation d inclusion, de type simple lien (cf. I.2.1.3), est utilisée pour construire progressivement les familles. Une séquence partielle peut être incluse dans une famille préexistante si elle remplit les conditions requises pour les séquences non partielles, et si sa longueur est supérieure ou égale à 100 acides aminés, ou si elle est supérieure à 50 % de la longueur des autres séquences. L inclusion transitive simple ne s applique pas pour les séquences partielles mais celles-ci peuvent être rattachées à plus d une famille. Les séquences partielles non classées sont regroupées dans une famille particulière, portant le numéro FAM Toutes les familles reçoivent un numéro, qui peut ne pas être conservé d une version de la banque à l autre. Puis les noms donnés aux familles sont créés à l aide d une procédure semi-automatique utilisant le contenu des champs DE et SIMILARITY des annotations de SWISS-PROT/TrEMBL (Figure II.11). Le programme construit tout d abord une table non-redondante obtenue par la fusion du champ DE des séquences appartenant à une famille donnée. Si la longueur de cette table est inférieure à un certain seuil, elle est utilisée pour construire le nom de la famille. Dans le cas contraire, le programme crée alors une seconde table obtenue par fusion des champs SIMILARITY. De nouveau, si la longueur de cette table est inférieure à un seuil, elle est utilisée pour
54 CHAPITRE II construire le nom de la famille. Si elle est également trop longue, une expertise manuelle est alors nécessaire pour fixer le nom. Annotations dans SPTREMBL des séquences d une famille Extraction des champs DE Extraction des champs SIMILARITY Table non redondante de tous les champs DE lg seuil lg < seuil Concaténation de la table Table non redondante de tous les champs SIMILARITY lg < seuil Concaténation de la table Nom de la famille lg seuil Expertise manuelle Figure II.11. Procédure d assignation du nom des familles d HOBACGEN. Une fois les numéros et les noms des familles fixés, les fichiers originaux contenant les séquences de SWISS-PROT/TrEMBL et EMBL sont modifiés. Pour les entrées EMBL la seule modification est l ajout d un descripteur /gene_family au niveau des annotations des CDS. Dans le cas des entrées SWISS-PROT/TrEMBL, ce même descripteur est introduit au niveau du champ CC. De plus, des informations sur la structure modulaire des protéines, prises à partir de la banque ProDom sont introduites. Parmi ces informations figurent les numéros d accession des modules considérés ainsi que leur position dans la séquence. Une fois ceci fait, deux banques au format ACNUC sont créées : une contenant les séquences protéiques, et une contenant les séquences nucléotidiques. Pour chaque famille un alignement multiple et un arbre phylogénétique sont calculés. Les alignements multiples sont déterminés avec CLUSTAL W, le programme étant utilisé avec tous ses paramètres par défaut, excepté le fait que l option permettant de calculer rapidement (mais de façon plus approximative) les alignements par paires est préférée. Sur chaque alignement on fait ensuite tourner un programme de construction d arbres phylogénétiques. Si la matrice de distances utilisée est complète, la méthode utilisée est BIONJ, une version améliorée de l algorithme NJ (Neighbor Joining) (Gascuel, 1997). Si l on a une matrice de distances incomplète, on utilise une méthode dérivée de celle proposée par Lapointe et Kirsch (1995) permettant d estimer les distances manquantes (A. Guénoche, non publié). Des matrices incomplètes sont obtenues lorsque des séquences partielles incluses dans une familles ne présentent qu une faible zone de recouvrement voire pas de recouvrement du tout entre elles. Les arbres obtenus sont racinés par la méthode du mid-point qui fait que les moyennes des longueurs de branches de part et d autre de la racine sont égales. Les alignements figurant dans HOBACGEN ne sont pas corrigés manuellement et les arbres sont construits en utilisant simplement le pourcentage de divergence entre les séquences protéiques. De ce fait, bien que ces arbres soient efficaces pour détecter des paralogues, il ne correspondent pas à de véritables arbres phylogénétiques, particulièrement quand ils comportent des branches internes courtes ou quand les vitesses d évolution diffèrent de façon importante entre les espèces
55 DÉVELOPPEMENTS INFORMATIQUES Protéines Familles Fréquence ,7 % ,9 % ,5 % ,1 % ,8 % ,6 % ,4 % % Table II.3. Distribution des familles de taille supérieure à un. À partir des protéines extraites de SWISS-PROT/TrEMBL, un total de (65 %) ont été classées en familles contenant au moins deux séquences, protéines sont uniques dans leur famille (29 %), et il y a séquences partielles non rattachées à une famille (5 %). La distribution des familles en fonction du nombre de séquences est représentée sur la Table II.3, tandis que les dix plus grandes familles sont reportées sur la Table II.4. Famille Nombre Transporteurs ABC 874 Flagellines bactériennes 559 Régulateurs de transcription NifH/FrxC 439 Protéines membranaires OmpC/OmpK 390 Régulateurs de transcription LuxR/UhpA 338 Protéines porines 338 Sous-unités σ 70 de la RNA polymérase 314 Topoisomérases de type II 305 Déhydrogénases/réductases à chaîne courte 293 Lipoprotéines membranaires majeures Table II.4. Les dix plus grandes familles d HOBACGEN. Par ailleurs, on constate que 190 familles sont communes aux bactéries, archées et à la levure. ces familles contiennent principalement des gènes codant pour des protéines impliquées dans la traduction (e.g., des protéines ribosomales et des aminoacyl-trna synthétases), dans la biosynthèse des nucléotides et des acides aminés et la glycolyse ACCÈS AUX DONNÉES Le principe choisi pour accéder à la banque est celui d un système client/serveur comme dans le cas d Entrez et d ACeDB ; une telle organisation supprimant les problèmes d installation et de mises à jour pour les utilisateurs. Ceci est un point critique si l on considère la vitesse à laquelle croissent les banques et l espace disque qui est requis pour leur utilisation. Cependant, l utilisation d une architecture de ce type ne présente pas que des avantages, et le problème principal est bien sûr celui de l encombrement du réseau. La solution à ce problème consiste en l installation de miroirs, les utilisateurs se connectant sur le serveur le plus proche géographiquement
56 CHAPITRE II C est ainsi qu il est possible de télécharger aussi bien le client seul que la banque complète à partir de la page dédiée à HOBACGEN sur le serveur du PBIL CLIENT JAVA Le client de nom HobacFetch est une application Java, ce qui autorise sa portabilité sur toute plate-forme pour laquelle une machine virtuelle est disponible, c est-à-dire la quasi-totalité des architectures courantes. Ce programme a été testé sur différentes machines Unix, sur Macintosh (avec toutes les versions de MacOS allant de 8.0 à 9.0) ainsi que sur Windows (95, 98 et NT 4.0). HobacFetch a été développé comme une application et non une applet ceci pour éviter les problèmes liés à ce genre de programmes : incompatibilités entre la version de Java utilisée pour le développement et celle disponible sur le navigateur Web de l utilisateur, nécessité de développer des applets «certifiées» afin d autoriser les accès disque sur la machine cliente, lenteur du chargement, ceci même dans le cas d un accès réseau rapide. File View Select Help Total number of families: Families HBG ORF; THIAMINE BIOSYNTHESIS LIPOPROTEIN APBE HBG THIAMINE BIOSYNTHESIS LIPOPROTEIN APBE; APB HBG CLASS B BACTERIAL ACID PHOSPHATASES; CLASS HBG ADENINE PHOSPHORIBOSYLTRANSFERASE 1; ADENIN HBG N-ACETYL-GAMMA-GLUTAMYL-PHOSPHATASE; N-ACET HBG ARGR / AHRC FAMILY; ARGININE HYDROXIMATE RE HBG AROMATIC AMINO ACID AMINOTRANSFERASE II; AR HBG EPSP SYNTHASE FAMILY HBG DEHYDROQUINATE SYNTHASE; DEHYDROQUINATE S HBG CHORISMATE SYNTHASE FAMILY; CHORISMATE SYNT HBG SHIKIMATE KINASE II HBG ASLB/ATSB FAMILY; ARYLSULFATASE REGULATORY HBG ACID SHOCK PROTEIN; HEAT SHOCK PROTEIN HSP. HBG FUMARATE HYDRATASE, MITOCHONDRIAL HBG ACID SHOCK PROTEIN; ASR_ECOLI HBG SUCCINYLARGININE DIHYDROLASE, ASTB_PSEAE Figure II.12. Fenêtre principale de l interface HobacFetch. À partir de cette fenêtre l utilisateur peut composer des requêtes lui permettant de n afficher que les familles correspondant à certains critères. Il peut également sélectionner une famille particulière afin de visualiser l arbre phylogénétique qui lui est associé ou éditer les préférences du système. Une particularité de ce client est qu il n est pas limité à HOBACGEN. Toute banque intégrant des alignements et des arbres phylogénétiques peut être interrogée avec HobacFetch, ceci à condition qu elle utilise le format de stockage des données d HOBACGEN. C est ainsi qu il est possible d interroger une version de la banque ProDom-CG au moyen de cette interface. De même, la banque HOFLYGEN, dédiée aux gènes homologues détectés entre D. melanogaster et l ensemble des autres organismes possédant des séquences dans les banques, peut être consultée au moyen de cette interface. Comme tous les logiciels récents, HobacFetch intègre une interface graphique permettant de consulter et d exploiter facilement les données. À partir de la fenêtre principale de l interface (Figure II.12) l utilisateur doit tout d abord charger une liste de famille ceci, soit directement depuis le serveur, soit au moyen d un sous-ensemble
57 DÉVELOPPEMENTS INFORMATIQUES préalablement défini puis sauvegardé dans un fichier. Une fois cette opération effectuée, il est possible d effectuer des requêtes sur la liste chargée afin de ne récupérer que les familles répondant à certains critères. Par exemple, il est possible de sélectionner des familles contenant au moins un certain nombre de séquences ou de taxons. De même une possibilité de recherche par mot clé est disponible. L utilisateur peut également sélectionner des séquences de SWISS-PROT/TrEMBL au moyen de n importe quel système d interrogation puis, en utilisant leurs mnémoniques ou leurs numéros d accession, récupérer les familles contenant ces protéines dans HOBACGEN. Enfin, il est possible d utiliser la fonction de recherche par croisement des taxons, décrite en II.1.2.3, et qui permet de récupérer un ensemble de familles contenant au moins une séquence provenant des taxons rentrés par l utilisateur. Les préférences générales du système sont également accessibles à partir de cette fenêtre. Parmi ces préférences figure tout d abord le choix du serveur qui sera interrogé au moyen de l interface ; une liste des serveurs publics étant disponible sur le site du PBIL. Ensuite figure le choix de la banque à interroger. Enfin, il est possible d éditer les différents couples taxon/couleur proposés à l utilisateur. Celui-ci pouvant modifier la couleur associée à un taxon ou bien le nom du taxon associé à une couleur. Family: HBG Tree APT2_YEAST APT1_YEAST APT_HELPY APT_MYCPN APT_MYCGE APT_STRCO O APT_MYCTU APT_PSEST APT_PSEAE APT_BORBU O31060 APT_HAEIN APT_ECOLI P73935 O34443 O32418 O84001 Select Subtree Outgroup Swap nodes Partial Length Use Leaf Up Colors Reset Close Help Figure II.13. Affichage de l arbre phylogénétique de la famille HBG Un code couleur permet de distinguer les différentes espèces et d identifier rapidement la présence de paralogies. Dans cet exemple, on constate que deux paralogues sont présents chez la levure (APT1_YEAST et APT2_YEAST). Après la sélection d une famille, l arbre phylogénétique correspondant est affiché (Figure II.13) dans une fenêtre spécifique. Dans cet arbre, les séquences sont colorées en utilisant un code relatif à la position taxonomique de l espèce dans laquelle elles ont été obtenues. Grâce à ce code, il est très facile d identifier les paralogues à l intérieur d une famille. L arbre affiché est actif, avec des options de re-racinement, d échange de nœuds ou de sélection de sous-arbres. Cette dernière option est particulièrement importante puisque, comme on l a vu précédemment, certaines familles peuvent contenir plusieurs centaines de gènes. Par ailleurs l utilisateur a la possibilité d afficher à l écran les longueurs de branches ou de masquer les séquences partielles courtes. Au
58 CHAPITRE II niveau de l affichage, une séquence sera déclarée comme partielle si sa longueur est inférieure à 85 % de la longueur moyenne des séquences non partielles de la famille. Par un simple clic sur une ou plusieurs feuille(s) de l arbre, on peut accéder soit aux entrées SWISS-PROT/TrEMBL ou EMBL, soit à l alignement des séquences sélectionnées (Figure II.14). Pour les séquences nucléotidiques, du fait de la redondance importante existant dans EMBL, on trouvera fréquemment plusieurs CDS associés à une entrée SWISS-PROT/TrEMBL. Dans ce cas, l utilisateur peut choisir, parmi les différents CDS référencés, celui qu il désire visualiser. L alignement des séquences sélectionnées n est pas calculé mais simplement reconstruit à partir de l alignement complet de la famille. Alignement Total div.: 95.9% Div. w/o gaps: 46.3% Gaps: 49.6% APT1_YEAST -MSIAS-YAQELKLAHQYPNFPSEGILFEDFLPIFRNFGLFQKLIDAFKLHLEEAF APT2_YEAST -MSISESYAKEIKTARQFTDFPIEGEQFEDFLPIIGNPTLFQKLVHTFKTHLEEKF APT_BORBU ----MKNKTEYYDQFSKIPNFPKKGVLFYDITSVLLKPEVYSSLINEVYSFYNF-- APT_ECOLI -MTATAQQLEYLKNSKSIQDYPKPGILFRDVTSLLEDPKAYALSIDLLVERYKN-- APT_HAEIN -MT-T--QLDLIKSSKSIPNYPKEGIIFRDITTLLEVPAAFKATIDLIVEQYRD-- APT_HELPY -MNET--LKEELLQSREVKDYPKKGILFKDITTLLNYPKLFNKLIDTLKKRYLA-- APT_MYCGE ----MDQNFKLLDQAKRFENFPNQGTLFYDITPVFSNPQLFNFVLTQMAQFIKA-- APT_MYCPN MAQFIQE-- APT_MYCTU RRSSAISVADVVASLRDVADFPVPGVEFKDLTPLFADRRGLAAVTEALADRAS--- APT_PSEAE ----MIFDEFTLKSQRAVPDFPKPGVVFRDITPLFQSPRALRMTVDSFVQRYIE-- APT_PSEST ----MIFDEFSIKTLRPVQDFPRPGVVFRDITPLFQSPKALRMVADSLIQRYVE-- APT_STRCO -MTEPTGITELLLSRRDVADYPEPGVVFKDITPLLADPGAFAALTDALAEAAGR-- O MKKVEDYRTIPDFPEPGIMFRDVTSILQDAEGFKLAIDEMIKLLDG-- O MDLKQYSEVQDWPKPGVSFKDITTIMDNGEAYGYATDKIVEYAKD-- O MDLKQYTIVPDYPKEGVQFKDITTLMDKGDVYRYATDQIVEYAKE-- O84001 RYHAPVDGHAALDRARKRIDFPKKGILYYDITGVLMNAAVFRYCLDQMVEFYRD-- O87330 ALSTFDRAREALDKKRYVQDFPEKGVLFEDLTPVLGDAESFVAVVDAMAEAAEK-- P MDLKALRDIPDFPKPGIMFRDITTLLNSPEGLRYTIDSLVEQCES-- Close Help Figure II.14. Alignement d un ensemble de séquences sélectionnées dans la famille HBG Des fonctions permettent à l utilisateur de sauvegarder dans des fichiers texte la liste des familles sélectionnées, les séquences, les alignements ou les arbres. Les formats utilisés sont ceux de SWISS-PROT et EMBL pour les séquences, de CLUSTAL pour les alignements, et de PHYLIP (Felsenstein, 1989) pour les arbres. Tous ces formats sont des standards, reconnus par la plupart des logiciels d analyse des séquences SERVEUR La partie serveur comprend trois composants bien distincts : un service Web, un programme d accès aux données, et la banque proprement dite. La présence d un «démon» assurant en permanence un service Web est nécessaire car toutes les transactions entre le client et le serveur se font au moyen de requêtes HTTP, les requêtes envoyées par un client étant interceptées par ce programme. Qui plus est, le démon Web en question doit être capable de manipuler des scripts CGI puisque c est de cette façon que le programme accédant aux données fonctionne. Bien qu il existe des logiciels de service Web pour la plupart des plates-formes, il est recommandé de n installer ce genre de système que sur des stations de travail Unix ou Windows NT. Le second composant du serveur est le programme Query_http, lancé par le serveur Web comme un script CGI à chaque fois qu une requête est transmise depuis un
59 DÉVELOPPEMENTS INFORMATIQUES client. Ce programme est écrit en C en utilisant les bibliothèques d ACNUC, il est capable de lire les données d HOBACGEN (séquences, alignements et arbres) et de les transmettre au démon Web, qui a son tour les enverra au client (Figure II.15). HobacFetch (Unix, Mac, Windows) Requêtes HTTP Serveur Web + Query_http (Unix) Arbres T-RRDLNHS TVRRDFQYI TVRRDIRKL TIRRDL-KL TIRRDI--L TIRRDLIN- Alignements SWISS- PROT EMBL Figure II.15. Organisation générale de l architecture client/serveur d HOBACGEN. La banque HOBACGEN poprement dite est constituée par l association de deux fichiers texte, l un contenant les séquences de SWISS-PROT/TrEMBL et l autre les séquences d EMBL. À ces fichiers sont associés les deux ensembles correspondants d index ACNUC, un fichier de description des familles et les fichiers contenant les alignements et les arbres. Du fait de la présence des fichiers texte et des index ACNUC, il est donc possible de consulter la partie protéique ou la partie nucléique d HOBACGEN au moyen du logiciel Query ACCÈS PAR LE WEB Tout comme pour les autres banques développées dans notre groupe, il est possible d accéder à HOBACGEN par le biais du service d interrogation des banques figurant sur le serveur Web du PBIL. Une fonctionnalité supplémentaire est offerte pour HOBACGEN et HOVERGEN par rapport aux autres banques : la possibilité, lors de la visualisation d un gène protéique, d accéder à tous les gènes appartenant à la même famille ainsi qu à l alignement et à l arbre de cette famille. L accès aux alignements et aux arbres est réalisé par le truchement de liens hypertexte qui vont déclencher le lancement d applications externes permettant leur visualisation. C est ainsi que les programmes SEAVIEW et NJplot peuvent être utilisés en association avec ces banques lors de leur consultation sur le serveur COMPARAISON AVEC D AUTRES SYSTÈMES Nous avons vu dans le premier chapitre que d autres systèmes qu HOBACGEN introduisaient des informations sur les relations d homologies existant entre les séquences. C est ainsi qu Entrez permet de visualiser toutes les similarités significatives détectées entre les différentes séquences de GenBank ou de GenPept. Cependant Entrez ne permet pas d accéder à des alignements multiples ou à des arbres. De leur côté, les banques ProtFam et ProtoMap contiennent également une classification des protéines en familles avec la possibilité d accéder aux alignements et aux arbres correspondants
60 CHAPITRE II La principale limitation de ProtFam et de ProtoMap est que ces systèmes se limitent aux protéines alors qu HOBACGEN permet d accéder aux séquences nucléotidiques. Un autre problème est l absence d une interface intégrée et conviviale pour accéder aux données. Dans le cas de ProtFam, comme pour la majeure partie des banques de séquences généralistes, la consultation et l interrogation se font par l intermédiaire d une interface Web. Comme nous l avons vu précédemment (cf. I.3.1), ce genre d interface présente d importantes limitations en particulier au niveau des interactions avec l utilisateur. En effet, si le serveur Web associé à ProtFam permet la visualisation des alignements multiples et des dendrogrammes associés à une famille, l affichage est complètement statique. Par exemple, il n est pas possible d exclure des séquences d un alignement multiple, ou d effectuer des manipulations simples sur l arbre (comme échanger deux nœuds, re-raciner l arbre ou sélectionner un sous-arbre). Le système ProtoMap possède une interactivité sensiblement améliorée du fait de l utilisation d applets Java, mais cette interface ne permet toujours pas de manipuler de façon souple les alignements et les arbres. Enfin, il n est pas possible d interroger ProtFam ou ProtoMap en utilisant des critères taxonomiques complexes comme cela est possible avec HOBACGEN (e.g., sélection de tous les homologues communs à un ensemble de taxons donnés). ProtFam possède l avantage de procurer un accès à la structure en domaines des protéines figurant dans la banque Pfam. De son côté, ProtoMap permet de visualiser les liens d homologie existant entre des protéines appartenant à des familles différentes, ce qui revient également à intégrer des données sur l existence de domaines. Bien que l information quant à l organisation des protéines en domaines ait été introduite dans HOBACGEN (à partir de ProDom), l interface ne permet pas, pour l instant, d exploiter ces données ÉVOLUTIONS FUTURES La banque HOBACGEN bien que désormais complètement opérationnelle est susceptible de voir sa structure évoluer de façon importante dans les prochaines années. Tout d abord son interface d interrogation et de consultation sera modifiée de façon à permettre à un utilisateur d introduire ses propres séquences dans les alignements et les arbres téléchargés depuis un serveur. Ensuite il sera nécessaire de rendre possible la manipulation des informations provenant de ProDom. En particulier nous comptons introduire la possibilité de basculer des données (alignements et arbres) obtenues sur les gènes complets aux données obtenues sur les domaines. Est également envisagée l introduction de données sur la structure des protéines, ceci en collaboration avec le groupe de G. Deléage. Dans ces données figureraient la présence de signatures PROSITE, la localisation de régions transmembranaires, prédites par la méthode d Argos et al. (1982), la mesure du degré d hydrophobicité en utilisant l indice de Kyte et Doolittle (1982), la mesure de l antigénicité au moyen de l indice de Parker et al. (1986), et la prédiction de la structure secondaire grâce à la méthode SOPMA (Self Optimized Prediction Method with Alignments) (Geourjon et Deléage, 1995)
61 RÉSULTATS BIOLOGIQUES III. RÉSULTATS BIOLOGIQUES Les différents outils informatiques présentés dans le chapitre précédent m ont servi à obtenir un certain nombre de résultats biologiques qui sont exposés ici. Trois résultats particulièrement marquants sont présentés. Tout d abord une étude effectuée au moyen de l ADC sur la prédiction de la localisation subcellulaire des protéines chez les bactéries Gram négatives. Puis une analyse sur la répartition asymétrique des gènes protéiques entre les deux brins du chromosomes chez les bactéries. La dernière étude concernant l influence des variations intragénomiques de composition en bases sur les résultats produits par les méthodes intrinsèques de prédiction de transferts horizontaux. 1. LOCALISATION SUBCELLULAIRE DES PROTÉINES Chez les bactéries Gram négatives, les protéines nouvellement synthétisées peuvent soit rester dans le cytoplasme soit être exportées. Dans le cas des protéines non sécrétées, quatre localisations subcellulaires existent : la membrane plasmique (ou membrane interne), l espace périplasmique, la paroi cellulaire et la membrane externe. Dans les banques généralistes, l information sur la localisation subcellulaire n est donnée que pour une partie des protéines. Dans le cas des bactéries Gram négatives, cette information n est donnée que pour protéines sur (soit 32 %) dans la version 38 de SWISS-PROT. En collaboration avec Jean Thioulouse, chercheur CNRS au Laboratoire de Biométrie et Biologie Évolutive, j ai décidé de construire une méthode simple de prédiction de la localisation subcellulaire pour ces bactéries en utilisant l ADC. En effet, les caractéristiques physico-chimiques fort différentes des protéines appartenant à ces classes impliquent l existence de compositions en acides aminés spécifiques. De ce fait l ADC se prêtait fort naturellement à une étude de ce type MATÉRIEL ET MÉTHODES L étude présentée dans cet ouvrage a été réalisée en utilisant la version 38 de SWISS- PROT. L intérêt d utiliser cette banque est que toutes les redondances exactes sont éliminées, ce qui fait qu aucun biais ne peut être introduit du fait de duplications. Nous n avons pas considéré la possibilité d utiliser les séquences de TrEMBL car celles-ci ne contiennent que peu d annotations. Or la constitution de notre jeu de données nécessitait que la localisation subcellulaire des protéines utilisées soit connue. De ce fait, l utilisation de TrEMBL ne nous aurait apporté que peu de séquences supplémentaires par rapport à SWISS-PROT seule. Partant des protéines de bactéries Gram négatives de SWISS-PROT 38 pour lesquelles la localisation subcellulaire était connue, nous avons tout d abord retiré les protéines hypothétiques, les séquences partielles ou de moins de 50 acides aminés, et les protéines pour lesquelles cette information était incertaine (i.e., annotée comme
62 CHAPITRE III potentielle, putative ou obtenue par similarité). Par la suite nous n avons considéré que les protéines cytoplasmiques, les protéines membranaires intégrales et les protéines périplasmiques. Nous n avons pas intégré les protéines localisées dans la paroi car celles-ci étaient en trop petit nombre. Qui plus est, nous n avons pas fait la distinction entre les protéines insérées dans la membrane plasmique et celles figurant dans la membrane externe, ceci du fait que cette distinction n était quasiment jamais donnée dans les annotations des séquences. De même les protéines simplement ancrées dans la membrane plasmique n ont pas été prises en compte du fait de la fréquente impossibilité de déterminer si la région non fixée à la membrane était localisée dans le cytoplasme ou dans l espace périplasmique. La sélection finale contenait séquences avec 850 (47 %) protéines cytoplasmiques, 665 (36,8 %) protéines membranaires intégrales, et 293 (16,2 %) protéines périplasmiques. L ensemble de ces protéines venait de 201 espèces différentes, les dix espèces les plus abondantes représentant 56,7 % du total (Table III.1). Le pourcentage de protéines membranaires intégrales obtenu était plus de trois fois supérieur à celui attendu (10 % chez E. coli suivant une estimation de Lobry et Gautier, 1994). Cette sur-représentation étant dûe au fait que cette catégorie de protéines est plus souvent documentée que les autres. Espèces Nb. % Escherichia coli ,2 Salmonella typhimurium 121 6,7 Haemophilus influenzae 107 5,9 Synechocystis sp. 70 3,9 Helicobacter pylori 53 2,9 Pseudomonas aeruginosa 45 2,5 Treponema pallidum 35 1,9 Thermus aquaticus 33 1,8 Paracoccus denitrificans 27 1,5 Klebsiella pneumoniae 26 1,4 Autres , ,0 Table III.1. Répartition par espèces de l ensemble des séquences sélectionnées. Nous avons ensuite séparé cette sélection de protéines en deux parties de taille égale : un jeu pour l analyse propremenent dite et un jeu de test, pour le calcul de la précision de la discrimination. Dans ces deux sous-ensembles, le nombre d individus présents dans les trois classes était égal (à ± 1 près). L assignation des individus à l un ou l autre de ces sous-ensembles étant effectuée par tirage au sort RÉSULTATS Du fait que le nombre de classes présentes dans le jeu de données est égal à trois, seuls deux facteurs sont extraits par l ADC. Le plan formé par le croisement de ces deux facteurs est représenté sur la Figure III.1. Sur ce plan, le premier facteur sépare les protéines membranaires intégrales d un groupe comprenant les protéines cytoplasmiques et périplasmiques tandis que le second facteur sépare les protéines périplasmiques des autres protéines. La moyenne des scores obtenus sur le premier facteur est égale à -1,060 (écart type : 0,754) pour le groupe des protéines membranaires intégrales, et à 0,577 (écart type : 0.629) pour le groupe composé des autres protéines. La valeur seuil permettant de classer une protéine dans l un ou l autre de ces deux groupes est donc égale à -0,167,
63 RÉSULTATS BIOLOGIQUES toute séquence obtenant un score inférieur étant classée comme étant une protéine membranaire intégrale. Si l on compare individuellement les trois classes, on constate que les protéines membranaires intégrales présentent la variance la plus forte (0,569 au lieu de 0,310 pour les protéines cytoplasmiques et 0,399 pour les protéines périplasmiques). La moyenne des scores obtenus sur le second facteur est égale à -1,699 (écart type : 0,937) pour les protéines périplasmiques, et à 0,268 (écart type : 0,829) pour le groupe comprenant les autres protéines. La valeur seuil permettant de les séparer est donc égale à -0,655, toute séquence obtenant un score inférieur étant classée comme étant une protéine périplasmique. Les acides aminés dont les valeurs au niveau du pouvoir discriminant sont positives sur les deux facteurs (arginine, acide glutamique et histidine) permettent de discriminer les protéines cytoplasmiques (Table III.2). Les acides aminés avec des valeurs négatives sur le premier facteur et positives sur le second (phénylalanine, leucine et isoleucine) discriminent les protéines membranaires intégrales. Enfin, les acides aminés avec des valeurs légèrement positives sur le premier facteur et négatives sur le second (asparagine, proline, glutamine, thréonine) discriminent les protéines périplasmiques. 4 F F Figure III.1. Plan factoriel formé par le croisement des deux facteurs de l ADC réalisée sur les 904 protéines constituant le jeu de données de l analyse. Chaque protéine est représentée par un point relié au centre de gravité du groupe auquel elle appartient (❶ cytoplasmiques, ❷ membranaires intégrales, ❸ périplasmiques). En projetant dans l analyse les 904 protéines du sous-ensemble de test et en utilisant les deux valeurs seuil données ci-dessus, il est possible de les assigner à l une des trois classes définies. On détermine alors la précision de la discrimination avec le pourcentage de protéines correctement classées. La discrimination sur le premier facteur entre les protéines membranaires intégrales et les autres protéines se fait ainsi avec une précision de 88,2 %. Pour ce qui est de la séparation entre les protéines
64 CHAPITRE III périplasmiques des autres protéines sur le deuxième facteur, on trouve une précision égale à 85,2 %. v j (1) v j (2) d j (1) d j (2) x ij x j u ij (1) u ij (2) Arg 0,1502 0,2423 0,1848 0, ,1302 0,2100 Leu -0,3233 0,5793-0,1270 0, ,3646 0,6534 Ser -0,3494-0,3040-0,1134-0, ,5070-0,4411 Thr -0,2155-0,0992-0,0322-0, ,1730-0,0796 Pro 0,3583-1,3684 0,0649-0, ,3512-1,3415 Ala -0,3243-0,1922-0,0791-0, ,4328-0,2565 Gly -0,1870 0,0249-0,0766-0, ,1630 0,0217 Val -0,3930 0,2492-0,0373 0, ,3791 0,2403 Lys 0,2215-1,3552 0,2367-0, ,0678-0,4146 Asn -0,1117-0,2529-0,0018-0, ,0769-0,1742 Gln -0,1558-0,3920 0,0395-0, ,0726-0,1827 His 0,3227 0,0111 0,1489 0, ,2651 0,0091 Glu 0,9552 1,5223 0,3345 0, ,2883 0,4595 Asp 0,8754 0,0362 0,2429-0, ,2152 0,0089 Tyr -0,1700 0,1507-0,0530-0, ,1449 0,1285 Cys 0,0815 0,0671 0,1789-0, ,0858 0,0706 Phe -0,1738 0,4322-0,2063 0, ,3696 0,9191 Ile -0,0813 1,0229-0,0858 0, ,1148 1,4431 Met -0,2522-0,2954-0,1236 0, ,5366-0,6284 Trp -0,2274-0,0787-0,3626-0, ,3576-0, ,2890 0,5220 Table III.2. Scores factoriels, pouvoir discriminant des acides aminés et exemple de projection dans l analyse pour la protéine BCR_ECOLI. Les notations utilisées correspondent à celles introduites dans le chapitre II. Les valeurs de v i (k ) correspondent aux scores factoriels, celles de d i (k) au pouvoir discriminant, x ij aux fréquences dans la protéine projetée, x j aux fréquences dans le jeu de données de l analyse, enfin les valeurs de u ij (k) correspondent au rapport x v j (k)x ij /x i x j. Les scores factoriels des acides aminés et un exemple de projection d une protéine du jeu de données de test dans l analyse sont donnés dans la Table III.2. La séquence utilisée dans cet exemple est BCR_ECOLI (numéro d accession P28246), une protéine intervenant dans la résistance à la bicyclomicine. Le score obtenu par projection de cette protéine sur le premier facteur est égal à -2,289 et celui sur le deuxième facteur à 0,522 ce qui permet de la classer comme une protéine membranaire. Dans ce cas, la prédiction est conforme avec la localisation subcellulaire telle qu elle est donnée dans les annotations de SWISS-PROT DISCUSSION La discrimination des protéines membranaires intégrales par l intermédiaire de la phénylalanile, la leucine et l isoleucine n est pas surprenante puisque ces acides aminés sont connus pour être hautement hydrophobes. Également, la discrimination des protéines cytoplasmiques par l arginine, l acide glutamique et l histidine s explique facilement par le fait que ces acides aminés sont chargés et/ou hydrophiles et donc ils auront tendance à se trouver plus fréquemment dans les protéines solubles. Enfin, la discrimination des protéines périplasmiques par l asparagine, la proline, la glutamine et la thréonine se comprend bien lorsque l on sait que ces acides aminés sont connus pour ralentir le repliement des protéines sur elles-mêmes, et qu un repliement lent est
65 RÉSULTATS BIOLOGIQUES une des conditions nécessaires pour que des protéines destinées à être exportées puissent franchir la membrane plasmique (Nakashima et Nishikawa, 1994). La variance plus forte observée pour les protéines membranaires intégrales sur le premier facteur de l analyse signifie que la composition en acides aminés de ces protéines est plus variable que celle des autres classes. Ceci est probablement dû au fait que ces protéines doivent tout de même posséder des régions de longueur variable non insérées dans la membrane plasmique, ces régions en question comprenant des acides aminés non hydrophobes. La bonne séparation obtenue entre les différentes classes sur les deux facteurs de l ADC fait que cette méthode peut être utilisée pour prédire la localisation subcellulaire des protéines. La meilleure méthode disponible à ce jour pour discriminer les protéines membranaires des protéines globulaires solubles est une AD réalisée sur différentes caractéristiques (telles que le maximum local d hydrophobicité). La précision de cette méthode atteint 95 % (Klein et al., 1985). La résolution de notre méthode est légèrement inférieure (88,2 %) et ceci est probablement dû au fait que que nous n utilisons que les fréquences absolues en acides aminés pour séparer nos différentes classes. D autre part, le meilleur programme précédemment publié permettant de discriminer, chez les bactéries Gram négatives, les protéines périplasmiques des autres est un système expert utilisant lui aussi des données sur la composition en acides aminés (Nakai et Kanehisa, 1991). Ce système permet d identifier de telles protéines avec une précision de 83 %, tandis que notre analyse donne des résultats comparables (85,2 %) par le biais de calculs beaucoup plus simples. Même si notre approche donne des résultats dont la précision est semblable à celle de méthodes déjà publiées auparavant, elle possède l avantage incontestable de permettre la discrimination des trois classes de protéines sur une seule analyse. En effet, les méthodes concurrentes citées précédemment permettent seulement de réaliser des bipartitions (protéines membranaires intégrales vs. protéines globulaires solubles ou protéines périplasmiques vs. tous les autres types de protéines). Ainsi, il est difficile de comparer directement les résultats obtenus avec l ADC et ceux obtenus avec ces méthodes. Un autre point est que ces analyses ont été réalisées sur des jeux de données beaucoup plus réduits que celui que nous avons utilisé. Klein et al. (1985) ont employé seulement 102 protéines et Nakai et Kanehisa (1991) 106, alors que nous en avons utilisé 904. Il n est pas du tout certain que ces deux méthodes produisent des résultats comparables avec des jeux de données actualisés. L ADC est une méthode beaucoup plus générale, en ce sens qu elle peut être utilisée pour bien d autres analyses dans lesquelles gènes ou protéines sont classés dans des groupes prédéfinis. Ainsi nous l avons utilisée afin de discriminer les parties codantes provenant de génomes bactériens en fonction de leur localisation sur l un ou l autre brin du chromosome (Perrière et al., 1996a). 2. RÉPARTITION ASYMÉTRIQUE DES GÈNES Avec Jean Lobry, enseignant-chercheur au Laboratoire de Biométrie et Biologie Évolutive, nous avons essayé de déterminer s il existait une asymétrie de répartition des gènes protéiques entre les deux brins du chromosome chez les bactéries. En effet, chez ces organismes, le chromosome est répliqué de façon bidirectionnelle après initiation au niveau de l origine de réplication. À chaque fourche de réplication est associé un complexe multi-enzymatique qui synthétise à la fois le brin direct et le brin retardé. Si un gène se trouve en cours de transcription alors que la fourche de réplication
66 CHAPITRE III approche, il est possible qu une collision entre la DNA et la RNA polymérase se produise. Du fait qu une collision codirectionnelle entre ces deux systèmes est supposée moins désavantageuse qu une collision frontale, on a émis l hypothèse qu il existe une pression de sélection apportant un avantage aux génomes pour lesquels les gènes se trouvent préférentiellement localisés sur le brin direct, ceci afin d éviter les collisions de ce type (Brewer, 1988 ; French, 1992 ; Liu et Alberts, 1995 ; Francino et Ochman, 1997). En conséquence de quoi, on s attend à ce qu il y ait une répartition asymétrique des gènes entre les brins directs et retardés, ceci avec une intensité d autant plus grande que les gènes considérés sont hautement exprimés. Chez E. coli, cette asymétrie de répartition est bien connue (Brewer, 1988 ; Liu et Alberts, 1995), et nous avons essayé de déterminer si un tel phénomène se retrouvait dans onze autres génomes bactériens complètement séquencés et pour lesquels l orientation des gènes sur le chromosome était connue MATÉRIEL ET MÉTHODES Pour effectuer cette étude, nous avons utilisé la banque EMGLib où l information sur la localisation des gènes figure dans les annotations que nous avons introduites. Les génomes considérés furent donc ceux pour lesquels la position et le terminus de réplication étaient connus ou étaient prédictibles par le programme de Frank et Lobry (2000). Ces génomes, obtenus à partir de la version 2.1 d EMGLib étaient donc ceux de : B. burgdorferi (Bb), B. subtilis (Bs), C. pneumoniae (Cp), C. trachomatis (Ct), E. coli (Ec), H. influenzae (Hi), H. pylori J99 (Hp), M. genitalium (Mg), M. pneumoniae (Mp), M. tuberculosis (Mt), R. prowazekii (Rp) et T. pallidum (Tp). Par ailleurs, EMGLib intégrant également les valeurs de CAI pour tous les gènes, nous avons utilisé cet indice afin d estimer leur expressivité. Ont été écartés de cette étude les gènes d une longueur < 150 pb, ceci afin d éviter des biais liés à des variations stochastiques dans la composition en codons pour des gènes courts. Afin de déterminer si la proportion de gènes hautement exprimés sur le brin direct est plus importante que la proportion des autres gènes sur ce même brin, nous avons classé toutes les parties codantes par valeur décroissante de CAI. Nous avons alors considéré que les gènes figurant dans les 5 % ayant les plus hautes valeurs de CAI étaient ceux hautement exprimés, les autres étant soit moyennement, soit faiblement exprimés RÉSULTATS L hypothèse nulle d une équirépartition des gènes sur les deux brins du chromosome est rejetée pour les douze espèces considérées, ceci avec un risque d erreur de première espèce égal à 5 %. Dans tous les cas on a constaté un excès de gènes sur le brin direct par rapport au brin retardé (Figure III.2). Cependant la proportion de gènes localisés sur le brin direct est très variable, puisqu elle va de 53,7 % (H. influenzae) à 79,6 % (M. genitalium). Maintenant, si l on compare la proportion de gènes hautement exprimés figurant sur le brin direct par rapport à celle des autres gènes, on constate qu elle est systématiquement plus forte dans onze espèces sur douze (Figure III.2). La seule espèce pour laquelle la proportion de gènes hautement exprimés localisés sur le brin direct est plus faible que la proportion pour les autres gènes est M. genitalium. Afin de déterminer si ces différences de proportion sont significatives, nous avons effectué un test de χ 2 sur l ensemble des tableaux de contingence à deux lignes et deux colonnes
67 RÉSULTATS BIOLOGIQUES croisant le caractére «brin» au caractère «expression». Dans six espèces seulement (B. burgdorferi, C. trachomatis, E. coli, H. influenzae, R. prowazekii et T. pallidum) cette différence s est avérée significative. 100 % 90 % 80 % 70 % 60 % 50 % 40 % 30 % 20 % 10 % 0 % Bb Bs Cj Ct Ec Hi Hp Mg Mp Mt Rp Tp Figure III.2. Pourcentages de gènes sur les brins directs ( ) et retardés ( ) pour les douze espèces étudiées. La proportion pour les gènes hautement exprimés figure sur la colonne de droite et la proportion pour les autres gènes sur la colonne de gauche. Un autre phénomène est que la dispersion des valeurs de CAI est variable suivant les espèces considérées (Figure III.3). La distribution étant large pour E. coli, plus étroite pour H. influenzae, B. subtilis et M. tuberculosis et très étroite pour l ensemble des huit autres espèces. La variabilité de l usage du code en fonction de l expressivité est donc différente suivant les organismes considérés, avec un maximum pour E. coli (écart type : 0,102), et un minimum pour M. genitalium (écart type : 0,031). 100 % % Cumulé 80 % 60 % 40 % 20 % Ec Hi Mt Bs Ct Tp Bb Mp Hp Cj Rp Mg 0 % 0 0,2 0,4 0,6 0,8 1 CAI Figure III.3. Fonction de répartition des valeurs de CAI. L ordre dans lequel sont listées les abréviations des espèces correspond à l ordre sur le graphe, de la gauche vers la droite (avec en premier E. coli et en dernier M. genitalium). Plus la pente d une courbe est forte et plus la variance des valeurs de CAI est faible dans l espèce considérée
68 CHAPITRE III On constate que cette variabilité du CAI est corrélée de façon significative (r 2 = 0,546, p = 0,0009) à la taille des génomes (Figure III.4). Plus un génome étant de petite taille et plus sa variabilité au niveau des valeurs du CAI étant faible. 0,11 0,1 0,09 Ecart type(cai) 0,08 0,07 0,06 0,05 0,04 0,03 0,02 0 0,5 1 1,5 2 2,5 3 3,5 4 4,5 5 Taille (Mb) Figure III.4. Droite de régression entre la taille des génomes et l écart type des valeurs de CAI DISCUSSION La conclusion de cette étude est qu il existe bien une pression de sélection visant à éviter les collisions frontales entre les DNA et RNA polymérases. Simplement, du fait que cet effet possède une intensité différente en fonction de l espèce considérée, l avantage sélectif procuré est faible (quasi-neutre). La différence entre les espèces peut être interprétée par plusieurs facteurs qui peuvent agir simultanément. Tout d abord l avantage sélectif peut varier du fait de différences dans les vitesses de croissance. Une bactérie possédant une vitesse de croissance rapide duplique son génome plus fréquemment, augmentant de ce fait la fréquence des collisions frontales. Ensuite, le taux d échange entre les brins pour une partie codante (le phénomène dit «d inversion de gène») peut également être différent entre les espèces. Un taux d échange élevé contrebalançant le petit avantage sélectif donné par une orientation sur le brin direct. Enfin, un petit avantage sélectif peut être masqué dans le cas d espèces où la taille des populations est petite, ceci du fait de dérives mutationnelles. 3. VARIATIONS DE COMPOSITION EN BASES ET TRANSFERTS HORIZONTAUX Les transferts horizontaux peuvent être définis comme le passage de DNA d un génome à un autre. Ce terme a été introduit pour mettre l accent sur la différence qui existe avec la transmission verticale de l information génétique des parents à la progéniture. Bien que les transferts horizontaux semblent se produire dans tous les organismes vivants (Mazodier et Davis, 1991), la plupart des études publiées à ce jour ont été réalisées chez les procaryotes. Ceci est compréhensible si l on tient compte du
69 RÉSULTATS BIOLOGIQUES fait que l homogénéité et la relative simplicité des génomes procaryotes permet de détecter aisément des séquences «anormales». Les transferts horizontaux ont fait l objet de nombreuses études car ils sont connus pour jouer un rôle important dans l évolution des génomes bactériens, en particulier dans le domaine de l acquisition de la pathogénicité (Baumler, 1997), ou de la résistance aux antibiotiques et aux métaux lourds (Bize et al., 1999). Dans ce contexte, en collaboration avec Stéphane Guindon, étudiant en thèse au Laboratoire d Informatique, de Robotique de Microelectronique de Montpellier, nous nous sommes intéressés à la validité de certaines des méthodes utilisées pour détecter de telles séquences DÉTECTION PAR APPROCHES INTRINSÈQUES Jusqu à présent, la plupart des méthodes développées pour identifier ces transferts étaient basées sur l hypothèse que les séquences d origine exogène possèdent des caractéristiques différentes de celles d origine endogène. Par exemple, il est possible de définir pour chaque génome un ensemble d oligonucléotides de longueur variable qui constitue le «vocabulaire» spécifique du génome considéré (Pietrokovski et al., 1990). Une confirmation de l existence de cette spécificité de vocabulaire est visualisable sur des AFC réalisées sur les fréquences en oligonucléotides de génomes complets découpés en fragments de un à plusieurs kb. Si l on considère la composition en tétranucléotides des génomes de E. coli, H. influenzae et B. subtilis découpés en fragments de 7 kb, la discrimination entre ces trois espèces atteint 99 % (Figure III.5). 0,6 F2 0,4 0,2 0 F1-0,2-0,4-0,6-0,6-0,4-0,2 0 0,2 0,4 0,6 Figure III.5. Croisement des deux premiers facteurs d une AFC réalisée sur les fréquences en tétranucléotides de E. coli ( ), H. influenzae (+) et B. subtilis ( )
70 CHAPITRE III Du fait de l existence de ces vocabulaires spécifiques, il est possible d utiliser des méthodes de prédiction de parties codantes basées sur des chaînes de Markov cachées pour déterminer si certains gènes ont été obtenus par transfert horizontal (Delcher et al., 1999). De telles méthodes donnent à chaque partie codante détectée une valeur de vraisemblance et, de ce fait, un gène dont on a démontré expérimentalement l existence et qui présentera une faible valeur pourra être considéré comme ayant été obtenu par transfert horizontal. Une autre façon de détecter si des séquences codantes présentent des caractéristiques permettant de les classer comme exogènes est d analyser leurs biais de composition en codons synonymes. De nombreuses études ont été réalisées en utilisant cette approche (Médigue et al., 1991 ; Lawrence et Ochman 1997, 1998 ; Aravind et al., 1998 ; Karlin et al., 1998a, 1998b ; Nelson et al., 1999), et l hypothèse qui leur est sous-jacente est que les codons synonymes utilisés par des gènes transférés depuis des espèces distantes sont différents de ceux figurant dans les séquences endogènes. Dans ces conditions, on peut utiliser des mesures de biais dans l usage des codons pour détecter de telles séquences. En fait, les résultats produits par des approches de ce type sont très dépendants des indices utilisés, mais aussi des variations de composition en bases qui peuvent se produire à l intérieur même des génomes bactériens. Si la composition en bases est inhomogène le long du chromosome pour des raisons n ayant rien à voir avec l existence de transferts horizontaux, de telles méthodes vont avoir tendance à surestimer le nombre des gènes obtenus de cette façon MATÉRIEL ET MÉTHODES Comme les études les plus complètes réalisées jusqu à présent sur les transferts horizontaux ont été effectuées sur E. coli (Lawrence et Ochman, 1997, 1998 ; Karlin et al., 1998b), nous avons décidé de centrer notre analyse sur cette espèce. Qui plus est, du fait qu une liste détaillée de gènes putativement obtenus par transfert horizontal est disponible, nous avons pu l utiliser comme référence (Lawrence et Ochman, 1997). Dans cette étude, les séquences supposées avoir été obtenues par transfert sont celles qui présentent des valeurs atypiques pour la combinaison de deux indices : le CAI et un χ 2 d usage des codons. Un total de 755 gènes fut ainsi identifié par ces deux auteurs. Parmi ces gènes, nous n avons utilisé dans notre étude que ceux qui étaient annotés dans le génome complet d E. coli, soit un total de 317 séquences. Ces gènes sont désignés par l abréviation HT* dans les sections suivantes tandis que les autres gènes sont désignés par non-ht*. La liste complète des gènes HT* est disponible sur le serveur FTP du PBIL 1. Cinq indices d usage des codons ont été utilisés dans notre étude afin de déterminer s il existait des différences significatives permettant de distinguer les gènes HT* des non-ht* : CAI, CAItot, BC, BCtot, et GC3c. Les valeurs de CAI utilisées sont celles figurant dans EMGLib ; la table de référence ayant été établie en utilisant 38 gènes d E. coli putativement hautement exprimés, et la détermination de ces gènes ayant été faite au moyen de la procédure décrite en II Pour les valeurs de CAItot, c est l ensemble des gènes de longueur 150 pb qui a été employé pour construire la table des valeurs de w i et non plus un sous-ensemble constitué de gènes hautement exprimés. 1 ftp://pbil.univ-lyon1.fr/pub/datasets/mbe00/
71 RÉSULTATS BIOLOGIQUES L indice BC (Bias in Codon) (Karlin et al., 1998a) a, quant à lui, été conçu spécialement pour étudier les différences existant au niveau de l usage du code entre deux ensembles de gènes. Les valeurs de BC ont été calculées en prenant comme jeu de données de référence les 38 gènes déjà utilisés pour construire la table de CAI d E. coli. Quant aux valeurs de BCtot, elles ont été calculées de la même façon que celles du CAItot, c est-à-dire en utilisant l ensemble des gènes de longueur 150 pb comme ensemble de référence. Les valeurs de CAItot et de BCtot permettent donc de voir les déviations par rapport à un usage du code «moyen», établi sur l ensemble des gènes d E. coli. Le cinquième indice, GC3c, est tout simplement la mesure du contraste de G+C en troisième position des codons. Pour calculer cet indice, on mesure tout simplement la différence entre le GC3 du gène considéré et la valeur moyenne pour l ensemble des gènes. Pour déterminer lequel de ces cinq indices permet de mieux discriminer les gènes HT* des non-ht*, nous avons employé deux tests non paramétriques. Tout d abord le test de Mann-Whitney pour déterminer si les différences entre les moyennes des distributions pour les valeurs des cinq indices étaient significatives. Puis le test de la variance des rangs pour estimer si les gènes HT* et non-ht* présentaient des valeurs intraclasses similaires mais des valeurs interclasses différentes. Le choix de l utilisation de tests non paramétriques a été guidé par le fait que les distributions de ces cinq scores dans les catégories HT* et non-ht* ne sont généralement pas gaussiennes RÉSULTATS La Table III.3 présente les résultats obtenus dans la caractérisation des gènes HT* au moyen des cinq indices définis précédemment. Comme prévu, les gènes HT* sont associés à des valeurs de CAI, CAItot, BC, BCtot, et GC3c qui sont significativement plus faibles que celles obtenues pour les autres gènes d E. coli. Globalement, les gènes HT* vont donc présenter un usage du code différent de celui des gènes hautement exprimés mais également différent de celui de la moyenne des gènes. Le test de la variance des rangs est également significatif (avec un risque d erreur de 5 %) pour les cinq indices, par contre le degré de significativité le plus élevé est obtenu pour le GC3c. Ce dernier indice est donc celui qui permet de mieux discriminer les gènes HT*. Indice HT* non-ht* MW VR* CAI 0,216 0,278 < ,145 CAItot 0,659 0,712 < ,555 BC 0,876 0,759 < ,150 BCtot 0,467 0,350 < ,567 GC3c 0,152 0,056 < ,758 Table III.3. Caractérisation des gènes HT* au moyen de différents indices d usage du code. Les valeurs moyennes des cinq indices pour les gènes HT* et non-ht* sont données respectivement dans la deuxième et la troisième colonne. MW donne les valeurs de probabilité pour le test de Mann-Whitney de comparaison de moyenne tandis que VR* donne les valeurs du test de variance des rangs. Alors que la distribution des valeurs de GC3c pour les gènes non-ht* suit approximativement une loi normale, celle des gènes HT* est bimodale, avec deux pics centrés autour de -0,17 et 0,17 (Figure III.6). À première vue, cette distribution bimodale semble corroborer l hypothèse selon laquelle les séquences exogènes présentent un contenu en bases atypique, Cependant le déséquilibre en faveur des valeurs négatives est totalement inattendu. Pourquoi le transfert de gènes vers E. coli se ferait-il majoritairement à partir de génomes ayant un contenu en GC3 inférieur? En
72 CHAPITRE III effet, cette espèce présente un taux de G+C proche de 50 %, et la valeur de ce paramètre est approximativement uniformément distribuée entre 25 % et 75 % chez les procaryotes (Sueoka, 1962) Nombre non-ht* HT* ,4-0,3-0,2-0,1 0 0,1 0,2 0,3 0,4 0-0,4-0,3-0,2-0,1 0 0,1 0,2 0,3 0,4 GC3c Figure III.6. Distribution des valeurs de GC3c pour les gènes non-ht* et HT*. Si la distribution est assimilable à une gaussienne pour les gènes non-ht*, les gènes HT* présentent une distribution bimodale décalée en faveur des valeurs négatives. Parmi les 317 gènes HT* étudiés, 254 (80,1 %) sont associés à une valeur négative de GC3c. Dans le but de tester une hypothèse alternative au transfert de gènes pour expliquer ces faibles valeurs, nous avons tracé le graphe des valeurs cumulées de GC3c des gènes d E. coli, ceci en fonction de leur position sur le chromosome (Figure III.7). La courbe obtenue montre clairement que les gènes situés près du terminus de réplication (TerC) ont des valeurs de GC3c négatives. Qui plus est, dans une région de 800 kb située de part et d autre du terminus de réplication, les valeurs de GC3c sont significativement (p < 10-4, test t) inférieures à celles des gènes situés en dehors de cette région CumSum(GC3c) TerC Position (Mb) Figure III.7. Variation des valeurs de GC3c le long du génome d E. coli. Cette courbe a été obtenue en reportant les valeurs cumulées de GC3c pour tous les gènes de longueur 150 pb en fonction de leur position sur le chromosome
73 RÉSULTATS BIOLOGIQUES Par ailleurs, si l on retire, dans ce calcul des valeurs des sommes cumulées de GC3c, les 317 gènes HT*, l allure de la courbe reste inchangée. Ceci prouve que l on a bien une tendance générale à une diminution des valeurs de GC3, et non un effet lié aux seuls gènes HT* DISCUSSION Comme je l ai déjà précisé, l analyse de l usage des codons synonymes a été fréquemment utilisée dans la détection des transferts horizontaux chez les procaryotes. Ceci se comprend parfaitement si l on considère qu il est bien établi que les différentes espèces de procaryotes présentent des variations importantes dans leur composition en codons (Sharp et Matassi, 1994). De ce fait, des séquences exogènes provenant d espèces distantes du point de vue phylogénétique présenteront un usage du code bien différent de celui des séquences endogènes. Toutefois, le problème est que ces méthodes reposent sur une hypothèse qui peut être aisément rejetée : à savoir que la variation intragénomique de l usage des codons synonymes est absente ou suffisamment faible pour pouvoir être négligée. En effet, comme nous l avons vu, la mesure du GC3c constitue le meilleur indice permettant de discriminer les gènes HT*, or il existe une tendance générale pour les gènes d E. coli à voir leurs valeurs de GC3 diminuer au fur et à mesure de l éloignement de l origine de réplication. Ce phénomène avait déjà été mis en évidence par Deschavanne et Filipski (1995), et ces auteurs montraient que cette variation était liée à l existence de différents mécanismes de réparation du DNA le long du génome. Les séquences proches du terminus de réplication existent en simple copie pendant la majeure partie du cycle cellulaire, aussi ont-elles moins la possibilité que celles proches de l origine d être réparées par des mécanismes impliquant une recombinaison homologue. De ce fait, elles ont plus souvent recours au mécanisme de réparation dit de translesion, or ce mécanisme implique l incorporation systématique d une adénine en complément des nucléotides modifiés. Un tel mécanisme va donc induire une dérive mutationnelle vers les bases A+T qui va introduire une composition en codons atypique, cet effet étant d autant plus fort que l on est plus proche du terminus de réplication. Ce phénomène est en accord avec les observations de Lawrence et Ochman (1997) qui montrent que la zone localisée près du terminus (entre les minutes 23 et 47) présente la proportion la plus importante de gènes HT* ; ces auteurs suggérant que ce fait est lié à une haute fréquence de recombinaison dans cette région. Cette affirmation doit être prise avec précaution du fait que Sharp (1991) a montré que le taux de substitutions silencieuses variait de façon significative avec la location sur le chromosome, les gènes proches du terminus présentant une divergence plus importante. De ce fait, plus les séquences sont distantes de l origine de réplication et moins elles vont présenter de similarités avec des orthologues. Ceci impliquant que la probabilité de rencontrer des séquences MEPS (Minimum Efficient Processing Segments), essentielles au mécanisme de recombinaison homologue (Shen and Huang 1989), est plus faible au niveau du terminus
74 CONCLUSIONS PERSPECTIVES
75 CONCLUSIONS PERSPECTIVES CONCLUSIONS PERSPECTIVES Outre la poursuite des développements sur les systèmes préexistants, qu il s agisse du serveur du PBIL ou des différentes banques de données auxquelles j ai contribué, les travaux présentés dans cet ouvrage ouvrent la porte à de nombreux projets de recherche à moyen et long terme. C est sur la description de ces différents projets dont certains ont d ores et déjà été initiés que je voudrais conclure cet ouvrage. 1. BANQUES DE DONNÉES DE GÈNES HOMOLOGUES Dans la continuité d HOBACGEN, nous envisageons de construire des systèmes similaires sur des organismes ou des familles de gènes bien précis. C est ainsi qu en collaboration avec Marc Robinson, enseignant-chercheur au Laboratoire de Biologie Moléculaire et Cellulaire de l ENS de Lyon, nous avons commencé le développement de NuReBase, une banque de données dédiées aux récepteurs nucléaires d hormones. La différence principale avec HOBACGEN et HOVERGEN vient du fait que les différentes familles qui sont présentes dans cette banque sont toutes dérivées d une grande super-famille. Par ailleurs, ce projet visant également à intégrer des données sur la structure tridimensionnelle de ces protéines, il sera nécessaire de modifier l interface HobacFetch pour lui permettre de gérer de telles données. Dans le même ordre d idée, en collaboration avec Hubert Charles, enseignantchercheur au Laboratoire de Biologie Appliquée de l INSA de Lyon, nous avons un projet de construction d une banque dédiée aux gènes de la bactérie Buchnera aphidicola. Cette banque contiendrait les gènes provenant du génome de cette bactérie (qui devrait être disponible sous peu), ainsi que l ensemble de leurs homologues détectés dans les banques. L idée étant d utiliser ces données d homologie pour aider à l annotation du génome, mais également pour étudier l implication de certains gènes dans l acquisition du caractère symbiotique de cette bactérie. Ce projet étant en connection étroite avec celui décrit plus bas, et qui vise à étudier la génomique des bactéries endocytobiotiques. À plus long terme, nous envisageons de construire une banque généraliste qui décrirait les relations d homologie existant entre les gènes de tous les organismes. Ce système remplacerait et complèterait les banques HOVERGEN et HOBACGEN. Ce projet pose des problèmes méthodologiques importants : comment gérer des alignements multiples comprenant des centaines ou milliers de séquences, comment calculer et représenter les arbres phylogénétiques correspondants. Par ailleurs l évolution modulaire des protéines (échange de domaines, par exemple par brassage d exons), et l épissage alternatif sont des processus particulièrement fréquents chez les eucaryotes multicellulaires, processus qui compliquent singulièrement la définition des familles de gènes homologues
76 CONCLUSIONS PERSPECTIVES 2. TRANSFERTS HORIZONTAUX Les différentes bases de données et les logiciels que j ai développés vont constituer les outils principaux utilisés par Vincent Daubin, (étudiant en thèse au Laboratoire de Biométrie et Biologie Évolutive) et moi-même afin d étudier les transferts horizontaux chez les procaryotes. Le but de ce projet est de déterminer quels sont les gènes ayant été obtenus par transfert horizontal chez les bactéries pathogènes dont le génome complet est connu. De nombreuses méthodes de détection de gènes obtenus par transfert horizontal existant, il sera nécessaire, dans un premier temps, de valider (ou d invalider) certaines approches. Une fois une méthodologie ad hoc définie, celle-ci sera appliquée sur un certain nombre d organismes modèles. Après identification d un ensemble de gènes possiblement obtenus par transfert horizontal, nous essaierons de déterminer s il existe un lien entre la présence de ces gènes et l acquisition de la pathogénicité. De nombreuses méthodes permettant d étudier l occurrence de transferts horizontaux ont été développées ces dernières années. Ces méthodes peuvent être classées en deux grands groupes : les méthodes intrinsèques et les méthodes extrinsèques. Les méthodes intrinsèques visent à identifier les gènes issus de transferts horizontaux en utilisant exclusivement l information stockée au sein du génome auquel ils appartiennent. De telles études privilégient ainsi l analyse d un génome du point de vue de la composition en bases ou en codons des séquences présentes en son sein. Ces méthodes sont celles qui ont été utilisées majoritairement pour étudier les transferts jusqu à ce jour. Or, les résultats présentés dans le troisième chapitre montrent que la variation de la composition en bases le long du génome de bactéries comme E. coli semble biaiser les estimations obtenues à partir des méthodes intrinsèques. Il parait donc difficile d accorder une grande confiance aux résultats obtenus avec ce type d approche. Dans le cas des méthodes extrinsèques, celles-ci utilisent des informations issues de différents génomes. Les arbres phylogénétiques constituent dès lors le principal outil d analyse. Qui plus est, contrairement à l approche intrinsèque, il est alors possible d émettre des hypothèses concernant le génome d origine dont est issu le gène transféré horizontalement. À ce jour aucune étude statistique utilisant la phylogénie moléculaire et destinée à identifier des gènes transférés horizontalement n a été réalisée. Il existe donc tout un champ de développement méthodologique à défricher afin de disposer de méthodes fiables faisant appel à ce type de données. Une possibilité serait de développer et de valider un modèle de méthode extrinsèque dont une première ébauche a déjà été definie par Stéphane Guindon au cours de son stage de DEA dans notre laboratoire. La méthode proposée s orienterait vers la comparaison automatique de topologies d arbres phylogénétiques, appliquée spécifiquement à la détection de transferts horizontaux. Avec une telle approche, il serait possible de travailler sur l ensemble des gènes disponibles dans HOBACGEN, ceci quels que soient les génomes auxquels ils appartiennent. Ce travail s articulerait autour de trois étapes : La constitution d une collection d arbres phylogénétiques de gènes orthologues. L extraction, à partir de cette collection, de l ensemble des couples d arbres comparables et le calcul de taux de similarité entre topologies pour chaque comparaison. La détection de séquences potentiellement issues de transferts horizontaux grâce à une analyse des topologies d arbres s écartant du «consensus»
77 CONCLUSIONS PERSPECTIVES La validation de ce modèle pourrait se faire en l appliquant à des organismes bien étudiés comme E. coli ou B. subtilis, pour lesquels de nombreux gènes potentiellement obtenus par transfert horizontal sont connus. Une fois cette validation acquise, nous comptons utiliser cette méthodologie dans le cadre d un projet de recherche conduit en collaboration avec Pascal Simonet du Laboratoire d Écologie Microbienne du Sol. Nous sommes en effet impliqués, avec ce laboratoire, dans un projet visant à étudier les transferts horizontaux chez un pathogène des plantes : Ralstonia solanacearum. L équipe dirigée par P. Simonet s occupant de l aspect expérimental de cette étude tandis que notre équipe se chargera des aspects bioinformatiques. Une fois le génome complet de R. solanacearum séquencé, nous comptons déterminer quels sont les gènes ayant été obtenus par transfert chez cette bactérie. Après cela, il sera possible de voir si les gènes impliqués dans l acquisition du caractère pathogène font partie ou non de la liste de ceux prédits comme ayant été obtenus par transfert horizontal. 3. GÉNOMIQUE DES BACTÉRIES ENDOCYTOBIOTIQUES L acquisition de la vie symbiotique au sens large, c est-à-dire incluant le parasitisme, s accompagne chez les eucaryotes comme chez les procaryotes de la perte d un certain nombre de fonctions, désormais assurées par l hôte. D un autre côté, d autres fonctions, responsables des relations particulières entre le parasite et son hôte, sont acquises. Figurent parmi ces fonctions celles liées à la spécificité et à l effet pathogène. C est donc au niveau des génomes que doivent être recherchés les témoins de cette évolution et les clés du fonctionnement actuel de l association. Parmi les bactéries parasites endocellulaires, nombreuses sont celles qu on ne peut cultiver. L analyse génomique de ces bactéries non cultivables, malgré tout son intérêt, est donc encore peu avancée et, par voie de conséquence, l interprétation des relations évolutives et fonctionnelles avec les hôtes en est à ses débuts. En collaboration avec P. Simonet et avec Yvan Rahbé, du Laboratoire de Biologie Appliquée de l INSA de Lyon, nous avons déposé un projet visant à effectuer une étude comparative de plusieurs bactéries associées à différents hôtes, ceci dans le but d élucider les phénomènes évolutifs ayant conduit à l acquisition et au perfectionnement de la fonction symbiotique (ou parasitaire). Les modèles biologiques considérés sont d une part, les bactéries parasites intracellulaires et pathogènes de l Homme et d autre part, les bactéries non cultivables des genres Wolbachia et Buchnera, symbiotes de différentes espèces d arthropodes. Des projets de séquençage complet sont pratiquement achevés (Buchnera) ou en cours (Wolbachia) pour deux espèces appartenant à ces genres. L intérêt de ces deux modèles est renforcé par leur importance agro-alimentaire et médicale. Buchnera est mutualiste obligatoire des pucerons, insectes ravageurs des cultures dont le contrôle chimique devenu très difficile pose d épineuses questions de sécurité alimentaire. Wolbachia, parasite de la reproduction des insectes, induit chez ses hôtes des pathologies dont on cherche à utiliser le caractère invasif pour contrôler les populations vectorielles (en particulier les moustiques). Les différents groupes considérés offrent donc une grande diversité à la fois au niveau de la spécificité d hôte (très faible pour des bactéries du genre Mycoplasma ou Buchnera, très large pour Wolbachia) et de leur façon d interagir avec celui-ci (du parasitisme au mutualisme). Les résultats attendus de cette étude devraient permettre de répondre aux questions centrales liées au parasitisme intracellulaire : Quelles sont les fonctions responsables d une spécificité d hôte plus ou moins grande? Quelles sont les différences au niveau
78 CONCLUSIONS PERSPECTIVES génomique qui peuvent expliquer qu une bactérie sera un symbiote plutôt qu un parasite? Quels sont les gènes qui ont été acquis ou perdus dans chacun de ces deux cas de figure? Y a-t-il eu des transferts horizontaux réalisés entre l hôte et le parasite, et si oui, quels gènes ont été impliqués? Quels mécanismes régulatoires caractérisent la réponse d une bactérie intracellulaire (vs. libre) à des stress imposés au symbiocosme (stress trophiques, changements d hôtes?)
79 RÉFÉRENCES BIBLIOGRAPHIQUES RÉFÉRENCES BIBLIOGRAPHIQUES A Alm, R.A., Ling, L.-S.L., Moir, D.T., King, B.L., Brown, E.D., Doig, P.C., Smith, D.R., Noonan, B., Guild, B.C., de Jonge, B.L. et al. (1999) Genomic sequence comparison of two unrelated isolates of the human gastric pathogen Helicobacter pylori. Nature, 397, Altschul, S.F., Gish, W., Miller, W., Myers, E.W. et Lipman, D.J. (1990) Basic local alignment search tool. J. Mol. Biol., 215, Altschul, S.F., Madden, T.L., Schäffer, A.A., Zhang, J., Zhang, Z., Miller, W. et Lipman, D.J. (1997) Gapped BLAST and PSI-BLAST : a new generation of protein database search programs. Nucleic Acids Res., 25, Andersson, S.G.E., Zomorodipour, A., Andersson, J.O., Sicheritz-Pontén, T., Alsmark, U.C.M., Podowski, R.M., Naeslund, A.K., Eriksson, A.S., Winkler, H.H. et Kurland, C.G. (1998) The genome sequence of Rickettsia prowazekii and the origin of mitochondria. Nature, 396, Aravind, L., Tatusov, R.L., Wolf, Y.I., Walker, D.R. et Koonin, E.V. (1998) Evidence for massive gene exchange between archaeal and bacterial hyperthermophiles. Trends Genet., 14, Argos, P., Rao, J.K. et Hargrave, P.A. (1982) Structural prediction of membrane-bound proteins. Eur. J. Biochem., 128, Attwood, T.K., Croning, M.D.R., Flower, D.R., Lewis, A.P., Mabey, J.E., Scordis, P., Selley, J.N. et Wright, W. (2000) PRINTS-S : the database formerly known as PRINTS. Nucleic Acids Res., 28, B Bairoch, A. (2000) The ENZYME database in Nucleic Acids Res., 28, Bairoch, A. et Apweiler, R. (2000) The SWISS-PROT protein sequence database and its supplement TrEMBL in Nucleic Acids Res., 28, Baker, W., Van den Broek, A., Camon, E., Hingamp, P., Sterk, P., Stoesser, G. et Tuli, M.A. (2000) The EMBL nucleotide sequence database. Nucleic Acids Res., 28, Ball, C.A., Dolinski, K., Dwight, S.S., Harris, M.A., Issel-Tarver, L., Kasarskis, A., Scafe, C.R., Sherlock, G., Binkley, G., Jin, H. et al. (2000) Integrating functional genomic information into the Saccharomyces Genome Database. Nucleic Acids Res., 28, Barker, W.C., Pfeiffer, F. et George, D.G. (1996) Superfamily classification in the PIR-International protein sequence database. Methods Enzymol., 266,
80 RÉFÉRENCES BIBLIOGRAPHIQUES Barker, W.C., Garavelli, J.S., Huang, H., McGarvey, P.B., Orcutt, B.C., Srinivasarao, G.Y., Xiao, C., Yeh, L.-S.L., Ledley, R.S., Janda, J.F. et al. (2000) The Protein Information Resource (PIR). Nucleic Acids Res., 28, Bateman, A., Birney, E., Durbin, R., Eddy, S.R., Howe, K.L. et Sonnhammer, E.L.L. (2000) The Pfam protein families database. Nucleic Acids Res., 28, Baumler, A.J. (1997) The record of horizontal gene transfer in Salmonella. Trends. Microbiol., 5, Benson, D.A., Karsch-Mizrachi, I., Lipman, D.J., Ostell, J., Rapp, B.A. et Wheeler, D.L. (2000) GenBank. Nucleic Acids Res., 28, Bergh, S. et Cole, S.T. (1994) MycDB : an integrated mycobacterial database. Mol. Microbiol., 12, Berman, H.M., Westbrook, J., Feng, Z., Gilliland, G., Bhat, T.N., Weissig, H., Shindyalov, I.N. et Bourne, P. (2000) The Protein Data Bank. Nucleic Acids Res., 28, Biaudet, V., Samson, F., Anagnostopoulos, C., Ehrlich, S.D. et Bessières, P. (1996) Computerized genetic map of Bacillus subtilis. Microbiology, 142, Biaudet, V., Samson, F. et Bessières, P. (1997) Micado a network-oriented database for microbial genomes. Comput. Applic. Biosci., 13, Bize, L., Muri, F., Samson, F., Rodolphe, F., Ehrlich, S.D., Prum, B. et Bessières, P. (1999) Searching gene transfers on Bacillus subtilis using hidden Markov models. In Proceedings of the Third Annual International Conference on Computational Molecular Biology, Istrail, S., Pevzner, P. et Waterman, M. (eds.), ACM Press, New-York, pp Blake, J.A., Eppig, J.T., Richardson, J.E., Davisson, M.T. et the Mouse Genome Database Group (2000) The Mouse Genome Database (MGD) : expanding genetic and genomic resources for the laboratory mouse. Nucleic Acids Res., 28, Blattner, F.R., Plunkett III, G., Bloch, C.A., Perna, N.T., Burland, V., Rilley, M., Collado-Vides, J., Glasner, J.D., Rode, C.K., Mayhew, G.F. et al. (1997) The complete genome sequence of Escherichia coli K-12. Science, 277, Bleasby, A.J., Akrigg, D. et Attwood, T.K. (1994) OWL a non-redundant composite protein sequence database. Nucleic Acids Res., 22, Brewer, B.J. (1988) When polymerase collide : replication and the transcriptional organization of the E. coli chromosome. Cell, 53, Bult, C.J., White, O., Olsen, G.J., Zhou, L., Fleischmann, R.D., Sutton, G.G., Blake, J.A., FitzGerald, L.M., Clayton, R.A., Gocayne, J.D. et al. (1996) Complete genome sequence of the methanogenic archeon, Methanococcus jannaschii. Science, 273, C Cherry, J.M., Cartinhour, S.W. et Goodman, H.M. (1992) AAtDB, an Arabidopsis thaliana database. Plant Mol. Biol. Rep., 10, Cole, S.T., Brosch, R., Parkhill, J., Garnier, T., Churcher, C., Harris, D., Gordon, S.V., Eiglmeier, K., Gas, S., Barry III, C.E. et al. (1998) Deciphering the biology of Mycobacterium tuberculosis from the complete genome sequence. Nature, 393,
81 RÉFÉRENCES BIBLIOGRAPHIQUES Corpet, F. (1988) Multiple sequence alignment with hierarchical clustering. Nucleic Acids Res., 16, Corpet, F., Servant, F., Gouzy, J. et Kahn, D. (2000) ProDom and ProDom-CG : tools for protein domain analysis and whole genome comparisons. Nucleic Acids Res., 28, Costanzo, M.C., Hogan, J.D., Cusick, M.E., Davis, B.P., Fancher, A.M., Hodges, P.E., Kondu, P., Lengieza, C., Lew-Smith, J.E., Lingner, C. et al. (2000) The Yeast Proteome Database (YPD) and Caenorhabditis elegans Proteome Database (WormPD) : comprehensive resources for the organization and comparison of model organism protein information. Nucleic Acids Res., 28, Cuff, J.A. et Barton, G.J. (1999) Evaluation and improvement of multiple sequence methods for protein secondary structure prediction. Proteins, 34, D-E Dayhoff, M.O., Eck, R.V., Chang, M.A. et Sochard, M.R. (1965) Atlas of Protein Sequence and Structure. National Biomedical Research Foundation, Silver Spring. Dayhoff, M.O., Schwartz, R.M. et Orcutt, B.C. (1978) A model of evolutionary changes in proteins. In Atlas of Protein Sequence and Structure, Vol. 5, Dayhoff, M.O. (ed.), National Biomedical Research Foundation, Washington DC, pp Deckert, G., Warren, P.V., Gaasterland, T., Young, W.G., Lenox, A.L., Graham, D.E., Overbeek, R., Snead, M.A., Keller, M., Aujay, M. et al. (1998) The complete genome of the hyperthermophilic bacterium Aquifex aeolicus. Nature, 392, Delcher, A.L., Harmon, D., Kasif, S., White, O. et Salzberg, S.L. (1999) Improved microbial gene identification with GLIMMER. Nucleic Acids Res., 27, De Rijk, P., Wuyts, J., Van de Peer, Y., Winkelmans, T. et De Wachter, R. (2000) The European large subunit ribosomal RNA database. Nucleic Acids Res., 28, Devereux, J., Haeberli, P. et Smithies, O. (1984) A comprehensive set of sequence analysis programs for the VAX. Nucleic Acids Res., 12, Deschavanne, P. et Filipski, J. (1995) Correlation of GC content with replication timing and repair mechanisms in weakly expressed E. coli genes. Nucleic Acids Res., 23, Durbin, R. et Thierry-Mieg, J. (1994) The ACeDB genome database. In Computational Methods in Genome Research, Suhai, S. (ed.), Plenum Press, New- York, pp Duret, L., Gasteiger, E. et Perrière, G. (1996) LALNVIEW : a graphical viewer for pairwise sequence alignment. Comput. Applic. Biosci., 12, Duret, L., Perrière, G. et Gouy, M. (1999) HOVERGEN : database and software for comparative analysis of homologous vertebrate genes. In Bioinformatics Databases and Systems, Letovsky, S. (ed.), Kluwer Academic Publishers, Boston, pp Etzold, T.A., Ulyanov, P. et Argos, P. (1996) SRS : information retrieval for molecular biology databanks. Methods Enzymol., 266, F Faulkner, D.V. et Jurka, J. (1988) Multiple aligned sequence editor (MASE). Trends Biochem. Sci., 13,
82 RÉFÉRENCES BIBLIOGRAPHIQUES Fichant, G. et Gautier, C. (1987) Statistical methods for predicting protein coding regions in nucleic acids sequences. Comput. Applic. Biosci., 3, Fichant, G.A. et Quentin,Y. (1995) A frameshift error detection algorithm for DNA sequencing projects. Nucleic Acids Res., 23, Fitch, W.M. (1966) Mutation values for the interconversion of amino acid pairs. J. Mol. Biol., 16, Felsenstein, J. (1989) PHYLIP : phylogeny inference package (version 3.2). Cladistics, 5, Fleischmann, R.D., Adams, M.D., White, O., Clayton, R.A., Kirkness, E.F., Kerlavage, A.R., Bult, C.J., Tomb, J.-F., Dougherty, B.A., Merrick, J.M. et al. (1995) Wholegenome random sequencing and assembly of Haemophilus influenzae Rd. Science, 269, FlyBase Consortium (1999) The FlyBase database of the Drosophila genome projects and community litterature. Nucleic Acids Res., 27, Francino, M.P. et Ochman, H. (1997) Strand asymetries in DNA evolution. Trends Genet., 13, Frank, A.C. et Lobry, J.R. (2000) Oriloc : prediction of replication boundaries in unannotated bacterial chromosomes, Bioinformatics, 16, sous presse. Fraser, C.M., Casjens, S., Huang, W.M., Sutton, G.G., Clayton, R., Lathigra, R., White, O., Ketchum, K.A., Dodson, R., Hickey, E.K. et al. (1997) Genomic sequence of a Lyme disease spirochaete, Borrelia burgdorferi. Nature, 390, Fraser, C.M., Gocayne, J.D., White, O., Adams, M.D., Clayton, R.A., Fleischmann, R.D., Bult, C.J., Kerlavage, A.R., Sutton, G., Kelley, J.M. et al. (1995) The minimal gene complement of Mycoplasma genitalium. Science, 270, Fraser, C.M., Norris, S.J., Weinstock, G.M., White, O., Sutton, G.G., Dodson, R., Gwinn, M., Hickey, E.K., Clayton, R., Ketchum, K.A. et al. (1998) Complete genome sequence of Treponema pallidum, the syphilis spirochete. Science, 281, French, S. (1992) Consequences of replication fork movement through transcription units in vivo. Science, 258, G Gai, X., Lal, S., Xing, L., Brendel, V. et Walbot, V. (2000) Gene discovery using the maize genome database ZmDB. Nucleic Acids Res., 28, Galtier, N., Gouy, M. et Gautier, C. (1996) SEAVIEW and PHYLO_WIN : two graphic tools for sequence alignment and molecular phylogeny. Comput. Applic. Biosci., 12, Gascuel, O. (1997) BIONJ : an improved version of the NJ algorithm based on a simple model of sequence data. Mol. Biol. Evol., 14, Geourjon, C. et Deléage, G. (1995) SOPMA : significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments. Comput Applic. Biosci., 11, Gibrat, J.F., Madej, T. et Bryant, S.H. (1996) Surprising similarities in structure comparison. Curr. Opin. Struct. Biol., 6,
83 RÉFÉRENCES BIBLIOGRAPHIQUES Goffeau, A., Barrell, B.G., Bussey, H., Davis, R.W., Dujon, B., Feldmann, H., Galibert, F., Hoheisel, J.D., Jacq, C., Johnston, M. et al. (1996) Life with 6000 genes. Science, 274, 546. Gouy, M. et Gautier, C. (1982) Codon usage in bacteria : correlation with gene expressivity. Nucleic Acids Res., 10, Gouy, M., Gautier, C., Attimonelli, M., Lanave, C. et di Paola, G. (1985) ACNUC a portable retrieval system for nucleic acid sequence databases : logical and physical designs and usage. Comput. Applic. Biosci., 1, Gouzy, J., Corpet, F. et Kahn, D. (1999) Whole genome protein domain analysis using a new method for domain clustering. Comput. Chem., 23, Gower, J.C. (1966) Some distance properties of latent root and vector methods used in multivariate analysis. Biometrika, 53, Gracy, J. et Argos, P. (1998) DOMO : a new database of aligned protein domains. Trends Biochem. Sci., 23, Grantham, R. et Gautier, C. (1980) Genetic distances from mrna sequences. Naturwissenschaften, 67, Grantham, R., Gautier, C. et Gouy, M. (1980a) Codon frequencies in 119 individual genes confirm consistent choices of degenerate base according to genome type. Nucleic Acids Res., 8, Grantham, R., Gautier, C., Gouy, M., Mercier, R. et Pavé, A. (1980b) Codon catalog usage and the genome hypothesis. Nucleic Acids Res., 8, r49-r62. Gribskov, M., Luethy, R. et Eisenberg, D. (1990) Profile analysis. Methods Enzymol., 183, H Henikoff, J.G., Greene, E.A., Pietrokovski, S. et Henikoff, S. (2000) Increased coverage of protein families with the Blocks database servers. Nucleic Acids Res., 28, Henikoff, S. et Henikoff, J.G. (1992) Amino acid substitution matrices from protein blocks. Proc. Natl. Acad. Sci. USA, 89, Higgins, D.G. (1992) Sequence ordinations : a multivariate analysis approach to analysing large sequence data sets. Comput. Applic. Biosci., 8, Higgins, D.G., Thompson, J.D. et Gibson, T.J. (1996) Using CLUSTAL for multiple sequence alignments. Methods Enzymol., 266, Hill, M.O. (1974) Correspondence analysis : a neglected multivariate method. Appl. Stat., 23, Himmelreich, R., Hilbert, H., Plagens, H., Pirkl, E., Li, B.-C. et Herrmann, R. (1996) Complete sequence analysis of the genome of the bacterium Mycoplasma pneumoniae. Nucleic Acids Res., 24, Hofmann, K., Bucher, P., Falquet, L. et Bairoch, A. (1999) The PROSITE database, its status in Nucleic Acids Res., 27, Holm, L. (1986) Codon usage and gene expression. Nucleic Acids Res., 14,
84 RÉFÉRENCES BIBLIOGRAPHIQUES Holm, L. et Sander, C. (1999) Protein folds and families : sequence and structure alignments. Nucleic Acids Res., 27, Hoogland, C., Sanchez, J.-C., Tonella, L., Binz, P.-A., Bairoch, A., Hochstrasser, D.F. et Appel, R.D. (2000) The 1999 SWISS-2DPAGE database update. Nucleic Acids Res., 28, Huang, H., Xiao, C. et Wu, C.H. (2000) ProClass protein family database. Nucleic Acids Res., 28, Huang, X. et Miller, W. (1991) A time-efficient, linear-space local similarity algorithm. Adv. Appl. Math., 12, I-K Iida, Y. (1988) Categorical discriminant analysis of 3 -splice site signals of mrna precursors in higher eucaryote genes. J. Theoret. Biol., 135, Ikemura, T. (1981) Correlation between the abundance of Escherichia coli transfer RNAs and the occurrence of the respective codons in its protein genes. J. Mol. Biol., 146, Kallberg, Y. et Persson, B. (1999) KIND a non-redundant protein database. Bioinformatics, 15, Kalman, S., Mitchell, W., Marathe, R., Lammel, C., Fan, J., Olinger, L., Grimwood, J., Davis, R.W. et Stephens, R.S. (1999) Comparative genomes of Chlamydia pneumoniae and C. trachomatis. Nature Genet., 21, Kanehisa, M. (1988) A multivariate analysis method for discriminating protein secondary structural segments. Protein Eng., 2, Kaneko, T., Sato, S., Kotani, H., Tanaka, A., Asamizu, E., Nakamura, Y., Miyajima, N., Hirosawa, M., Sugiura, M., Sasamoto, S. et al. (1996) Sequence analysis of the genome of the unicellular cyanobacterium Synechocystis sp. PCC6803. II. Sequence determination of the entire genome and assignment of potential proteincoding regions. DNA Res., 3, Karlin, S., Campbell, A.M. et Mrazek, J. (1998a) Comparative DNA analysis across diverse genomes. Annu. Rev. Genet., 32, Karlin, S., Mrazek, J. et Campbell, A.M. (1998b) Codon usage in different classes of the Escherichia coli genome. Mol. Microbiol., 29, Kawarabayasi, Y., Hino, Y., Horikawa, H., Yamazaki, S., Haikawa, Y., Jin-no, K., Takahashi, M., Sekine, M., Baba, S., Ankai, A., Kosugi, H. et al. (1999) Complete genome sequence of an aerobic hyper-thermophilic crenarchaeon, Aeropyrum pernix K1. DNA Res., 6, Kawarabayasi, Y., Sawada, M., Horikawa, H., Haikawa, Y., Hino, Y., Yamamoto, S., Sekine, M., Baba, S., Kosugi, H., Hosoyama, A. et al. (1998) Complete sequence and gene organization of the genome of a hyper-thermophilic archaebacterium, Pyrococcus horikoshii OT3. DNA Res., 5, Klein, P., Jacquez, J.A. et DeLisi, C. (1986) Prediction of protein function by discriminant analysis. Math. Biosci., 81, Klein, P., Kanehisa, M. et DeLisi, C. (1984) Prediction of protein function from sequence properties discriminant analysis of a data base. Biochim. Biophys. Acta, 787,
85 RÉFÉRENCES BIBLIOGRAPHIQUES Klein, P., Kanehisa, M. et DeLisi, C. (1985) The detection and classification of membrane-spanning proteins. Biochim. Biophys. Acta, 815, Klenk, H.P., Clayton, R.A., Tomb, J., White, O., Nelson, K.E., Ketchum, K.A., Dodson, R.J., Gwinn, M., Hickey, E.K., Peterson, J.D. et al. (1997) The complete genome sequence of the hyperthermophilic, sulphate-reducing archaeon Archaeoglobus fulgidus. Nature, 390, Krause, A., Stoye, J. et Vingron, M. (2000) The SYSTERS protein sequence cluster set. Nucleic Acids Res., 28, Kunst, F., Ogasawara, N., Moszer, I., Albertini, A.M., Alloni, G., Azevedo, V., Bertero, M.G., Bessières, P., Bolotin, A., Borchert, S. et al. (1997) The complete genome sequence of the Gram-positive bacterium Bacillus subtilis. Nature, 390, Kyte, J. et Doolittle, R.F. (1982) A simple method for displaying the hydropathic character of a protein. J. Mol. Biol., 157, L Labedan, B. et Riley, M. (1999) Genetic inventory : Escherichia coli as a window on ancestral proteins. In Organization of the Prokaryotic Genome, Charlebois, R. (ed.), ASM Press, Washington DC, pp Lafay, B., Lloyd, A.T., McLean, M.J., Devine, K.M., Sharp, P.M. et Wolfe, K.H. (1999) Proteome composition and codon usage in spirochaetes : species-specific and DNA strand-specific mutational biases. Nucleic Acids Res., 27, Lapointe, F.J. et Kirsch, J.A.W. (1995) Estimating phylogenies from lacunose distances matrices, with special reference to DNA hybridization data. Mol. Biol. Evol., 12, Lawrence, J.G. et Ochman, H. (1997) Amelioration of bacterial genomes : rates of change and exchange. J. Mol. Evol., 44, Lawrence, J.G. et Ochman, H. (1998) Molecular archaeology of the Escherichia coli genome. Proc. Natl. Acad. Sci. USA, 95, Letovsky, S. (1999) GDB : integrating genomic maps. In Bioinformatics Databases and Systems, Letovsky, S. (ed.), Kluwer Academic Publishers, Boston, pp Liu, B. et Alberts, B.M. (1995) Head-on collision between a DNA replication apparatus and RNA polymerase transcription complex. Science, 267, Lobry, J.R. (1996a) Asymmetric substitution patterns in the two DNA strands of bacteria. Mol. Biol. Evol., 13, Lobry, J.R. (1996b) Origin of replication of Mycoplasma genitalium. Science, 272, Lobry, J.R. et Gautier, C. (1994) Hydrophobicity, expressivity and aromaticity are the major trends of amino-acid usage in 999 Escherichia coli chromosome-encoded genes. Nucleic Acids Res., 22, Lo Conte, L., Ailey, B., Hubbard, T.J.P., Brenner, S.E., Murzin, A.G. et Chothia, C. (2000) SCOP : a Structural Classification of Proteins database. Nucleic Acids Res., 28, Luethy, R., Xenarios, I. et Bucher, P. (1994) Improving the sensitivity of the sequence profile method. Protein Sci., 3,
86 RÉFÉRENCES BIBLIOGRAPHIQUES M McEntyre, J. (1998) Linking up with Entrez. Trends Genet., 14, McInerney, J.O. (1997) Replicational and transcriptional selection on codon usage in Borrelia burgdorferi. Proc. Natl. Acad. Sci. USA, 95, Maidak, B.L., Cole, J.R., Lilburn, T.G., Parker Jr., C.T., Saxman, P.R., Stredwick, J.M., Garrity, G.M., Li, B., Olsen, G.J., Pramanik, S. et al. (2000) The RDP (Ribosomal Database Project) continues. Nucleic Acids Res., 28, Marsh, R. et Worcel, A. (1977) A DNA fragment containing the origin of replication of the Escherichia coli chromosome. Proc. Natl. Acad. Sci. USA, 74, Mazodier, P. et Davies, J. (1991) Gene transfer between distantly related bacteria. Annu. Rev. Genet., 25, Médigue, C., Rouxel, T., Vigier, P., Hénaut, A. et Danchin, A. (1991) Evidence for horizontal gene transfer in Escherichia coli speciation. J. Mol. Biol., 222, Médigue, C., Viari, A., Hénaut, A. et Danchin, A. (1993) Colibri : a functional data base for the Escherichia coli genome. Microbiol. Rev., 57, Mewes, H.W., Frishman, D., Gruber, C., Geier, B., Haase, D., Kaps, A., Lemcke, K., Mannhaupt, G. Pfeiffer, F., Schüller, C., Stocker, S. et Weil, B. (2000) MIPS : a database for genomes and protein sequences. Nucleic Acids Res., 28, Moszer, I., Glaser, P. et Danchin, A. (1995) SubtiList : a relational data base for the Bacillus subtilis genome. Microbiology, 141, Murvai, J., Vlahovicek, K., Barta, E., Cataletto, B. et Pongor, S. (2000) The SBASE protein domain library, release 7.0 : a collection of annotated protein sequence segments. Nucleic Acids Res., 28, Mushegian, A.R. et Koonin, E.V. (1996) A minimal gene set for cellular life derived by comparison of complete bacterial genomes. Proc. Natl. Acad. Sci. USA, 93, N-O Nakai, K. et Kanehisa, M. (1991) Expert system for predicting protein localization sites in gram-negative bacteria. Proteins, 11, Nakashima, H. et Nishikawa, K. (1994) Discrimination of intracellular and extracellular proteins using amino acid composition and residue-pair frequencies. J. Mol. Biol., 238, Nakata, K., Kanehisa, M. et DeLisi, C. (1985) Prediction of splice junctions in mrna sequences. Nucleic Acids Res., 13, Nelson, K.E., Clayton, R.A., Gill, S.R., Gwinn, M.L., Dodson, R.J., Haft, D.H., Hickey, E.K., Peterson, J.D., Nelson, W.C., Ketchum, K.A. et al. (1999) Evidence of lateral gene transfer between archaea and bacteria from genome sequence of Thermotoga maritima. Nature, 399, Ogasawara, N., Mizumoto, S. et Yoshikawa, H. (1984) Replication origin of the Bacillus subtilis chromosome determined by hybridization of the first-replicating DNA with cloned fragments from the replication region of the chromosome. Gene, 30,
87 RÉFÉRENCES BIBLIOGRAPHIQUES Okazaki, R., Okazaki, T. Sakabe, K., Sugimoto, K., Kainuma, R., Sugino, A., et Iwatsuki, N. (1968) In vivo mechanism of DNA chain growth. Cold Spring Harbor Symp. Quant. Biol., 33, Oliver, S.G. (1996) A network approach to the systematic analysis of yeast gene function. Trends Genet., 12, P Parker, J.M.R., Guo, D. et Hodges, R.S. (1986) New hydrophilicity scale derived from high-performance liquid chromatography peptide retention data : correlation of predicted surface residues with antigencity and x-ray-derived accessible sites. Biochemistry, 25, Parkhill, J., Wren, B.W., Mungall, K., Ketley, J.M., Churcher, C., Basham, D., Chillingworth, T., Davies, R.M., Feltwell, T., Holroyd, S. et al. (2000) The genome sequence of the food-borne pathogen Campylobacter jejuni reveals hypervariable sequences. Nature, 403, Pattabiraman, N., Namboodiri, K., Lowrey, A. et Gaber, B.P. (1990) NRL_3D : a sequence-structure database derived from the protein data bank (PDB) and searchable within the PIR environment. Protein Seq. Data Anal., 3, Patthy, L. (1991) Modular exchange principles in proteins. Curr. Opin. Struct. Biol., 1, Patthy, L. (1994) Introns and exons. Curr. Opin. Struct. Biol., 4, Pearson, W.R. et Lipman, D.J. (1988) Improved tools for biological sequence comparison. Proc. Natl. Acad. Sci. USA, 85, Perrière, G., Duret, L. et Gouy, M. (2000) HOBACGEN : database system for comparative genomics in bacteria. Genome Res., 10, Perrière, G. et Gouy, M. (1996) WWW-Query : an on-line retrieval system for biological sequence banks. Biochimie, 78, Perrière, G., Gouy, M. et Gojobori, T. (1994) NRSub : a non-redundant data base for the Bacillus subtilis genome. Nucleic Acids Res., 22, Perrière, G., Gouy, M. et Gojobori, T. (1998) The non-redundant Bacillus subtilis (NRSub) database : update Nucleic Acids Res., 26, Perrière, G., Labedan, B. et Bessières, P. (1999) EMGLib : the enhanced microbial genomes library. Nucleic Acids Res., 27, Perrière, G., Labedan, B. et Bessières, P. (2000) EMGLib : the Enhanced Microbial Genomes Library (update 2000). Nucleic Acids Res., 28, Perrière, G., Lobry, J.R. et Thioulouse, J. (1996a) Correspondence discriminant analysis : a multivariate method for comparing classes of protein and nucleic acids sequences. Comput. Applic. Biosci., 12, Perrière, G., Moszer, I. et Gojobori, T. (1996b) NRSub : a non-redundant database for Bacillus subtilis. Nucleic Acids Res., 24, Perrière, G. et Thioulouse, J. (1996) On-line tools for sequence retrieval and multivariate statistics in molecular biology. Comput. Applic. Biosci., 12, Petsch, M.C., Well, T.N., Stampf, D.R. et Sussman, J.L. (1995) The SWISS- 3DIMAGE collection and PDB-browser on the World-Wide Web. Trends Biochem. Sci., 20,
88 RÉFÉRENCES BIBLIOGRAPHIQUES Pietrokovski, S., Hirshon, J. et Trifonov, E.N. (1990) Linguistic measure of taxonomic and functional relatedness of nucleotide sequences. J. Biomol. Struct. Dyn., 7, Q-R Quentin, Y. (1988) The Alu family developed through successive waves of fixation closely connected with primate lineage history. J. Mol. Evol., 27, Quentin, Y. (1989) Successive waves of fixation of B1 variants in rodent lineage history. J. Mol. Evol., 28, Read, T.D., Brunham, R.C., Shen, C., Gill, S.R., Heidelberg, J.F., White, O., Hickey, E.K., Peterson, J., Utterback, T., Berry, K. et al. (2000) Genome sequences of Chlamydia trachomatis MoPn and Chlamydia pneumoniae AR39. Nucleic Acids Res., 28, Rhee, S.Y., Weng, S., Bongard-Pierce, D.K., Garcia-Hernández, M., Malekian, A., Flanders, D.J. et Cherry, J.M. (2000) Unified display of Arabidopsis thaliana physical maps from AtDB, the A. thaliana database. Nucleic Acids Res., 28, Riley, M. et Labedan, B. (1997) Protein evolution viewed through Escherichia coli protein sequences : introducing the notion of structural segment of homology, the module. J. Mol. Biol., 269, Ritter, O., Kocab, P., Senger, M., Wolf, D. et Suhai, S. (1994) Prototype implementation of the Integrated Genomic Database. Comput. Biomed. Res., 27, Rudd, K.E. (1993) Maps, genes, sequences, and computers : an Escherichia coli case study. ASM News, 59, Rudd, K.E. (2000) EcoGene : a genome sequence database for Escherichia coli K-12. Nucleic Acids Res., 28, S Sakata, K., Antonio, B.A., Mukai, Y., Nagasaki, H., Sakai, Y., Makino, K. et Sasaki, T. (2000) INE : a rice genome database with an integrated map view. Nucleic Acids Res., 28, Salazar, L., Fsihi, H., de Rossi, E., Riccardi, G., Rios, C., Cole, S.T. et Takiff, H.E. (1996) Organization of the origins of replication of the chromosomes of Mycobacterium smegmatis, Mycobacterium leprae and Mycobacterium tuberculosis and isolation of a functional origin from M. smegmatis. Mol. Microbiol., 20, Schuler, G.D., Epstein, J.A., Ohkawa, H. et Kans, J.A. (1996) Entrez : molecular biology database and retrieval system. Methods Enzymol., 266, Scott, A.F., Amberger, J., Brylawski, B. et McKusick, V.A. (1999) OMIM : Online Mendelian Inheritance in Man. In Bioinformatics Databases and Systems, Letovsky, S. (ed.), Kluwer Academic Publishers, Boston, pp Seavey, B.R., Farr, E.A., Westler, W.M. et Markley, J.L. (1991) A relational database for sequence-specific protein NMR data. J. Biomol. NMR, 1, Sharp, P.M. (1991) Determination of DNA sequence divergence between Escherichia coli and Salmonella typhimurium : codon usage, map position, and concerted evolution. J. Mol. Evol., 33,
89 RÉFÉRENCES BIBLIOGRAPHIQUES Sharp, P.M., Higgins, D.G., Shields, D.C., Devine, K.M. et Hoch, J.A. (1990) Bacillus subtilis gene sequences. In Genetics and Biotechnology of Bacilli, Zukowski, M.M., Ganesan, A.T. et Hoch, J.A. (eds.), Academic Press, San Diego, pp Sharp, P.M. et Li, W.-H. (1987) The codon adaptation index a measure of directional synonymous codon usage bias, and its potential applications. Nucleic Acids Res., 15, Sharp, P.M. et Matassi, G. (1994) Codon usage and genome evolution. Curr. Opin. Genet., 6, Sharp, P.M., Tuohy, T.M.F. et Mosurski, K.R. (1986) Codon usage in yeast : cluster analysis clearly differentiates highly and lowly expressed genes. Nucleic Acids Res., 14, Shen, P. et Huang, H.V. (1989) Effect of base pair mismatches on recombination via the RecBCD pathway. Mol. Gen. Genet., 218, Shields, D.C. et Sharp, P.M. (1987) Synonymous codon usage in Bacillus subtilis reflects both translational selection and mutational biases. Nucleic Acids Res., 15, Shields, D.C., Sharp, P.M., Higgins, D.G. et Wright, F. (1988) Silent sites in Drosophila genes are not neutral : evidence of selection among synonymous codons. Mol. Biol. Evol., 5, Smith, D.R., Doucette-Stamm, L.A., Deloughery, C., Lee, H.-M., Dubois, J., Aldredge, T., Bashirzadeh, R., Blakely, D., Cook, R., Gilbert, K. et al. (1997) Complete genome sequence of Methanobacterium thermoautotrophicum delta H : functional analysis and comparative genomics. J. Bacteriol., 179, Smith, T.F. et Waterman, M.S. (1981) Identification of common molecular subsequences. J. Mol. Biol., 147, Solovyev, V.V., Salamov, A.A. et Lawrence, C.B. (1994) Predicting internal exons by oligonucleotide composition and discriminant analysis of spliceable open reading frames. Nucleic Acids Res., 22, Sprinzl, M., Horn, C., Brown, M., Ioudovitch, A. et Steinberg, S. (1998) Compilation of trna sequences and sequences of trna genes. Nucleic Acids Res., 26, Stenico, M., Lloyd, A.T. et Sharp, P.M. (1994) Codon usage in Caenorhabditis elegans : delineation of translational selection and mutation biases. Nucleic Acids Res., 22, Stephens, R.S., Kalman, S., Lammel, C.J., Fan, J., Marathe, R., Aravind, L., Mitchell, W.P., Olinger, L., Tatusov, R.L., Zhao, Q. et al. (1998) Genome sequence of an obligate intracellular pathogen of humans : Chlamydia trachomatis. Science, 282, Sueoka, N. (1962) On the genetic basis of variation and heterogeneity of DNA base composition. Genetics, 48, T Tateno, Y., Miyazaki, S., Ota, M., Sugawara, H. et Gojobori, T. (2000) DNA Data Bank of Japan (DDBJ) in collaboration with mass sequencing teams. Nucleic Acids Res., 28,
90 RÉFÉRENCES BIBLIOGRAPHIQUES Tatusov, R.L., Galperin, M.Y., Natale, D.A. et Koonin, E.V. (2000) The COG database : a tool for genome-scale analysis of protein functions and evolution. Nucleic Acids Res., 28, Tettelin, H., Saunders, N.J., Heidelberg, J., Jeffries, A.C., Nelson, K.E., Eisen, J.A., Ketchum, K.A., Hood, D.W., Peden, J.F., Dodson, R.J. et al. (2000) Complete genome sequence of Neisseria meningitidis serogroup B strain MC58. Science, 287, Thieffry, D., Salgado, H., Huerta, A.M. et Collado-Vides, J. (1998) Prediction of transcriptional regulatory sites in the complete genome sequence of Escherichia coli K-12 Bioinformatics, 14, Thierry-Mieg, J., Thierry-Mieg, D. et Stein, L. (1999) ACeDB : the ace database manager. In Bioinformatics Databases and Systems, Letovsky, S. (ed.), Kluwer Academic Publishers, Boston, pp Thioulouse, J. et Chevenet, F. (1996) NetMul, a World-Wide Web user interface for multivariate analysis sofware. Comput. Stat. Data Anal., 21, Thioulouse, J., Chessel, D., Dolédec, S. et Olivier, J.M. (1997) ADE-4 : a multivariate analysis and graphical display software. Stat. Comput., 7, Thompson, J.D., Higgins, D.G. et Gibson, T.J. (1994) CLUSTAL W : improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice. Nucleic Acids Res., 22, Tomb, J.F., White, O., Kerlavage, A.R., Clayton, R.A., Sutton, G.G., Fleischmann, R.D., Ketchum, K.A., Klenk, H.P., Gill, S., Dougherty, B.A. et al. (1997) The complete genome sequence of the gastric pathogen Helicobacter pylori. Nature, 388, V-Y Van de Peer, Y., De Rijk, P., Wuyts, J., Winkelmans, T. et De Wachter, R. (2000) The European small subunit ribosomal RNA database. Nucleic Acids Res., 28, Venter, J.C. (1991) Complementary DNA sequencing : expressed sequence tags and human genome project. Science, 252, Wahl, R. et Kroeger, M. (1995) ECDC a totally integrated and interactively usable genetic map of Escherichia coli K12. Microbiol. Res., 150, Wang, Y., Addess, K.J., Geer, L., Madej, T., Marchier-Bauer, A., Zimmerman, D. et Bryant, S.H. (2000) MMDB : 3D structure data in Entrez. Nucleic Acids Res., 28, White, O., Eisen, J.A., Heidelberg, J.F., Hickey, E.K., Peterson, J.D., Dodson, R.J., Haft, D.H., Gwinn, M.L., Nelson, W.C., Richardson, D.L. et al. (1999) Genome sequence of the radioresistant bacterium Deinococcus radiodurans R1. Science, 286, Wootton, J.C. et Federhen, S. (1996) Analysis of compositionally biased regions in sequence databases. Methods Enzymol., 266, Wu, C.H., Shivakumar, S., Shivakumar, C.V. et Chen, S. (1998) GeneFIND web server for protein family identification and information retrieval. Bioinformatics, 14,
91 RÉFÉRENCES BIBLIOGRAPHIQUES Yoccoz, G. (1988) Le Rôle du Modèle Euclidien d Analyse des Données en Biologie Évolutive. Thèse de Doctorat, Université Claude Bernard Lyon 1, Lyon. Yona, G., Linial, N. et Linial, M. (2000) ProtoMap : automatic classification of protein sequences and hierarchy of protein families. Nucleic Acids Res., 28,
92 TITRES ET TRAVAUX
93 TITRES ET TRAVAUX ANNEXE : TITRES ET TRAVAUX ARTICLES PUBLIÉS DANS DES REVUES INTERNATIONALES Cortay, J.-C., Nègre, D., Galinier, A., Duclos, B., Perrière, G. et Cozzone, A.J. (1991) Regulation of the acetate operon in Escherichia coli : purification and functional characterization of the IclR repressor. EMBO J., 10, Galinier, A., Bleicher, F., Nègre, D., Perrière, G., Duclos, B., Cozzone, A.J. et Cortay J.-C. (1991) Primary structure of the intergenic region between acek and iclr in the Escherichia coli chromosome. Gene, 97, Perrière, G. et Gautier, C. (1993) ColiGene : object-centered representation for the study of E. coli gene expressivity by sequence analysis. Biochimie, 75, Perrière, G., Gouy, M. et Gojobori, T. (1994) NRSub : a non-redundant data base for the Bacillus subtilis genome. Nucleic Acids Res., 22, Perrière, G., Moszer, I. et Gojobori, T. (1996) NRSub : a non-redundant database for Bacillus subtilis. Nucleic Acids Res., 24, Perrière, G. et Thioulouse, J. (1996) On-line tools for sequence retrieval and multivariate statistics in molecular biology. Comput. Applic. Biosci., 12, Perrière, G. et Gouy, M. (1996) WWW-Query : an on-line retrieval system for biological sequence banks. Biochimie, 78, Perrière, G., Lobry, J.R. et Thioulouse, J. (1996) Correspondence discriminant analysis : a multivariate method for comparing classes of protein and nucleic acids sequences. Comput. Applic. Biosci., 12, Duret, L., Gasteiger, E. et Perrière, G. (1996) LALNVIEW : a graphical viewer for pairwise sequence alignment. Comput. Applic. Biosci., 12, Perrière, G., Moszer, I. et Gojobori, T. (1997) The NRSub database : update Nucleic Acids Res., 25, Perrière, G., Gouy, M. et Gojobori, T. (1998) The non-redundant Bacillus subtilis (NRSub) database : update Nucleic Acids Res., 26, Perrière, G., Labedan, B. et Bessières, P. (1999) EMGLib : the enhanced microbial genomes library. Nucleic Acids Res., 27, Gonçalves, I., Robinson, M., Perrière, G. et Mouchiroud, D. (1999) JaDis : computing distances between nucleic acid sequences. Bioinformatics, 15, Perrière, G., Labedan, B. et Bessières, P. (2000) EMGLib : the Enhanced Microbial Genomes Library (update 2000). Nucleic Acids Res., 28, Perrière, G., Duret, L. et Gouy, M. (2000) HOBACGEN : database system for comparative genomics in bacteria. Genome Res., 10,
94 TITRES ET TRAVAUX Perrière, G. et Thioulouse, J. (2000) Use of correspondence discriminant analysis to predict the subcellular location of bacterial proteins. J. Comput. Biol., 7, sous presse. AUTRES PUBLICATIONS Perrière, G. (1991) ColiGene. In Les Cahiers IMABIO, Vol. 2, CNRS, Paris, pp Dorkeld, F., Perrière, G. et Gautier, C. (1993) Object-oriented modelling in molecular biology. In Proceedings of the 13 th IJCAI Workshop on Artificial Intelligence and Genome, Ganascia, J.-G. (ed.), IJCAI, pp Médigue, C., Willamowski, J., Schmeltzer, O., Uvietta, P., Rechenmann, F., Chevenet, F., Perrière, G. et Gautier, C. (1993) Modelling tasks for problem solving in molecular biology. In Proceedings of the 13 th IJCAI Workshop on Artificial Intelligence and Genome, Ganascia, J.-G. (ed.), IJCAI, pp Perrière, G., Dorkeld, F., Rechenmann, F. et Gautier, C. (1993) Object-oriented knowledge bases for the analysis of prokaryotic and eukaryotic genomes. In Proceedings of the 1 st International Conference on Intelligent Systems for Molecular Biology. Hunter, L., Searls, D. et Shavlik, J. (eds.), AAAI/MIT Press, Menlo Park, pp Schmeltzer, O., Médigue, C., Uvietta, P., Rechenmann, F., Dorkeld, F., Perrière, G. et Gautier, C. (1993) Building large knowledge bases in molecular biology. In Proceedings of the 1 st International Conference on Intelligent Systems for Molecular Biology, Hunter, L., Searls, D. et Shavlik, J. (eds.) AAAI/MIT Press, Menlo Park, pp Perrière, G., Chevenet, F., Dorkeld, F., Vermat, T. et Gautier, C. (1994) Building integrated systems for data representation and analysis in molecular biology. In Proceedings of the 27 th Hawaï International Conference on Systems Science, Vol. 5, Hunter, L. (ed.), IEEE/ACM, pp Perrière, G. (1995) NRSub : a non-redundant Bacillus subtilis data base. DDBJ Newslett., 15, Perrière, G. et Gautier, C. (1995) ColiGene exemple d une base de connaissances centrée-objet pour l étude de l expressivité des gènes de E. coli. In Rapport de Recherche INRIA nº 2530, Pavé, A. et Gouzé, J.-L. (eds.), INRIA, Sophia-Antipolis, pp Perrière, G. et Lobry, J.R. (1998) Asymmetrical coding sequence repartition and codon adaptation index values between leading and lagging strand in seven bacterial species. In Proceedings of the 1 st International Conference on Bioinformatics of Genome Regulation and Structure, Vol. 2, Kolchanov, N. (ed.), CIG, Novosibirsk, pp Duret, L., Perrière, G. et Gouy, M. (1999) HOVERGEN : database and software for comparative analysis of homologous vertebrate genes. In Bioinformatics Databases and Systems, Letovsky, S. (ed.), Kluwer Academic Publishers, Boston, pp Perrière, G. et Duret, L. (1999) HOBACGEN : a database of homologous genes in bacteria. In Proceedings of the German Conference on Bioinformatics, Giegerich, R. (ed.), Université de Bielefield, Bielefield, pp
95 TITRES ET TRAVAUX COLLOQUES INTERNATIONAUX Gautier, C., Perrière, G., Dorkeld, F., Jacobzone, M., Rechenmann, F. et Willamowski, J. (1991) Genomic sequence analysis and management using knowledge representation based upon objects. The Role of Biocomputing in the Characterization of Human Genome Sequences. Bari, octobre. Dorkeld, F., Perrière, G. et Gautier, C. (1993) Object-oriented modelling in molecular biology. 13 th IJCAI Workshop on Artificial Intelligence and Genome, Chambery, août. Médigue, C., Willamowski, J., Schmeltzer, O., Uvietta, P., Rechenmann, F., Chevenet, F., Perrière, G. et Gautier, C. (1993) Modelling tasks for problem solving in molecular biology. 13 th IJCAI Workshop on Artificial Intelligence and Genome, Chambery, août. Perrière, G. (1994) ColiGene : an object-oriented knowledge base for the representation of E. coli genome data. 3 rd International E. coli Genome Meeting. Woods Hole, 4-8 novembre. Borodovsky, M., Hayes, W., McIninch, J., Perrière, G., Moszer, I. et Danchin, A. (1995) Clustering of B. subtilis gene sequences with regard to their oligonucleotide composition. 8 th International Conference on Bacilli, Stanford, 8-12 juillet. Perrière, G. et Lobry, J.R. (1995) Comparison of codon and amino-acid usage between Escherichia coli and Bacillus subtilis. Jacques Monod Conference on Evolutionary Genetics and Adaptation, Aussois, septembre. Perrière, G. (1997) Use of Correspondence Discriminant Analysis to predict the subcellular location of bacterial proteins. Mathematics Applied to Biological Sequences, Rouen, août. Perrière, G. et Lobry, J.R. (1998) Asymmetrical coding sequence repartition and codon adaptation index values between leading and lagging strand in seven bacterial species. 1 st International Conference on Bioinformatics of Genome Regulation and Structure, Novosibirsk, août. Perrière, G. et Duret, L. (1999) HOBACGEN : a database of homologous genes in bacteria. German Conference on Bioinformatics, Hanovre, 4-6 octobre. COLLOQUES FRANÇAIS Gautier, C. et Perrière, G. (1989) Les banques de séquences génomiques : un outil de modélisation du génome. Rencontres Biologie Moléculaire et Informatique, Paris, 8-9 juin. Perrière, G. (1991) ColiGene. École IMABIO Traitement de l Information des Séquences Biologiques, Paris, 2-3 avril. Duret, L. et Perrière, G. (1997) HOBACGEN : banque de données de gènes homologues de bactéries. 3 ème Rencontre Alphy, Villeurbanne, mai. Perrière, G., Duret, L. et Gouy, M. (1999) HOBACGEN : une banque de données de familles de gènes chez les bactéries. 6 ème Rencontre Alphy, Villeurbanne, février
96 TITRES ET TRAVAUX Lefèbvre, I., Perrière, G. et Thioulouse, J. (1999) Les pièges de l analyse factorielle des correspondances dans l étude des séquences. Journées de la Société Française de Biométrie, Grenoble, mai. Perrière, G. (1999) HOBACGEN, une banque de données de familles de gènes chez les bactéries. Colloque Annuel de la Société Française de Génétique, Toulouse, avril. Guindon, S. et Perrière, G. (2000) Détection des transferts horizontaux : la variation intra-génomique de composition en bases est une source de biais. Journées Ouvertes : Biologie, Informatique et Mathématiques. Montpellier, 3-5 mai. POSTERS Perrière G., Willamowski J., Rechenmann F., Jacobzone M. et Gautier C. (1991) Knowledge representation issues in molecular biology. Bioinformatics in the 90 s. Maastricht, novembre. Perrière G., Dorkeld F., Rechenmann F. et Gautier C. (1993) Object-oriented knowledge bases for the analysis of prokaryotic and eukaryotic genomes. 1 st International Conference on Intelligent Systems for Molecular Biology. Washington, 6-9 juillet. Spataro, B., Duret, L. et Perrière, G. (1998) Comparative genomics in mammals and bacteria using Java software. Objects in Bioinformatics 1998 Conference, Hinxton- Cambridge, 3-4 août. Perrière, G. et Duret, L. (1999) Database systems for bacterial genomics. 3 rd International Conference on Computational Molecular Biology, Lyon, avril. Perrière, G., Blanchet, C., Duret, L., Geourjon, C., Thioulouse, J., Combet, C., Gouy, M. et Deléage, G. (1999) The Bioinformatics Pole of Lyon. 3 rd International Conference on Computational Molecular Biology, Lyon avril. Lafaye, L., Gonçalves, I., Robinson, M. et Perrière, G. (1999) Java applications for molecular evolution and phylogenetic studies. 3 rd International Conference on Computational Molecular Biology, Lyon, avril. THÈSE DE DOCTORAT Application d une représentation par objets des connaissances à la modélisation de certains aspects de l expression des gènes chez Escherichia coli. Laboratoire de Biométrie, Génétique et Biologie des Populations, Université Claude Bernard Lyon 1, Lyon, 7 octobre Mention Très Honorable. SÉMINAIRES Utilisation d une base de connaissances centrée-objet pour l étude de l expressivité des gènes chez Escherichia coli. Laboratoire de Biométrie, Génétique et Biologie des Populations, Université Claude Bernard Lyon 1, Lyon, 3 décembre
97 TITRES ET TRAVAUX Données sur les opérons d E. coli intégrées dans ColiGene. Laboratoire de Biométrie, Génétique et Biologie des Populations, Université Claude Bernard Lyon 1, Lyon, 19 décembre ColiGene, une base de connaissances sur le génome d E. coli. Département de Biochimie Médicale, Université de Genève, Genève, 29 octobre Object-oriented systems for data representation and analysis in molecular biology. DNA Research Center, National Institute of Genetics, Mishima, 10 avril Heterogeneous biomolecular databases. DNA Research Center, National Institute of Genetics, Mishima, 9 septembre Object-oriented databases for the modeling of prokaryotic and eukaryotic genomes. Université d Hiroshima, 20 septembre Object-oriented modelling for biological sequences knowledge bases. Université de Tokyo, 6 octobre Recherche des parties codantes dans l ADN des procaryotes. Forum de l Institut d Analyse des Systèmes Biologiques et Socio-Économiques, Université Claude Bernard Lyon 1, Lyon, 19 mai Bases de données et projets de séquençage de génomes : le cas de Bacillus subtilis. Laboratoire de Biométrie, Génétique et Biologie des Populations, Université Claude Bernard Lyon 1, Lyon, 22 mai Informatique et génomes : le rôle grandissant d Internet. Journées du Laboratoire de Biométrie, Génétique et Biologie des Populations, Villebois, 15 avril Building non-redundant databases in the context of bacterial genome sequencing projects. Glaxo Institute for Molecular Biology, Genève, 30 août HOBACGEN : une dase de données pour l analyse comparative de gènes homologues bactériens. Département de Microbiologie, Centre INRA de Jouy-en-Josas, 20 janvier La génomique comparative : un outil indispensable du post-séquençage. Centre de Génétique Moléculaire et Cellulaire, Université Claude Bernard Lyon 1, Lyon, 2 avril La banque de données de gènes homologues de bactéries HOBACGEN. Muséum d Histoire Naturelle, Paris, 25 juin ORGANISATION DE COLLOQUES ET DE SÉMINAIRES Depuis le mois d octobre 1997, je suis en charge de l organisation des séminaires du Laboratoire de Biométrie et Biologie Évolutive. À cette responsabilité est venue s ajouter, en janvier 1999, celle de co-organiser les séminaires de l IFR 41 «Sciences et Méthodes de l Écologie et de l Évolution». À ces deux postes, j essaie d équilibrer les interventions en fonction des différentes thématiques représentées dans notre laboratoire: biométrie, génétique et biologie des populations, évolution, phylogénie moléculaire et bioinformatique. À ce jour, 31 invités extérieurs et 22 invités appartenant à l Université Claude Bernard Lyon 1 (UCBL) ont participé à ces séminaires. En 1999, j ai également été responsable de l organisation du sixième colloque Alphy (Alignements et phylogénie) qui s est tenu à Lyon les 11 et 12 février. Près de 70 personnes ont assisté à cette réunion qui faisait le bilan de nombreux travaux réalisés en France sur les génomes bactériens. Parmi les thèmes abordés au cours de ce
98 TITRES ET TRAVAUX colloque figuraient : les asymétries dans la structure des chromosomes, les banques de données, la phylogénie et la construction de familles de gènes ou de domaines homologues. PARTICIPATION À DES CONTRATS Depuis 1994 j ai participé à six contrats déposés sur des projets de recherche en (ou incluant une partie) bioinformatique dont un en tant que coordonnateur scientifique («Développement, diffusion et exploitation d une base de données dédiée à différents aspects de la génomique bactérienne») : Structures secondaires de protéines et banques de données de séquences. Ministère de l Éducation Nationale et de la Recherche, ACC-SV n 13 «Bioinformatique». Coordonnateur : Gilbert Deléage (Institut de Biologie et de Chimie des Protéines, UPR CNRS n 412) : Distribution du système ACNUC d interrogation des banques de séquences et de VGDB, une base de gènes homologues alignés de vertébrés. Ministère de l Éducation Nationale et de la Recherche, Groupement de Recherches et d Études sur les Génomes. Coordonnateur : Manolo Gouy (Laboratoire de Biométrie et Biologie Évolutive, UMR CNRS n 5558) : Bioinformatique des interactions moléculaires et des réseaux régulateurs à l échelle des génomes. Ministère de l Éducation Nationale et de la Recherche. Coordonnateur : Bernard Jacq (Laboratoire de Génétique et Physiologie du Développement, UMR CNRS n 6545) : Relations séquence-structure des protéines, entre gènes et fonctions. Ministère de l Éducation Nationale et de la Recherche, Programme Génome. Cordonnateur : Jacques Chomilier (Systèmes Moléculaires et Biologie Structurale, Laboratoire de Minéralogie Cristallographie, URA CNRS n 09) : Développement, diffusion et exploitation d une base de données dédiée à différents aspects de la génomique bactérienne. INSERM, Programme de Recherche Fondamentale en Microbiologie, Maladies Infectieuses et Parasitaires : Analyse génomique de Wolbachia, bactéries endocytobiotiques d arthropodes. Ministère de l Éducation Nationale et de la Recherche, Soutien Scientifique de Projets de Recherches en Sciences du Vivant au Sein des IFR. Coordonnateur : Pascal Simonet (Laboratoire d Écologie Microbienne, UMR CNRS n 5557). ENCADREMENT TROISIÈME CYCLE Alain Garreau (Diplôme d Ingénieur CNAM Informatique, Grenoble) Étude et réalisation d une interface cartographique d aide à l analyse des séquences génomiques, (membre du jury). Thomas Gueret (DEA Analyse et Modélisation de Systèmes Biologiques, Lyon) Recherche de corrélations entre données globales et de terrain sur des bassins versants de rivières, (parrain)
99 TITRES ET TRAVAUX Elie Debreuve (DEA Analyse et Modélisation de Systèmes Biologiques, Lyon) Usage des données de traits biologiques en écologie, (parrain). Stéphane Guindon (DEA Analyse et Modélisation de Systèmes Biologiques, Lyon) Conception et validation de méthodes de détection de transferts horizontaux de matériel génétique, (encadrant principal). Vincent Daubin (Thèse de l École Doctorale Évolution, Écosystèmes, Microbiologie et Modélisation, Lyon) Recherche de gènes obtenus par transfert horizontal chez les bactéries pathogènes de l Homme, (co-encadrant). STAGES DE SECOND CYCLE Laurent Lafaye (Licence et Maitrise) Programme de visualisation et de manipulation d arbres phylogénétiques, (stage TER). Jihène Serkhane (Maitrise) Interface client/serveur de consultation et d interrogation des banques de séquences, (stage TER). Khamlorn Chanou (Maitrise) Création d une interface Java au programme de phylogénie moléculaire PHYLO_LINE, (stage TER). ENSEIGNEMENT Ma double compétence informatique et biologie moléculaire m a permis d intervenir dans des enseignements se situant à l interface de ces deux disciplines. Depuis 1996 j interviens régulièrement dans différents enseignements et formations en bioinformatique. TP ET TD Depuis j organise en collaboration avec Laurent Duret des TP et TD d analyse des séquences dans le cadre du DEA Différenciation, Génétique et Immunologie de l UCBL à raison de 33h/an. Parmi les sujets abordés dans cet enseignement figurent : interrogation de banques de séquences, utilisation des logiciels de prédiction de régions fonctionnelles, recherches de similarités, alignements, construction d arbres phylogénétiques, utilisation des outils disponibles sur Internet. Par ailleurs, je suis également intervenu dans diverses formations et stages pratiques en bioinformatique : Cours EMBO «Analyse des Séquences et Évolution Moléculaire» (4h en 1995). Stage de formation continue BioMérieux (11h en 1997). Stage de formation continue ORSTOM (8h en 1998). COURS MAGISTRAUX Depuis 1996 j interviens, à raison de 5h/an, dans l UV de Biologie Évolutive de la maîtrise de Biologie des Populations et des Écosystèmes de l UCBL. Ce cours introduisant des notions sur les banques de séquences, les alignements simples et multiples, ainsi que l utilisation des statistiques multivariées pour l analyse des séquences. J ai également donné des cours dans les formations suivantes : Stage de formation continue ORSTOM (2h30 en 1998)
100 TITRES ET TRAVAUX École thématique du CNRS «Traitement de l Information en Génétique Moléculaire» (1h30 en 1998). DEA Écologie Microbienne (UCBL), «Biodiversité et Phylogénie» (2h en 1999). Certificat Physiopathologie des Maladies Transmissibles (UCBL), «Plasticité des Génomes Bactériens» (2h en 1999). DEA Bioinformatique de Genève (4h en 2000). COLLABORATIONS Internes au Laboratoire de Biométrie et Biologie Évolutive : Laurent Duret, Manolo Gouy, Jean Lobry, Jean Thioulouse. Externes (en France et à l étranger) : Philippe Bessières (Laboratoire de Génétique Microbienne, Institut National de la Recherche en Agronomie, Jouy-en-Josas). Gilbert Deléage et Christophe Geourjon (Institut de Biologie et de Chimie des Protéines, Lyon). Takashi Gojobori (National Institute of Genetics, Mishima, Japon). Bernard Labedan (Institut de Génétique et Microbiologie, Université Paris Sud, Orsay). Ivan Moszer (Unité de Régulation de l Expression Génétique, Institut Pasteur, Paris). Marc Robinson (Laboratoire de Biologie Moléculaire et Cellulaire, École Normale Supérieure de Lyon). STAGE POST-DOCTORAL Pendant un an, dans le cadre d une mise à disposition par le CNRS, j ai travaillé au National Institute of Genetics de Mishima (Japon), ceci dans le laboratoire dirigé par le Professeur Takashi Gojobori. Pendant cette année de stage post-doctoral j ai développé la banque NRSub, dédiée à B. subtilis, et j ai conçu le noyau de ce qui devait devenir le système d interrogation des banques figurant sur le serveur du PBIL. DIVERS Membre titulaire de la section 67 de la Commission de Spécialistes de l Enseignement Supérieur de l UCBL. Rapporteur régulier pour les revues Bioinformatics, Gene et Molecular Biology and Evolution
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST.
 La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
Introduction aux bases de données: application en biologie
 Introduction aux bases de données: application en biologie D. Puthier 1 1 ERM206/Technologies Avancées pour le Génome et la Clinique, http://tagc.univ-mrs.fr/staff/puthier, [email protected] ESIL,
Introduction aux bases de données: application en biologie D. Puthier 1 1 ERM206/Technologies Avancées pour le Génome et la Clinique, http://tagc.univ-mrs.fr/staff/puthier, [email protected] ESIL,
Formavie 2010. 2 Différentes versions du format PDB...3. 3 Les champs dans les fichiers PDB...4. 4 Le champ «ATOM»...5. 6 Limites du format PDB...
 Formavie 2010 Les fichiers PDB Les fichiers PDB contiennent les informations qui vont permettre à des logiciels de visualisation moléculaire (ex : RasTop ou Jmol) d afficher les molécules. Un fichier au
Formavie 2010 Les fichiers PDB Les fichiers PDB contiennent les informations qui vont permettre à des logiciels de visualisation moléculaire (ex : RasTop ou Jmol) d afficher les molécules. Un fichier au
Module Analyse de Génomes 2011-2012 Master 2 module FMBS 326 Immunoinformatique
 Module Analyse de Génomes 2011-2012 Master 2 module FMBS 326 Immunoinformatique Planning du Module : Date Heure Salle 12/12 9h-12h TD info TA1Z bat 25 13h-17h TD info TA1Z bat 25 13/12 9h-12h TD info TA1Z
Module Analyse de Génomes 2011-2012 Master 2 module FMBS 326 Immunoinformatique Planning du Module : Date Heure Salle 12/12 9h-12h TD info TA1Z bat 25 13h-17h TD info TA1Z bat 25 13/12 9h-12h TD info TA1Z
Extraction d information des bases de séquences biologiques avec R
 Extraction d information des bases de séquences biologiques avec R 21 novembre 2006 Résumé Le module seqinr fournit des fonctions pour extraire et manipuler des séquences d intérêt (nucléotidiques et protéiques)
Extraction d information des bases de séquences biologiques avec R 21 novembre 2006 Résumé Le module seqinr fournit des fonctions pour extraire et manipuler des séquences d intérêt (nucléotidiques et protéiques)
Base de données bibliographiques Pubmed-Medline
 Chapitre 1 ; Domaine 1 ; Documentation ; Champs référentiels 1.1.1, 1.1.2 et 1.1.3 Base de données bibliographiques Pubmed-Medline D r Patrick Deschamps,, 30 mai 2007 PLAN C2i métiers de la santé Introduction
Chapitre 1 ; Domaine 1 ; Documentation ; Champs référentiels 1.1.1, 1.1.2 et 1.1.3 Base de données bibliographiques Pubmed-Medline D r Patrick Deschamps,, 30 mai 2007 PLAN C2i métiers de la santé Introduction
Big data et sciences du Vivant L'exemple du séquençage haut débit
 Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard [email protected] INRA - MIAT - Plate-forme
Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard [email protected] INRA - MIAT - Plate-forme
Bibliographie Introduction à la bioinformatique
 Bibliographie Introduction à la bioinformatique 5. Les bases de données biologiques, SQL et la programmation Python/C++ Zvelebil et Baum, Understanding bioinformatics Beighley, Head First SQL Chari, A
Bibliographie Introduction à la bioinformatique 5. Les bases de données biologiques, SQL et la programmation Python/C++ Zvelebil et Baum, Understanding bioinformatics Beighley, Head First SQL Chari, A
MABioVis. Bio-informatique et la
 MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
! Séquence et structure des macromolécules. " Séquences protéiques (UniProt) " Séquences nucléotidiques (EMBL / ENA, Genbank, DDBJ)
 Introduction à la Bioinformatique Introduction! Les bases de données jouent un rôle crucial dans l organisation des connaissances biologiques.! Nous proposons ici un tour rapide des principales bases de
Introduction à la Bioinformatique Introduction! Les bases de données jouent un rôle crucial dans l organisation des connaissances biologiques.! Nous proposons ici un tour rapide des principales bases de
CHAPITRE 3 LA SYNTHESE DES PROTEINES
 CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
Semestre 2 Spécialité «Analyse in silico des complexes macromolécules biologiques-médicaments»
 Master In silico Drug Design Semestre 2 Spécialité «Analyse in silico des complexes macromolécules biologiques-médicaments» 30NU01IS INITIATION A LA PROGRAMMATION (6 ECTS) Responsables : D. MESTIVIER,
Master In silico Drug Design Semestre 2 Spécialité «Analyse in silico des complexes macromolécules biologiques-médicaments» 30NU01IS INITIATION A LA PROGRAMMATION (6 ECTS) Responsables : D. MESTIVIER,
GMIN206 Info. Biologique et Outils bioinformatiques. Elodie Cassan
 M Bioinformatique, Connaissances et Données Année 24-25 GMIN206 Info. Biologique et Outils bioinformatiques Banques de données biologiques (3h de Cours +,5h de TD + 4h de TP) Elodie Cassan Anne-Muriel
M Bioinformatique, Connaissances et Données Année 24-25 GMIN206 Info. Biologique et Outils bioinformatiques Banques de données biologiques (3h de Cours +,5h de TD + 4h de TP) Elodie Cassan Anne-Muriel
IMMUNOLOGIE. La spécificité des immunoglobulines et des récepteurs T. Informations scientifiques
 IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
Identification de nouveaux membres dans des familles d'interleukines
 Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
Incertitude et variabilité : la nécessité de les intégrer dans les modèles
 Incertitude et variabilité : la nécessité de les intégrer dans les modèles M. L. Delignette-Muller Laboratoire de Biométrie et Biologie Evolutive VetAgro Sup - Université de Lyon - CNRS UMR 5558 24 novembre
Incertitude et variabilité : la nécessité de les intégrer dans les modèles M. L. Delignette-Muller Laboratoire de Biométrie et Biologie Evolutive VetAgro Sup - Université de Lyon - CNRS UMR 5558 24 novembre
L utilisation d un réseau de neurones pour optimiser la gestion d un firewall
 L utilisation d un réseau de neurones pour optimiser la gestion d un firewall Réza Assadi et Karim Khattar École Polytechnique de Montréal Le 1 mai 2002 Résumé Les réseaux de neurones sont utilisés dans
L utilisation d un réseau de neurones pour optimiser la gestion d un firewall Réza Assadi et Karim Khattar École Polytechnique de Montréal Le 1 mai 2002 Résumé Les réseaux de neurones sont utilisés dans
7. Recherche des essais
 7. Recherche des essais Le chapitre précédent a insisté sur la nécessité de réaliser une recherche des essais aussi exhaustive que possible. Seule la conjonction de tous les moyens disponibles peut laisser
7. Recherche des essais Le chapitre précédent a insisté sur la nécessité de réaliser une recherche des essais aussi exhaustive que possible. Seule la conjonction de tous les moyens disponibles peut laisser
Perl Orienté Objet BioPerl There is more than one way to do it
 Perl Orienté Objet BioPerl There is more than one way to do it Bérénice Batut, [email protected] DUT Génie Biologique Option Bioinformatique Année 2014-2015 Perl Orienté Objet - BioPerl Rappels
Perl Orienté Objet BioPerl There is more than one way to do it Bérénice Batut, [email protected] DUT Génie Biologique Option Bioinformatique Année 2014-2015 Perl Orienté Objet - BioPerl Rappels
Analyse des données de séquençage massif par des méthodes phylogénétiques
 Analyse des données de séquençage massif par des méthodes phylogénétiques Roux S., Taib N., Mangot J.F., Hugoni M., Mary I., Ravet V., Bronner G., Enault F., Debroas D. Équipe Microbiologie de l'environnement
Analyse des données de séquençage massif par des méthodes phylogénétiques Roux S., Taib N., Mangot J.F., Hugoni M., Mary I., Ravet V., Bronner G., Enault F., Debroas D. Équipe Microbiologie de l'environnement
La reconnaissance moléculaire: la base du design rationnel Modélisation moléculaire: Introduction Hiver 2006
 La reconnaissance moléculaire: la base du design rationnel En 1890 Emil Fisher a proposé le modèle "serrure et clé" pour expliquer la façon de fonctionner des systèmes biologiques. Un substrat rentre et
La reconnaissance moléculaire: la base du design rationnel En 1890 Emil Fisher a proposé le modèle "serrure et clé" pour expliquer la façon de fonctionner des systèmes biologiques. Un substrat rentre et
Chapitre 1 : Introduction aux bases de données
 Chapitre 1 : Introduction aux bases de données Les Bases de Données occupent aujourd'hui une place de plus en plus importante dans les systèmes informatiques. Les Systèmes de Gestion de Bases de Données
Chapitre 1 : Introduction aux bases de données Les Bases de Données occupent aujourd'hui une place de plus en plus importante dans les systèmes informatiques. Les Systèmes de Gestion de Bases de Données
E-Biothon : Une plate-forme pour accélérer les recherches en biologie, santé et environnement.
 E-Biothon : Une plate-forme pour accélérer les recherches en biologie, santé et environnement. N.Bard, S.Boin, F.Bothorel, P.Collinet, M.Daydé, B. Depardon, F. Desprez, M.Flé, A.Franc, J.-F. Gibrat, D.
E-Biothon : Une plate-forme pour accélérer les recherches en biologie, santé et environnement. N.Bard, S.Boin, F.Bothorel, P.Collinet, M.Daydé, B. Depardon, F. Desprez, M.Flé, A.Franc, J.-F. Gibrat, D.
Dr E. CHEVRET UE2.1 2013-2014. Aperçu général sur l architecture et les fonctions cellulaires
 Aperçu général sur l architecture et les fonctions cellulaires I. Introduction II. Les microscopes 1. Le microscope optique 2. Le microscope à fluorescence 3. Le microscope confocal 4. Le microscope électronique
Aperçu général sur l architecture et les fonctions cellulaires I. Introduction II. Les microscopes 1. Le microscope optique 2. Le microscope à fluorescence 3. Le microscope confocal 4. Le microscope électronique
MASTER (LMD) PARCOURS MICROORGANISMES, HÔTES, ENVIRONNEMENTS (MHE)
 MASTER (LMD) PARCOURS MICROORGANISMES, HÔTES, ENVIRONNEMENTS (MHE) RÉSUMÉ DE LA FORMATION Type de diplôme : Master (LMD) Domaine ministériel : Sciences, Technologies, Santé Mention : BIOLOGIE DES PLANTES
MASTER (LMD) PARCOURS MICROORGANISMES, HÔTES, ENVIRONNEMENTS (MHE) RÉSUMÉ DE LA FORMATION Type de diplôme : Master (LMD) Domaine ministériel : Sciences, Technologies, Santé Mention : BIOLOGIE DES PLANTES
WHITE PAPER. Quels avantages la déduplication offre-t-elle aux entreprises? Livre blanc Acronis
 Quels avantages la déduplication offre-t-elle aux entreprises? Livre blanc Acronis Copyright Acronis, Inc. 2000 2009 Table des matières Résumé... 3 Qu est-ce que la déduplication?... 4 Déduplication au
Quels avantages la déduplication offre-t-elle aux entreprises? Livre blanc Acronis Copyright Acronis, Inc. 2000 2009 Table des matières Résumé... 3 Qu est-ce que la déduplication?... 4 Déduplication au
Introduction au Data-Mining
 Introduction au Data-Mining Alain Rakotomamonjy - Gilles Gasso. INSA Rouen -Département ASI Laboratoire PSI Introduction au Data-Mining p. 1/25 Data-Mining : Kèkecé? Traduction : Fouille de données. Terme
Introduction au Data-Mining Alain Rakotomamonjy - Gilles Gasso. INSA Rouen -Département ASI Laboratoire PSI Introduction au Data-Mining p. 1/25 Data-Mining : Kèkecé? Traduction : Fouille de données. Terme
INF6304 Interfaces Intelligentes
 INF6304 Interfaces Intelligentes filtres collaboratifs 1/42 INF6304 Interfaces Intelligentes Systèmes de recommandations, Approches filtres collaboratifs Michel C. Desmarais Génie informatique et génie
INF6304 Interfaces Intelligentes filtres collaboratifs 1/42 INF6304 Interfaces Intelligentes Systèmes de recommandations, Approches filtres collaboratifs Michel C. Desmarais Génie informatique et génie
Résonance Magnétique Nucléaire : RMN
 21 Résonance Magnétique Nucléaire : RMN Salle de TP de Génie Analytique Ce document résume les principaux aspects de la RMN nécessaires à la réalisation des TP de Génie Analytique de 2ème année d IUT de
21 Résonance Magnétique Nucléaire : RMN Salle de TP de Génie Analytique Ce document résume les principaux aspects de la RMN nécessaires à la réalisation des TP de Génie Analytique de 2ème année d IUT de
LE CHEMINEMENT COMPLEXE D UN VACCIN
 LE CHEMINEMENT COMPLEXE D UN VACCIN Chaîne de production, exigences réglementaires et accès aux vaccins International Federation of Pharmaceutical Manufacturers & Associations LE CHEMINEMENT COMPLEXE D
LE CHEMINEMENT COMPLEXE D UN VACCIN Chaîne de production, exigences réglementaires et accès aux vaccins International Federation of Pharmaceutical Manufacturers & Associations LE CHEMINEMENT COMPLEXE D
e-biogenouest CNRS UMR 6074 IRISA-INRIA / Plateforme de Bioinformatique GenOuest [email protected] Programme fédérateur Biogenouest co-financé
 e-biogenouest Coordinateur : Olivier Collin Animateur : Yvan Le Bras CNRS UMR 6074 IRISA-INRIA / Plateforme de Bioinformatique GenOuest [email protected] Programme fédérateur Biogenouest co-financé
e-biogenouest Coordinateur : Olivier Collin Animateur : Yvan Le Bras CNRS UMR 6074 IRISA-INRIA / Plateforme de Bioinformatique GenOuest [email protected] Programme fédérateur Biogenouest co-financé
INTERSYSTEMS CACHÉ COMME ALTERNATIVE AUX BASES DE DONNÉES RÉSIDENTES EN MÉMOIRE
 I N T E RS Y S T E M S INTERSYSTEMS CACHÉ COMME ALTERNATIVE AUX BASES DE DONNÉES RÉSIDENTES EN MÉMOIRE David Kaaret InterSystems Corporation INTERSySTEMS CAChé CoMME ALTERNATIvE AUx BASES de données RéSIdENTES
I N T E RS Y S T E M S INTERSYSTEMS CACHÉ COMME ALTERNATIVE AUX BASES DE DONNÉES RÉSIDENTES EN MÉMOIRE David Kaaret InterSystems Corporation INTERSySTEMS CAChé CoMME ALTERNATIvE AUx BASES de données RéSIdENTES
Domaine : Sciences, Technologies et Santé Mention : Nutrition, Sciences des aliments, Agroalimentaire
 Contexte Domaine : Sciences, Technologies et Santé Mention : Nutrition, Sciences des aliments, Agroalimentaire Fédération des spécialités de Master des 5 pôles universitaires partenaires de la région Nord-Pas-de-Calais
Contexte Domaine : Sciences, Technologies et Santé Mention : Nutrition, Sciences des aliments, Agroalimentaire Fédération des spécialités de Master des 5 pôles universitaires partenaires de la région Nord-Pas-de-Calais
CONSEILS POUR LA REDACTION DU RAPPORT DE RECHERCHE. Information importante : Ces conseils ne sont pas exhaustifs!
 CONSEILS POUR LA REDACTION DU RAPPORT DE RECHERCHE Information importante : Ces conseils ne sont pas exhaustifs! Conseils généraux : Entre 25 et 60 pages (hormis références, annexes, résumé) Format d un
CONSEILS POUR LA REDACTION DU RAPPORT DE RECHERCHE Information importante : Ces conseils ne sont pas exhaustifs! Conseils généraux : Entre 25 et 60 pages (hormis références, annexes, résumé) Format d un
Séquence 6. Mais ces espèces pour autant ne sont pas identiques et parfois d ailleurs ne se ressemblent pas vraiment.
 Sommaire Séquence 6 Nous avons vu dans les séances précédentes qu au cours des temps géologiques des espèces différentes se sont succédé, leur apparition et leur disparition étant le résultat de modifications
Sommaire Séquence 6 Nous avons vu dans les séances précédentes qu au cours des temps géologiques des espèces différentes se sont succédé, leur apparition et leur disparition étant le résultat de modifications
Introduction à la B.I. Avec SQL Server 2008
 Introduction à la B.I. Avec SQL Server 2008 Version 1.0 VALENTIN Pauline 2 Introduction à la B.I. avec SQL Server 2008 Sommaire 1 Présentation de la B.I. et SQL Server 2008... 3 1.1 Présentation rapide
Introduction à la B.I. Avec SQL Server 2008 Version 1.0 VALENTIN Pauline 2 Introduction à la B.I. avec SQL Server 2008 Sommaire 1 Présentation de la B.I. et SQL Server 2008... 3 1.1 Présentation rapide
Les OGM. 5 décembre 2008. Nicole Mounier
 Les OGM 5 décembre 2008 Nicole Mounier Université Claude Bernard Lyon 1 CGMC, bâtiment Gregor Mendel 43, boulevard du 11 Novembre 1918 69622 Villeurbanne Cedex OGM Organismes Génétiquement Modifiés Transfert
Les OGM 5 décembre 2008 Nicole Mounier Université Claude Bernard Lyon 1 CGMC, bâtiment Gregor Mendel 43, boulevard du 11 Novembre 1918 69622 Villeurbanne Cedex OGM Organismes Génétiquement Modifiés Transfert
INTRODUCTION À L ANALYSE FACTORIELLE DES CORRESPONDANCES
 INTRODUCTION À L ANALYSE FACTORIELLE DES CORRESPONDANCES Dominique LAFFLY Maître de Conférences, Université de Pau Laboratoire Société Environnement Territoire UMR 5603 du CNRS et Université de Pau Domaine
INTRODUCTION À L ANALYSE FACTORIELLE DES CORRESPONDANCES Dominique LAFFLY Maître de Conférences, Université de Pau Laboratoire Société Environnement Territoire UMR 5603 du CNRS et Université de Pau Domaine
Conférence technique internationale de la FAO
 Décembre 2009 ABDC-10/7.2 F Conférence technique internationale de la FAO Biotechnologies agricoles dans les pays en développement: choix et perspectives pour les cultures, les forêts, l élevage, les pêches
Décembre 2009 ABDC-10/7.2 F Conférence technique internationale de la FAO Biotechnologies agricoles dans les pays en développement: choix et perspectives pour les cultures, les forêts, l élevage, les pêches
INF 1250 INTRODUCTION AUX BASES DE DONNÉES. Guide d étude
 INF 1250 INTRODUCTION AUX BASES DE DONNÉES Guide d étude Sous la direction de Olga Mariño Télé-université Montréal (Québec) 2011 INF 1250 Introduction aux bases de données 2 INTRODUCTION Le Guide d étude
INF 1250 INTRODUCTION AUX BASES DE DONNÉES Guide d étude Sous la direction de Olga Mariño Télé-université Montréal (Québec) 2011 INF 1250 Introduction aux bases de données 2 INTRODUCTION Le Guide d étude
La gestion des correctifs de sécurité avec WinReporter et RemoteExec
 White Paper La gestion des correctifs de sécurité avec WinReporter et RemoteExec Ce document décrit les fonctionnalités de WinReporter et RemoteExec permettant de maintenir les systèmes Windows à jour
White Paper La gestion des correctifs de sécurité avec WinReporter et RemoteExec Ce document décrit les fonctionnalités de WinReporter et RemoteExec permettant de maintenir les systèmes Windows à jour
d évaluation Objectifs Processus d élaboration
 Présentation du Programme pancanadien d évaluation Le Programme pancanadien d évaluation (PPCE) représente le plus récent engagement du Conseil des ministres de l Éducation du Canada (CMEC) pour renseigner
Présentation du Programme pancanadien d évaluation Le Programme pancanadien d évaluation (PPCE) représente le plus récent engagement du Conseil des ministres de l Éducation du Canada (CMEC) pour renseigner
Mieux comprendre les certificats SSL THAWTE EST L UN DES PRINCIPAUX FOURNISSEURS DE CERTIFICATS SSL DANS LE MONDE
 Mieux comprendre les certificats SSL THAWTE EST L UN DES PRINCIPAUX FOURNISSEURS DE CERTIFICATS SSL DANS LE MONDE sommaire MIEUX COMPRENDRE LES CERTIFICATS SSL...1 SSL et certificats SSL : définition...1
Mieux comprendre les certificats SSL THAWTE EST L UN DES PRINCIPAUX FOURNISSEURS DE CERTIFICATS SSL DANS LE MONDE sommaire MIEUX COMPRENDRE LES CERTIFICATS SSL...1 SSL et certificats SSL : définition...1
D une part, elles ne peuvent faire table rase de la richesse contenue dans leur système d information.
 PACBASE «Interrogez le passé, il répondra présent.». Le Module e-business Les entreprises doivent aujourd hui relever un triple défi. D une part, elles ne peuvent faire table rase de la richesse contenue
PACBASE «Interrogez le passé, il répondra présent.». Le Module e-business Les entreprises doivent aujourd hui relever un triple défi. D une part, elles ne peuvent faire table rase de la richesse contenue
Nom de l application
 Ministère de l Enseignement Supérieur et de la Recherche Scientifique Direction Générale des Etudes Technologiques Institut Supérieur des Etudes Technologiques de Gafsa Département Technologies de l Informatique
Ministère de l Enseignement Supérieur et de la Recherche Scientifique Direction Générale des Etudes Technologiques Institut Supérieur des Etudes Technologiques de Gafsa Département Technologies de l Informatique
COMMENT LIRE UN DEVIS DE CREATION DE SITE WEB?
 COMMENT LIRE UN DEVIS DE CREATION DE SITE WEB? Lorraine En matière de création ou de refonte d un site Internet, il apparaît souvent difficile de faire un choix parmi les propositions qui font suite à
COMMENT LIRE UN DEVIS DE CREATION DE SITE WEB? Lorraine En matière de création ou de refonte d un site Internet, il apparaît souvent difficile de faire un choix parmi les propositions qui font suite à
Rédiger et administrer un questionnaire
 Rédiger et administrer un questionnaire Ce document constitue une adaptation, en traduction libre, de deux brochures distinctes : l une produite par l American Statistical Association (Designing a Questionnaire),
Rédiger et administrer un questionnaire Ce document constitue une adaptation, en traduction libre, de deux brochures distinctes : l une produite par l American Statistical Association (Designing a Questionnaire),
Base de données relationnelle et requêtes SQL
 Base de données relationnelle et requêtes SQL 1e partie Anne-Marie Cubat Une question pour commencer : que voyez-vous? Cela reste flou Les plans de «Prison Break»? Non, cherchons ailleurs! Et de plus près,
Base de données relationnelle et requêtes SQL 1e partie Anne-Marie Cubat Une question pour commencer : que voyez-vous? Cela reste flou Les plans de «Prison Break»? Non, cherchons ailleurs! Et de plus près,
Code d'éthique de la recherche
 Code d'éthique de la recherche Ce texte reprend le code d'éthique de la FPSE de l'université de Genève, avec une adaptation des références aux instances académiques. Cette version est plus particulièrement
Code d'éthique de la recherche Ce texte reprend le code d'éthique de la FPSE de l'université de Genève, avec une adaptation des références aux instances académiques. Cette version est plus particulièrement
Modernisation et gestion de portefeuilles d applications bancaires
 Modernisation et gestion de portefeuilles d applications bancaires Principaux défis et facteurs de réussite Dans le cadre de leurs plans stratégiques à long terme, les banques cherchent à tirer profit
Modernisation et gestion de portefeuilles d applications bancaires Principaux défis et facteurs de réussite Dans le cadre de leurs plans stratégiques à long terme, les banques cherchent à tirer profit
Analyses croisées de sites Web pour détecter les sites de contrefaçon. Prof. Dr. Olivier Biberstein
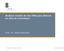 Analyses croisées de sites Web pour détecter les sites de contrefaçon Prof. Dr. Olivier Biberstein Division of Computer Science 14 Novembre 2013 Plan 1. Présentation générale 2. Projet 3. Travaux futurs
Analyses croisées de sites Web pour détecter les sites de contrefaçon Prof. Dr. Olivier Biberstein Division of Computer Science 14 Novembre 2013 Plan 1. Présentation générale 2. Projet 3. Travaux futurs
HighPush. document 3.0 18/06/2009 Révision pour version 3.0 2.0 20/11/2008 Revision pour la 2.0 1.0 01/10/2008 Documentation initiale.
 Version du Date document 3.0 18/06/2009 Révision pour version 3.0 2.0 20/11/2008 Revision pour la 2.0 1.0 01/10/2008 Documentation initiale Commentaires 1 Table des matières 1 Introduction / Identification...
Version du Date document 3.0 18/06/2009 Révision pour version 3.0 2.0 20/11/2008 Revision pour la 2.0 1.0 01/10/2008 Documentation initiale Commentaires 1 Table des matières 1 Introduction / Identification...
Système d information pour la gestion d un réseau d Université
 Système d information pour la gestion d un réseau d Université Ibticem BEN SAID, [email protected] Sophie BOURGERET, [email protected] Jean-Yves COLLIER, [email protected]
Système d information pour la gestion d un réseau d Université Ibticem BEN SAID, [email protected] Sophie BOURGERET, [email protected] Jean-Yves COLLIER, [email protected]
Objectifs. Clustering. Principe. Applications. Applications. Cartes de crédits. Remarques. Biologie, Génomique
 Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
présentée DEVANT L UNIVERSITÉ DE RENNES 1 pour obtenir le grade de : DOCTEUR DE L UNIVERSITÉ DE RENNES 1 PAR Emilie GUÉRIN TITRE DE LA THÈSE :
 N Ordre de la Thèse 3282 THÈSE présentée DEVANT L UNIVERSITÉ DE RENNES 1 pour obtenir le grade de : DOCTEUR DE L UNIVERSITÉ DE RENNES 1 Mention : BIOLOGIE PAR Emilie GUÉRIN Équipe d accueil : École Doctorale
N Ordre de la Thèse 3282 THÈSE présentée DEVANT L UNIVERSITÉ DE RENNES 1 pour obtenir le grade de : DOCTEUR DE L UNIVERSITÉ DE RENNES 1 Mention : BIOLOGIE PAR Emilie GUÉRIN Équipe d accueil : École Doctorale
Logiciel XLSTAT version 7.0. 40 rue Damrémont 75018 PARIS
 Logiciel XLSTAT version 7.0 Contact : Addinsoft 40 rue Damrémont 75018 PARIS 2005-2006 Plan Présentation générale du logiciel Statistiques descriptives Histogramme Discrétisation Tableau de contingence
Logiciel XLSTAT version 7.0 Contact : Addinsoft 40 rue Damrémont 75018 PARIS 2005-2006 Plan Présentation générale du logiciel Statistiques descriptives Histogramme Discrétisation Tableau de contingence
Orientations pour la gestion documentaire des courriels au gouvernement du Québec
 Orientations pour la gestion documentaire des courriels au gouvernement du Québec Janvier 2009 Dépôt légal Bibliothèque et Archives nationales du Québec, 2010 ISBN : 978-2-550-59635-6 Table des matières
Orientations pour la gestion documentaire des courriels au gouvernement du Québec Janvier 2009 Dépôt légal Bibliothèque et Archives nationales du Québec, 2010 ISBN : 978-2-550-59635-6 Table des matières
Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant
 Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant Parcours: Master 1 : Bioinformatique et biologie des Systèmes dans le Master
Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant Parcours: Master 1 : Bioinformatique et biologie des Systèmes dans le Master
Impact du mobile banking sur les comportements d épargne et de transferts à Madagascar. Florence Arestoff Baptiste Venet
 Impact du mobile banking sur les comportements d épargne et de transferts à Madagascar Florence Arestoff Baptiste Venet 1 Introduction : contexte du contrat de recherche Ce contrat de recherche fait suite
Impact du mobile banking sur les comportements d épargne et de transferts à Madagascar Florence Arestoff Baptiste Venet 1 Introduction : contexte du contrat de recherche Ce contrat de recherche fait suite
Il y a trois types principaux d analyse des résultats : l analyse descriptive, l analyse explicative et l analyse compréhensive.
 L ANALYSE ET L INTERPRÉTATION DES RÉSULTATS Une fois les résultats d une investigation recueillis, on doit les mettre en perspective en les reliant au problème étudié et à l hypothèse formulée au départ:
L ANALYSE ET L INTERPRÉTATION DES RÉSULTATS Une fois les résultats d une investigation recueillis, on doit les mettre en perspective en les reliant au problème étudié et à l hypothèse formulée au départ:
Génétique et génomique Pierre Martin
 Génétique et génomique Pierre Martin Principe de la sélections Repérage des animaux intéressants X Accouplements Programmés Sélection des meilleurs mâles pour la diffusion Index diffusés Indexation simultanée
Génétique et génomique Pierre Martin Principe de la sélections Repérage des animaux intéressants X Accouplements Programmés Sélection des meilleurs mâles pour la diffusion Index diffusés Indexation simultanée
Cellules procaryotes Service histologie Pr.k.mebarek
 Cellules procaryotes Service histologie Pr.k.mebarek I) Les cellules procaryotes II) Les cellules eucaryotes o 1) Caractéristiques générales des cellules eucaryotes o 2) Organisation des cellules eucaryotes
Cellules procaryotes Service histologie Pr.k.mebarek I) Les cellules procaryotes II) Les cellules eucaryotes o 1) Caractéristiques générales des cellules eucaryotes o 2) Organisation des cellules eucaryotes
Didacticiel Études de cas. Description succincte de Pentaho Data Integration Community Edition (Kettle).
 1 Objectif Description succincte de Pentaho Data Integration Community Edition (Kettle). L informatique décisionnelle («Business Intelligence BI» en anglais, ça fait tout de suite plus glamour) fait référence
1 Objectif Description succincte de Pentaho Data Integration Community Edition (Kettle). L informatique décisionnelle («Business Intelligence BI» en anglais, ça fait tout de suite plus glamour) fait référence
La visio-conférence holographique : Pourquoi? Comment?
 La visio-conférence holographique : Pourquoi? Comment? Francis Felix Labo LSIS / Arts & Métiers Paritech (ENSAM) 2 Cours des Arts et Métiers 13100 Aix-en-Provence Thierry Henocque AIP-Primeca Dauphiné
La visio-conférence holographique : Pourquoi? Comment? Francis Felix Labo LSIS / Arts & Métiers Paritech (ENSAM) 2 Cours des Arts et Métiers 13100 Aix-en-Provence Thierry Henocque AIP-Primeca Dauphiné
EXCEL TUTORIEL 2012/2013
 EXCEL TUTORIEL 2012/2013 Excel est un tableur, c est-à-dire un logiciel de gestion de tableaux. Il permet de réaliser des calculs avec des valeurs numériques, mais aussi avec des dates et des textes. Ainsi
EXCEL TUTORIEL 2012/2013 Excel est un tableur, c est-à-dire un logiciel de gestion de tableaux. Il permet de réaliser des calculs avec des valeurs numériques, mais aussi avec des dates et des textes. Ainsi
Transmission d informations sur le réseau électrique
 Transmission d informations sur le réseau électrique Introduction Remarques Toutes les questions en italique devront être préparées par écrit avant la séance du TP. Les préparations seront ramassées en
Transmission d informations sur le réseau électrique Introduction Remarques Toutes les questions en italique devront être préparées par écrit avant la séance du TP. Les préparations seront ramassées en
E-COMMERCE VERS UNE DÉFINITION INTERNATIONALE ET DES INDICATEURS STATISTIQUES COMPARABLES AU NIVEAU INTERNATIONAL
 E-COMMERCE VERS UNE DÉFINITION INTERNATIONALE ET DES INDICATEURS STATISTIQUES COMPARABLES AU NIVEAU INTERNATIONAL Bill Pattinson Division de la politique de l information, de l informatique et de la communication
E-COMMERCE VERS UNE DÉFINITION INTERNATIONALE ET DES INDICATEURS STATISTIQUES COMPARABLES AU NIVEAU INTERNATIONAL Bill Pattinson Division de la politique de l information, de l informatique et de la communication
UTILISATION DE LA PLATEFORME WEB D ANALYSE DE DONNÉES GALAXY
 UTILISATION DE LA PLATEFORME WEB D ANALYSE DE DONNÉES GALAXY Yvan Le Bras [email protected] Cyril Monjeaud, Mathieu Bahin, Claudia Hériveau, Olivier Quenez, Olivier Sallou, Aurélien Roult, Olivier
UTILISATION DE LA PLATEFORME WEB D ANALYSE DE DONNÉES GALAXY Yvan Le Bras [email protected] Cyril Monjeaud, Mathieu Bahin, Claudia Hériveau, Olivier Quenez, Olivier Sallou, Aurélien Roult, Olivier
Vision industrielle et télédétection - Détection d ellipses. Guillaume Martinez 17 décembre 2007
 Vision industrielle et télédétection - Détection d ellipses Guillaume Martinez 17 décembre 2007 1 Table des matières 1 Le projet 3 1.1 Objectif................................ 3 1.2 Les choix techniques.........................
Vision industrielle et télédétection - Détection d ellipses Guillaume Martinez 17 décembre 2007 1 Table des matières 1 Le projet 3 1.1 Objectif................................ 3 1.2 Les choix techniques.........................
Introduction MOSS 2007
 Introduction MOSS 2007 Z 2 Chapitre 01 Introduction à MOSS 2007 v. 1.0 Sommaire 1 SharePoint : Découverte... 3 1.1 Introduction... 3 1.2 Ce que vous gagnez à utiliser SharePoint... 3 1.3 Dans quel cas
Introduction MOSS 2007 Z 2 Chapitre 01 Introduction à MOSS 2007 v. 1.0 Sommaire 1 SharePoint : Découverte... 3 1.1 Introduction... 3 1.2 Ce que vous gagnez à utiliser SharePoint... 3 1.3 Dans quel cas
Module BDR Master d Informatique (SAR)
 Module BDR Master d Informatique (SAR) Cours 6- Bases de données réparties Anne Doucet [email protected] 1 Bases de Données Réparties Définition Conception Décomposition Fragmentation horizontale et
Module BDR Master d Informatique (SAR) Cours 6- Bases de données réparties Anne Doucet [email protected] 1 Bases de Données Réparties Définition Conception Décomposition Fragmentation horizontale et
Windows Internet Name Service (WINS)
 Windows Internet Name Service (WINS) WINDOWS INTERNET NAME SERVICE (WINS)...2 1.) Introduction au Service de nom Internet Windows (WINS)...2 1.1) Les Noms NetBIOS...2 1.2) Le processus de résolution WINS...2
Windows Internet Name Service (WINS) WINDOWS INTERNET NAME SERVICE (WINS)...2 1.) Introduction au Service de nom Internet Windows (WINS)...2 1.1) Les Noms NetBIOS...2 1.2) Le processus de résolution WINS...2
Introduction aux concepts d ez Publish
 Introduction aux concepts d ez Publish Tutoriel rédigé par Bergfrid Skaara. Traduit de l Anglais par Benjamin Lemoine Mercredi 30 Janvier 2008 Sommaire Concepts d ez Publish... 3 Système de Gestion de
Introduction aux concepts d ez Publish Tutoriel rédigé par Bergfrid Skaara. Traduit de l Anglais par Benjamin Lemoine Mercredi 30 Janvier 2008 Sommaire Concepts d ez Publish... 3 Système de Gestion de
Chapitre 5 LE MODELE ENTITE - ASSOCIATION
 Chapitre 5 LE MODELE ENTITE - ASSOCIATION 1 Introduction Conception d une base de données Domaine d application complexe : description abstraite des concepts indépendamment de leur implémentation sous
Chapitre 5 LE MODELE ENTITE - ASSOCIATION 1 Introduction Conception d une base de données Domaine d application complexe : description abstraite des concepts indépendamment de leur implémentation sous
Convergence Grand public professionnelle
 Note de synthèse Convergence Grand public professionnelle Cette note synthétise les réflexions d une des tables rondes des entretiens Télécom de Mars 2006, organisés par Finaki. A cette table étaient à
Note de synthèse Convergence Grand public professionnelle Cette note synthétise les réflexions d une des tables rondes des entretiens Télécom de Mars 2006, organisés par Finaki. A cette table étaient à
Rapport d'analyse des besoins
 Projet ANR 2011 - BR4CP (Business Recommendation for Configurable products) Rapport d'analyse des besoins Janvier 2013 Rapport IRIT/RR--2013-17 FR Redacteur : 0. Lhomme Introduction...4 La configuration
Projet ANR 2011 - BR4CP (Business Recommendation for Configurable products) Rapport d'analyse des besoins Janvier 2013 Rapport IRIT/RR--2013-17 FR Redacteur : 0. Lhomme Introduction...4 La configuration
Le scoring est-il la nouvelle révolution du microcrédit?
 Retour au sommaire Le scoring est-il la nouvelle révolution du microcrédit? BIM n 32-01 octobre 2002 Frédéric DE SOUSA-SANTOS Le BIM de cette semaine se propose de vous présenter un ouvrage de Mark Schreiner
Retour au sommaire Le scoring est-il la nouvelle révolution du microcrédit? BIM n 32-01 octobre 2002 Frédéric DE SOUSA-SANTOS Le BIM de cette semaine se propose de vous présenter un ouvrage de Mark Schreiner
Instructions relatives à la soumission d une question sur le sujet d étude
 Programme de bourses de recherche Amy Mahan pour évaluer l impact de l accès public aux TIC Instructions relatives à la soumission d une question sur le sujet d étude Table des matières À propos la Question
Programme de bourses de recherche Amy Mahan pour évaluer l impact de l accès public aux TIC Instructions relatives à la soumission d une question sur le sujet d étude Table des matières À propos la Question
Cours Base de données relationnelles. M. Boughanem, IUP STRI
 Cours Base de données relationnelles 1 Plan 1. Notions de base 2. Modèle relationnel 3. SQL 2 Notions de base (1) Définition intuitive : une base de données est un ensemble d informations, (fichiers),
Cours Base de données relationnelles 1 Plan 1. Notions de base 2. Modèle relationnel 3. SQL 2 Notions de base (1) Définition intuitive : une base de données est un ensemble d informations, (fichiers),
MAB Solut. vos projets. MABLife Génopole Campus 1 5 rue Henri Desbruères 91030 Evry Cedex. www.mabsolut.com. intervient à chaque étape de
 Mabsolut-DEF-HI:Mise en page 1 17/11/11 17:45 Page1 le département prestataire de services de MABLife de la conception à la validation MAB Solut intervient à chaque étape de vos projets Création d anticorps
Mabsolut-DEF-HI:Mise en page 1 17/11/11 17:45 Page1 le département prestataire de services de MABLife de la conception à la validation MAB Solut intervient à chaque étape de vos projets Création d anticorps
Biochimie I. Extraction et quantification de l hexokinase dans Saccharomyces cerevisiae 1. Assistants : Tatjana Schwabe Marcy Taylor Gisèle Dewhurst
 Biochimie I Extraction et quantification de l hexokinase dans Saccharomyces cerevisiae 1 Daniel Abegg Sarah Bayat Alexandra Belfanti Assistants : Tatjana Schwabe Marcy Taylor Gisèle Dewhurst Laboratoire
Biochimie I Extraction et quantification de l hexokinase dans Saccharomyces cerevisiae 1 Daniel Abegg Sarah Bayat Alexandra Belfanti Assistants : Tatjana Schwabe Marcy Taylor Gisèle Dewhurst Laboratoire
Utiliser un tableau de données
 Utiliser un tableau de données OBJECTIFS : - Définir une Base de Données. - Présentation : tableau de données. - Création d un tableau de données - Gestion d un tableau de données. - Trier et Filtrer des
Utiliser un tableau de données OBJECTIFS : - Définir une Base de Données. - Présentation : tableau de données. - Création d un tableau de données - Gestion d un tableau de données. - Trier et Filtrer des
Sujet proposé par Yves M. LEROY. Cet examen se compose d un exercice et de deux problèmes. Ces trois parties sont indépendantes.
 Promotion X 004 COURS D ANALYSE DES STRUCTURES MÉCANIQUES PAR LA MÉTHODE DES ELEMENTS FINIS (MEC 568) contrôle non classant (7 mars 007, heures) Documents autorisés : polycopié ; documents et notes de
Promotion X 004 COURS D ANALYSE DES STRUCTURES MÉCANIQUES PAR LA MÉTHODE DES ELEMENTS FINIS (MEC 568) contrôle non classant (7 mars 007, heures) Documents autorisés : polycopié ; documents et notes de
La sécurité dans un réseau Wi-Fi
 La sécurité dans un réseau Wi-Fi Par Valérian CASTEL. Sommaire - Introduction : Le Wi-Fi, c est quoi? - Réseau ad hoc, réseau infrastructure, quelles différences? - Cryptage WEP - Cryptage WPA, WPA2 -
La sécurité dans un réseau Wi-Fi Par Valérian CASTEL. Sommaire - Introduction : Le Wi-Fi, c est quoi? - Réseau ad hoc, réseau infrastructure, quelles différences? - Cryptage WEP - Cryptage WPA, WPA2 -
La virtualisation des serveurs ou «loin des yeux, loin de l esprit»...
 Acceleris GmbH Webergutstr. 2 CH-3052 Zollikofen Téléphone: 031 911 33 22 [email protected] La virtualisation des serveurs ou «loin des yeux, loin de l esprit»... Des entreprises de toutes les branches
Acceleris GmbH Webergutstr. 2 CH-3052 Zollikofen Téléphone: 031 911 33 22 [email protected] La virtualisation des serveurs ou «loin des yeux, loin de l esprit»... Des entreprises de toutes les branches
Présentation Générale
 Mars 2009 Présentation Générale 1- Le Master Recherche en Sciences de la Vie et de la Santé à Nice Objectifs, environnement scientifique, organisation Enseignements, les spécialités, les cours et les stages
Mars 2009 Présentation Générale 1- Le Master Recherche en Sciences de la Vie et de la Santé à Nice Objectifs, environnement scientifique, organisation Enseignements, les spécialités, les cours et les stages
AA-SO5 KIDA/GSOV/VAMDC
 AA-SO5 Centres de traitement et d archivage de données KIDA - 2 mars 2015 AA-SO5 KIDA/GSOV/VAMDC Contexte général L observation des molécules dans le milieu interstellaire, mais aussi dans les atmosphères
AA-SO5 Centres de traitement et d archivage de données KIDA - 2 mars 2015 AA-SO5 KIDA/GSOV/VAMDC Contexte général L observation des molécules dans le milieu interstellaire, mais aussi dans les atmosphères
SOCLE COMMUN - La Compétence 3 Les principaux éléments de mathématiques et la culture scientifique et technologique
 SOCLE COMMUN - La Compétence 3 Les principaux éléments de mathématiques et la culture scientifique et technologique DOMAINE P3.C3.D1. Pratiquer une démarche scientifique et technologique, résoudre des
SOCLE COMMUN - La Compétence 3 Les principaux éléments de mathématiques et la culture scientifique et technologique DOMAINE P3.C3.D1. Pratiquer une démarche scientifique et technologique, résoudre des
La classification automatique de données quantitatives
 La classification automatique de données quantitatives 1 Introduction Parmi les méthodes de statistique exploratoire multidimensionnelle, dont l objectif est d extraire d une masse de données des informations
La classification automatique de données quantitatives 1 Introduction Parmi les méthodes de statistique exploratoire multidimensionnelle, dont l objectif est d extraire d une masse de données des informations
Bases de données et outils bioinformatiques utiles en génétique
 Bases de données et outils bioinformatiques utiles en génétique Collège National des Enseignants et Praticiens de Génétique Médicale C. Beroud Date de création du document 2010-2011 Table des matières
Bases de données et outils bioinformatiques utiles en génétique Collège National des Enseignants et Praticiens de Génétique Médicale C. Beroud Date de création du document 2010-2011 Table des matières
Serveur Appliance IPAM et Services Réseaux
 Page 1 Datasheet Serveur Appliance IPAM et Services Réseaux SIMPLIFER LE DEPLOIEMENT DE VOS ARCHITECTURES & DHCP Les services d adressage et de nommage sont au cœur de votre système d information, car
Page 1 Datasheet Serveur Appliance IPAM et Services Réseaux SIMPLIFER LE DEPLOIEMENT DE VOS ARCHITECTURES & DHCP Les services d adressage et de nommage sont au cœur de votre système d information, car
A.-M. Cubat PMB - Import de lecteurs - Généralités Page 1 Source : http://amcubat.be/docpmb/import-de-lecteurs
 A.-M. Cubat PMB - Import de lecteurs - Généralités Page 1 Diverses méthodes d import de lecteurs Les données (noms, prénoms, adresses. des lecteurs) proviennent en général du secrétariat, et se trouvent
A.-M. Cubat PMB - Import de lecteurs - Généralités Page 1 Diverses méthodes d import de lecteurs Les données (noms, prénoms, adresses. des lecteurs) proviennent en général du secrétariat, et se trouvent
Bases de données des mutations
 Bases de données des mutations CFMDB CFTR2 CFTR-France / Registre Corinne THEZE, Corinne BAREIL Laboratoire de génétique moléculaire Montpellier Atelier Muco, Lille, 25-27 septembre 2014 Accès libre http://www.genet.sickkids.on.ca/app
Bases de données des mutations CFMDB CFTR2 CFTR-France / Registre Corinne THEZE, Corinne BAREIL Laboratoire de génétique moléculaire Montpellier Atelier Muco, Lille, 25-27 septembre 2014 Accès libre http://www.genet.sickkids.on.ca/app
Groupe Eyrolles, 2006, ISBN : 2-212-11734-5
 Groupe Eyrolles, 2006, ISBN : 2-212-11734-5 Chapitre 6 La gestion des incidents Quelles que soient la qualité du système d information mis en place dans l entreprise ou les compétences des techniciens
Groupe Eyrolles, 2006, ISBN : 2-212-11734-5 Chapitre 6 La gestion des incidents Quelles que soient la qualité du système d information mis en place dans l entreprise ou les compétences des techniciens
Recherche et veille documentaire scientifique
 Recherche et veille documentaire scientifique Élodie Chattot BU Médecine Pharmacie juin 2006 Introduction Le but de cet atelier est de vous initier à la méthodologie de recherche documentaire scientifique,
Recherche et veille documentaire scientifique Élodie Chattot BU Médecine Pharmacie juin 2006 Introduction Le but de cet atelier est de vous initier à la méthodologie de recherche documentaire scientifique,
Les Réseaux sans fils : IEEE 802.11. F. Nolot
 Les Réseaux sans fils : IEEE 802.11 F. Nolot 1 Les Réseaux sans fils : IEEE 802.11 Historique F. Nolot 2 Historique 1er norme publiée en 1997 Débit jusque 2 Mb/s En 1998, norme 802.11b, commercialement
Les Réseaux sans fils : IEEE 802.11 F. Nolot 1 Les Réseaux sans fils : IEEE 802.11 Historique F. Nolot 2 Historique 1er norme publiée en 1997 Débit jusque 2 Mb/s En 1998, norme 802.11b, commercialement
Bientôt plus d'1 Français sur 10 client d'une banque en ligne.
 www.audirep.fr Bientôt plus d'1 Français sur 10 client d'une banque en ligne. Restitution des résultats d une étude menée par internet sur un échantillon de 1 001 personnes représentatives de la population
www.audirep.fr Bientôt plus d'1 Français sur 10 client d'une banque en ligne. Restitution des résultats d une étude menée par internet sur un échantillon de 1 001 personnes représentatives de la population
EVALUATION DE LA QUALITE DES SONDAGES EN LIGNE : CAS D UN SONDAGE D OPINION AU BURKINA FASO
 EVALUATION DE LA QUALITE DES SONDAGES EN LIGNE : CAS D UN SONDAGE D OPINION AU BURKINA FASO Auteur Baguinébié Bazongo 1 Ingénieur Statisticien Economiste Chef de l Unité de recherche à l Institut national
EVALUATION DE LA QUALITE DES SONDAGES EN LIGNE : CAS D UN SONDAGE D OPINION AU BURKINA FASO Auteur Baguinébié Bazongo 1 Ingénieur Statisticien Economiste Chef de l Unité de recherche à l Institut national
