Manipuler des données volumineuses : les
|
|
|
- Zoé Mongeau
- il y a 10 ans
- Total affichages :
Transcription
1 Fiche TD avec le logiciel : tdr16 Manipuler des données volumineuses : les génomes bactériens J.R. Lobry & D. Chessel Pour tester l aptitude de R à manipuler de grands ensembles de données, la fiche propose d implanter un tableau séquences - codons de taille respectable. Table des matières 1 Introduction 2 2 Les données 3 3 Importer un grand tableau 4 4 Sauver et restaurer un grand tableau 5 5 Taille mémoire 6 6 Tableaux dérivés Créer le tableau espèces-codons Créer le tableau séquences-acides aminés Créer le tableau espèces-acides aminés Créer le tableau séquences-bases Créer le tableau espèces-bases Exercices 9 Références 10 1
2 1 Introduction Soit C l ensemble des 64 codons possibles dans les séquences codantes, C = {AAA, AAC, AAG, AAT,..., T T T }, et soit A l ensemble formé de l union de l ensemble vide et des 20 acides aminés des protéine, A = {, Ala, Arg,..., V al}, où l ensemble vide représente les codons non-sens utilisés comme fin de signal de la traduction et les codons non-assignés. Un code génétique est une application surjective de C dans A, comme dans l exemple ci-dessous du code génétique standard. Si l on néglige les cas particuliers du codon d initiation de la traduction, ainsi qu un certain nombre d exceptions telles que celle de la traduction de la sélénocystéine, un code génétique est donné par une chaîne de 64 caractères pouvant prendre 21 valeurs. Les codes génétiques connus sont représentés dans l alignement ci-dessous, où les déviations par rapport au code génétique standard sont mises en évidence : Base1 Base2 Base3 = TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG = TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG 1 FFLLSSSSYY??CC?WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG 4...W C W...M...?? W.TTTT...M W...M...SS... Logiciel R version ( ) tdr16.rnw Page 2/10 Compilé le
3 21...W...M...N...SS W...N...SS Y...W...N...SS W...M...GG L ?...L ? Q QQ S The Standard Code. 4. The Mold, Protozoan, and Coelenterate Mitochondrial Code and the Mycoplasma/Spiroplasma Code. 10. The Euplotid Nuclear Code. 2. The Vertebrate Mitochondrial Code. 3. The Yeast Mitochondrial Code. 5. The Invertebrate Mitochondrial Code. 21. Trematode Mitochondrial Code. 9. The Echinoderm Mitochondrial Code. 14. The Flatworm Mitochondrial Code. 13. The Ascidian Mitochondrial Code. 16. Chlorophycean Mitochondrial Code. 22. Scenedesmus obliquus mitochondrial Code. 23. Thraustochytrium Mitochondrial Code. 15. Blepharisma Nuclear Code. 6. The Ciliate, Dasycladacean and Hexamita Nuclear Code. 12. The Alternative Yeast Nuclear Code. (source : La plupart des codes variants sont trouvés dans les génomes mitochondriaux. Chez les bactéries, seul le code standard est utilisé à l exception des Mycoplasmes et Spiroplasmes qui utilisent le plus courant des codes variants (numéro 4) où le codon stop standard TGA code pour le tryptophane (W). Nous ferons l impasse sur cette particularité. 2 Les données Elles sont dans quatre fichiers à récupérer dans http ://pbil.univ-lyon1.fr/r/donnees. Récupérer ces quatre fichiers dans un nouveau dossier de travail : path <- " telecharger <- function(quoi) { download.file(url = paste(path, quoi, sep = "/"), destfile = paste(getwd(), quoi, sep =.Platform$file.sep)) } telecharger("fracod.txt") telecharger("fracod_aa.txt") telecharger("fracod_cod.txt") telecharger("fracod_esp.txt") Il se peut que ce code ne marche pas si vous passez par un proxy pour votre connexion internet. Vous pouvez essayer de neutraliser le proxy avec Sys.putenv(no_proxy = ""), ou télécharger les fichiers à la main dans votre espace de travail. Vous devez avoir les quatre fichiers dans votre espace de travail (donné par getwd()) pour pouvoir continuer cette fiche de TD : Logiciel R version ( ) tdr16.rnw Page 3/10 Compilé le
4 Lire directement fracod_cod.txt comme vecteur de chaînes de caractères : codcol <- readlines("fracod_cod.txt") codcol [1] "TTT" "TTC" "TTA" "TTG" "TCT" "TCC" "TCA" "TCG" "TAT" "TAC" "TAA" "TAG" "TGT" [14] "TGC" "TGA" "TGG" "CTT" "CTC" "CTA" "CTG" "CCT" "CCC" "CCA" "CCG" "CAT" "CAC" [27] "CAA" "CAG" "CGT" "CGC" "CGA" "CGG" "ATT" "ATC" "ATA" "ATG" "ACT" "ACC" "ACA" [40] "ACG" "AAT" "AAC" "AAA" "AAG" "AGT" "AGC" "AGA" "AGG" "GTT" "GTC" "GTA" "GTG" [53] "GCT" "GCC" "GCA" "GCG" "GAT" "GAC" "GAA" "GAG" "GGT" "GGC" "GGA" "GGG" On a les 64 codons. Faire avec fracod_esp.txt un facteur appelé codlig : codlig <- as.factor(readlines("fracod_esp.txt")) summary(codlig) AERPECG AQUAECG ARCFUCG BACHDCG BACSUCG BORBUCG BUCAICG CAMJECG CHLMUCG CHLPNACG CHLPNCCG CHLPNJCG CHLTRCG DEIRAC1 DEIRAC2 DEIRACP1 DEIRAMP1 ECOLICG HAEINCG HALSPCG HALSPP1 HALSPP2 HELPJCG HELPYCG METJACG METTHCG MYCGECG MYCPNCG MYCTUCG NEIMACG NMENCG PSEAECG PYRABCG PYRHOCG RICPRCG SYNY3CG THEACCG THEMACG TREPACG UREURCG VIBCHC1 VIBCHC2 XYLFACG Les codes des espèces sont les suivants : AERPECG Aeropyrum pernix K1 complete genome. AQUAECG Aquifex aeolicus complete genome. ARCFUCG Archaeoglobus fulgidus complete genome. BACHDCG Bacillus halodurans C-125, complete genome. BACSUCG Bacillus subtilis complete genome. BORBUCG Borrelia burgdorferi complete genome. BUCAICG Buchnera sp. APS complete genome. CAMJECG Campylobacter jejuni complete genome. CHLMUCG Chlamydia muridarum complete genome. CHLPNACG Chlamydophila pneumoniae AR39 complete genome. CHLPNCCG Chlamydophila pneumoniae CWL029 complete genome. CHLPNJCG Chlamydophila pneumoniae J138 complete genome. CHLTRCG Chlamydia trachomatis complete genome. DEIRAC1 Deinococcus radiodurans R1 complete chromosome 1. DEIRAC2 Deinococcus radiodurans R1 complete chromosome 2. DEIRACP1 Deinococcus radiodurans Plasmid CP1. DEIRAMP1 Deinococcus radiodurans Plasmid MP1. ECOLICG Escherichia coli K-12 MG1655. HAEINCG Haemophilus influenzae Rd complete genome. HALSPCG Halobacterium sp. NRC-1 complete genome. HALSPP1 Halobacterium sp. NRC-1 plasmid pnrc100, complete sequence. HALSPP2 Halobacterium sp. NRC-1 plasmid pnrc200 complete genome. HELPJCG Helicobacter pylori, strain J99 complete genome. HELPYCG Helicobacter pylori METJACG Methanococcus jannaschii complete genome. METTHCG Genome of Methanobacterium thermoautotrophicum delta H. MYCGECG Mycoplasma genitalium complete genome. MYCPNCG Mycoplasma pneumoniae complete genome. MYCTUCG Mycobacterium tuberculosis H37Rv complete genome. NEIMACG Neisseria meningitidis serogroup A strain Z2491 complete genome. NMENCG Neisseria meningitidis serogroup B strain MC58 complete genome. PSEAECG Pseudomonas aeruginosa PA01, complete genome. PYRABCG Pyrococcus abyssi complete genome. PYRHOCG Pyrococcus horikoshii OT3 complete genome. RICPRCG Rickettsia prowazekii strain Madrid E, complete genome. SYNY3CG Synechocystis PCC6803 complete genome. THEACCG Thermoplasma acidophilum, complete genome. THEMACG Thermotoga maritima complete genome. TREPACG Treponema pallidum complete genome. UREURCG Ureaplasma urealyticum complete genome. VIBCHC1 Vibrio cholerae chromosome I, complete chromosome. VIBCHC2 Vibrio cholerae chromosome II, complete chromosome. XYLFACG Xylella fastidiosa, complete genome. On pourra consulter le lien utile pour en savoir plus sur ces génomes : http :// 3 Importer un grand tableau Il faut savoir que la fonction read.table(), qui est très pratique pour les petits jeux de données, n est pas adaptée aux jeux de données de taille plus conséquente. En effet, read.table() fait appel à la fonction scan() pour lire les données puis fait une analyse de ce qui a été lu pour essayer de déterminer Logiciel R version ( ) tdr16.rnw Page 4/10 Compilé le
5 au mieux les types des colonnes du data.frame qui va être construit. Cette opération d analyse demande beaucoup de temps calcul. Quand on sait ce que l on veut importer, il est préférable d utiliser directement la fonction scan(). Cette dernière renvoie un vecteur que l on transforme en matrice puis en data.frame. tempsimport <- system.time(fracod <- as.data.frame(matrix(data = scan(file = "fracod.txt", what = integer(0)), nrow = 67712, ncol = 64, byrow = TRUE))) tempsimport user system elapsed Ainsi, pour importer un tableau de 67,712 lignes et 64 colonnes, soit 4,333,568 éléments en tout, nous avons consommé ici 1.66 secondes de CPU utilisateur (ce qui correspond dans la vie réelle à une attente de 1.96 secondes lors de la dernière compilation de ce document). A titre indicatif, une importation du même tableau dans les mêmes conditions avec fracod <- read.table("fracod.txt") demande environ secondes de CPU utilisateur et secondes d attente réelle, soit approximativement 4 minutes. Il est donc clair que scan() est beaucoup plus rapide que read.table(), mais on peut encore faire mieux. 4 Sauver et restaurer un grand tableau L idée est qu après avoir importé des données sous R, on aime bien en général les polir. Après cette phase de polissage il suffit de les sauvegarder comme des objets R (en binaire compatible multi-ordinateur) pour pouvoir ultérieurement les recharger très rapidement. Par exemple, dans notre cas l objet fracod est un data.frame dont le nom des colonnes n est pas très explicite : names(fracod) [1] "V1" "V2" "V3" "V4" "V5" "V6" "V7" "V8" "V9" "V10" "V11" "V12" "V13" [14] "V14" "V15" "V16" "V17" "V18" "V19" "V20" "V21" "V22" "V23" "V24" "V25" "V26" [27] "V27" "V28" "V29" "V30" "V31" "V32" "V33" "V34" "V35" "V36" "V37" "V38" "V39" [40] "V40" "V41" "V42" "V43" "V44" "V45" "V46" "V47" "V48" "V49" "V50" "V51" "V52" [53] "V53" "V54" "V55" "V56" "V57" "V58" "V59" "V60" "V61" "V62" "V63" "V64" On décide donc de mieux documenter l objet : names(fracod) <- readlines("fracod_cod.txt") names(fracod) [1] "TTT" "TTC" "TTA" "TTG" "TCT" "TCC" "TCA" "TCG" "TAT" "TAC" "TAA" "TAG" "TGT" [14] "TGC" "TGA" "TGG" "CTT" "CTC" "CTA" "CTG" "CCT" "CCC" "CCA" "CCG" "CAT" "CAC" [27] "CAA" "CAG" "CGT" "CGC" "CGA" "CGG" "ATT" "ATC" "ATA" "ATG" "ACT" "ACC" "ACA" [40] "ACG" "AAT" "AAC" "AAA" "AAG" "AGT" "AGC" "AGA" "AGG" "GTT" "GTC" "GTA" "GTG" [53] "GCT" "GCC" "GCA" "GCG" "GAT" "GAC" "GAA" "GAG" "GGT" "GGC" "GGA" "GGG" Maintenant que nous sommes satisfaits, nous sauvegardons l objet fracod en binaire au format XDR (c est un format utilisé par toutes les variétés de R, on peut sauvegarder sous Linux et relire sous Mac ou PC, c est rapide et portable) : save(fracod, file = "fracod.rdata") Vous pouvez maintenant quitter votre session R, puis relancer R pour recharger le jeu de données (n oubliez pas de sélectionner le bon répertoire de travail avant d exécuter le code suivant, sinon le fichier ne sera pas trouvé) : tempsxrd <- system.time(load("fracod.rdata")) tempsxrd Logiciel R version ( ) tdr16.rnw Page 5/10 Compilé le
6 user system elapsed Ainsi, pour restaurer un tableau de 67,712 lignes et 64 colonnes, soit 4,333,568 éléments en tout, correspondant à 22,619,749 observations, nous avons consommé ici 0.63 secondes de CPU utilisateur (ce qui correspond dans la vie réelle à une attente de 0.71 secondes lors de la dernière compilation de ce document). R est donc bien capable de manipuler de grands ensembles de données, cependant un couplage avec un SGBD sera préférable pour extraire les informations pertinentes à la volée pour des applications plus conséquentes (il y a des packages R pour cela). 5 Taille mémoire Tous les objets R sont placés dans la mémoire vive de l ordinateur. Une estimation de la place occupée par un objet est donnée par la fonction object.size() : taillefracod <- object.size(fracod) taillefracod bytes Ainsi, notre objet fracod occupe environ 34,945,216 octets en mémoire (soit Mio). La taille mémoire maximum disponible dépend de votre machine et de son système d exploitation. Si vous dépassez cette capacité vous aurez un message du type : Error: cannot allocate vector of size Kb In addition: Warning message: Reached total allocation of 476Mb: see help(memory.size) 6 Tableaux dérivés Faire avec fracod_aa.txt un facteur appelé codaa : codaa <- factor(readlines("fracod_aa.txt")) codaa [1] Phe Phe Leu Leu Ser Ser Ser Ser Tyr Tyr Stp Stp Cys Cys Stp Trp Leu Leu Leu Leu [21] Pro Pro Pro Pro His His Gln Gln Arg Arg Arg Arg Ile Ile Ile Met Thr Thr Thr Thr [41] Asn Asn Lys Lys Ser Ser Arg Arg Val Val Val Val Ala Ala Ala Ala Asp Asp Glu Glu [61] Gly Gly Gly Gly 21 Levels: Ala Arg Asn Asp Cys Gln Glu Gly His Ile Leu Lys Met Phe Pro Ser... Val Réorganiser le tableau pour que les codons apparaissent par blocs d acides aminés et vérifier. frasort <- fracod[, order(codaa)] codsort <- codaa[order(codaa)] names(frasort) [1] "GCT" "GCC" "GCA" "GCG" "CGT" "CGC" "CGA" "CGG" "AGA" "AGG" "AAT" "AAC" "GAT" "GAC" [15] "TGT" "TGC" "CAA" "CAG" "GAA" "GAG" "GGT" "GGC" "GGA" "GGG" "CAT" "CAC" "ATT" "ATC" [29] "ATA" "TTA" "TTG" "CTT" "CTC" "CTA" "CTG" "AAA" "AAG" "ATG" "TTT" "TTC" "CCT" "CCC" [43] "CCA" "CCG" "TCT" "TCC" "TCA" "TCG" "AGT" "AGC" "TAA" "TAG" "TGA" "ACT" "ACC" "ACA" [57] "ACG" "TGG" "TAT" "TAC" "GTT" "GTC" "GTA" "GTG" dim(frasort) [1] sum(frasort) Logiciel R version ( ) tdr16.rnw Page 6/10 Compilé le
7 [1] codsort [1] Ala Ala Ala Ala Arg Arg Arg Arg Arg Arg Asn Asn Asp Asp Cys Cys Gln Gln Glu Glu Gly [22] Gly Gly Gly His His Ile Ile Ile Leu Leu Leu Leu Leu Leu Lys Lys Met Phe Phe Pro Pro [43] Pro Pro Ser Ser Ser Ser Ser Ser Stp Stp Stp Thr Thr Thr Thr Trp Tyr Tyr Val Val Val [64] Val 21 Levels: Ala Arg Asn Asp Cys Gln Glu Gly His Ile Leu Lys Met Phe Pro Ser Stp... Val 6.1 Créer le tableau espèces-codons espcod <- aggregate(x = frasort, by = list(espece = codlig), FUN = sum) rownames(espcod) <- espcod$espece espcod <- espcod[, -1] library(ade4) scatter(dudi.coa(espcod, scann = FALSE, nf = 2)) Eigenvalues d = 0.5 UREURCG TTA AAT ATT HAEINCG BUCAICG ECOLICG CAMJECG MYCGECG HELPYCG HELPJCG SYNY3CG VIBCHC2 VIBCHC1 AAA TTT MYCPNCG NMENCG NEIMACG AGT TGT TAA XYLFACG GCG CGG RICPRCG TCT CHLMUCG ACT TAT GGC CHLTRCG GAT BACHDCG CHLPNCCG CHLPNACG CHLPNJCG GAA BACSUCG GGT ATC ACC CCG CAG DEIRAMP1 MYCTUCG DEIRAC1 DEIRAC2 PSEAECG BORBUCG GCT TREPACG GCC CTG TGG GTG CCT GCA ATG DEIRACP1 TGC TCG ACG AAC TGA AGC HALSPP1 CAC HALSPCG HALSPP2 GTC METJACG AGA CAA CGT ACA TCA GTA CTA CCA CTT GTT GGA CGA CAT TTG GGG GAC TCC CCC TAG TTC TAC CTC GAG AAG THEMACG PYRHOCG THEACCG AQUAECG ARCFUCG AERPECG METTHCG PYRABCG CGC ATA AGG 6.2 Créer le tableau séquences-acides aminés fbact1 <- function() { w <- data.frame(matrix(0, nrow(frasort), length(levels(codsort)))) names(w) <- levels(codsort) row.names(w) <- row.names(frasort) for (i in 1:21) { a0 <- levels(codsort)[i] c0 <- which(codsort == a0) w[, i] <- apply(data.frame(frasort[, c0]), 1, sum) } return(w) } fraa <- fbact1() frasort[1, ] Logiciel R version ( ) tdr16.rnw Page 7/10 Compilé le
8 GCT GCC GCA GCG CGT CGC CGA CGG AGA AGG AAT AAC GAT GAC TGT TGC CAA CAG GAA GAG AERPECG GGT GGC GGA GGG CAT CAC ATT ATC ATA TTA TTG CTT CTC CTA CTG AAA AAG ATG TTT TTC AERPECG CCT CCC CCA CCG TCT TCC TCA TCG AGT AGC TAA TAG TGA ACT ACC ACA ACG TGG TAT TAC AERPECG GTT GTC GTA GTG AERPECG fraa[1, ] Ala Arg Asn Asp Cys Gln Glu Gly His Ile Leu Lys Met Phe Pro Ser Stp Thr Trp Tyr AERPECG Val AERPECG Créer le tableau espèces-acides aminés espaa <- aggregate(x = fraa, by = list(espece = codlig), FUN = sum) rownames(espaa) <- espaa$espece espaa <- espaa[, -1] scatter(dudi.coa(espaa, scann = FALSE, nf = 2)) Eigenvalues d = 0.2 Gln MYCPNCG MYCGECG UREURCG VIBCHC2 SYNY3CG His Cys VIBCHC1 CHLMUCG BUCAICG ECOLICG CHLTRCG CHLPNCCG CHLPNACG CHLPNJCG HAEINCG HELPJCG HELPYCG RICPRCG CAMJECG DEIRAMP1 XYLFACG NEIMACG NMENCG DEIRAC2 Ala Thr Leu Ser Stp PSEAECG Trp DEIRACP1 Phe DEIRAC1 TREPACG BACSUCG BORBUCG Asp BACHDCG MYCTUCG Pro Met HALSPP2 HALSPP1 Gly Ile Lys Tyr HALSPCG THEACCG Arg AERPECG Val Glu METTHCG THEMACG ARCFUCG PYRHOCG AQUAECG PYRABCG Asn METJACG 6.4 Créer le tableau séquences-bases fbact2 <- function() { codons <- names(frasort) pos1 <- as.factor(substr(codons, 1, 1)) pos2 <- as.factor(substr(codons, 2, 2)) pos3 <- as.factor(substr(codons, 3, 3)) loc1 <- function(x) { x <- as.numeric(x) w <- xtabs(x ~ pos1) w <- w + xtabs(x ~ pos2) w <- w + xtabs(x ~ pos3) Logiciel R version ( ) tdr16.rnw Page 8/10 Compilé le
9 return(w) } w <- t(apply(frasort, 1, loc1)) return(w) } frabase <- fbact2() frabase[1, ] A C G T sum(frabase)/3 [1] Créer le tableau espèces-bases espbase <- aggregate(x = frabase, by = list(espece = codlig), FUN = sum) rownames(espbase) <- espbase$espece espbase <- espbase[, -1] scatter(dudi.coa(espbase, scann = FALSE, nf = 2)) Eigenvalues d = 0.2 CHLPNCCG CHLPNACG CHLPNJCG CHLTRCG CHLMUCG SYNY3CG C XYLFACG TREPACG DEIRAMP1 ECOLICG VIBCHC1 VIBCHC2 T HAEINCG PSEAECG MYCPNCG DEIRAC1 DEIRAC2 MYCTUCG DEIRACP1 NEIMACG NMENCG HELPJCG HELPYCG HALSPP2 AERPECG BACSUCG BACHDCG HALSPCG HALSPP1 G THEACCG METTHCG THEMACG PYRHOCG A ARCFUCG PYRABCG AQUAECG MYCGECG CAMJECG RICPRCG BORBUCG BUCAICG UREURCG METJACG 7 Exercices Vérifiez sur ces données le résultat classique [1] selon lequel les organismes qui ont un fort taux de G+C utilisent préférentiellement des acides aminés codés par des codons riches en G+C. Pour cela, vous agrégerez les données par espèces avant de représenter l évolution des fréquences des acides-aminés en fonction du taux de G+C. Question subsidiaire : peut on dire que le taux de G+C est le facteur majeur de variabilité inter-spécifique au niveau des codons, au niveau des acides-aminés? Logiciel R version ( ) tdr16.rnw Page 9/10 Compilé le
10 Références [1] N. Sueoka. Correlation between base composition of deoxyribonucleic acid and amino acid composition of protein. Proc. Natl. Acad. Sci. USA, 47 : , Logiciel R version ( ) tdr16.rnw Page 10/10 Compilé le
Analyse d échantillons alimentaires pour la présence d organismes génétiquement modifiés
 Analyse d échantillons alimentaires pour la présence d organismes génétiquement modifiés Module 11 Détection quantitative du soja Roundup Ready par PCR en temps réel N. Foti WORLD HEALTH ORGANIZATION REGIONAL
Analyse d échantillons alimentaires pour la présence d organismes génétiquement modifiés Module 11 Détection quantitative du soja Roundup Ready par PCR en temps réel N. Foti WORLD HEALTH ORGANIZATION REGIONAL
Leslie REGAD ; Gaëlle LELANDAIS. leslie.regad@univ- paris- diderot.fr ; gaelle.lelandais@univ- paris- diderot.fr
 Programmation avec R Polycopié d exercices Leslie REGAD ; Gaëlle LELANDAIS leslie.regad@univ- paris- diderot.fr ; gaelle.lelandais@univ- paris- diderot.fr Ce polycopié rassemble différents exercices vous
Programmation avec R Polycopié d exercices Leslie REGAD ; Gaëlle LELANDAIS leslie.regad@univ- paris- diderot.fr ; gaelle.lelandais@univ- paris- diderot.fr Ce polycopié rassemble différents exercices vous
Initiation à l analyse en composantes principales
 Fiche TD avec le logiciel : tdr601 Initiation à l analyse en composantes principales A.B. Dufour & J.R. Lobry Une première approche très intuitive et interactive de l ACP. Centrage et réduction des données.
Fiche TD avec le logiciel : tdr601 Initiation à l analyse en composantes principales A.B. Dufour & J.R. Lobry Une première approche très intuitive et interactive de l ACP. Centrage et réduction des données.
Détection des duplications en tandem au niveau nucléique à l'aide de la théorie des flots
 Université Toulouse 3 Paul Sabatier(UT3 Paul Sabatier) Informatique Spécialité Bioinformatique Eric AUDEMARD lundi 28 novembre 2011 Détection des duplications en tandem au niveau nucléique à l'aide de
Université Toulouse 3 Paul Sabatier(UT3 Paul Sabatier) Informatique Spécialité Bioinformatique Eric AUDEMARD lundi 28 novembre 2011 Détection des duplications en tandem au niveau nucléique à l'aide de
SEMIN. Données sous R : stockage et échange. Julio PEDRAZA ACOSTA
 SEMIN Données sous R : stockage et échange Julio PEDRAZA ACOSTA UMR 5202 Origine Structure et Evolution de la Biodiversité Département Systématique et Evolution, MNHN [email protected] SEMIN R du MNHN 10
SEMIN Données sous R : stockage et échange Julio PEDRAZA ACOSTA UMR 5202 Origine Structure et Evolution de la Biodiversité Département Systématique et Evolution, MNHN [email protected] SEMIN R du MNHN 10
Comment reproduire les résultats de l article : POP-Java : Parallélisme et distribution orienté objet
 Comment reproduire les résultats de l article : POP-Java : Parallélisme et distribution orienté objet Beat Wolf 1, Pierre Kuonen 1, Thomas Dandekar 2 1 icosys, Haute École Spécialisée de Suisse occidentale,
Comment reproduire les résultats de l article : POP-Java : Parallélisme et distribution orienté objet Beat Wolf 1, Pierre Kuonen 1, Thomas Dandekar 2 1 icosys, Haute École Spécialisée de Suisse occidentale,
Cours 1. I- Généralités sur R II- Les fonctions de R et autres objets III-Les vecteurs
 Cours 1 I- Généralités sur R II- Les fonctions de R et autres objets III-Les vecteurs IV-Les facteurs I-1 Généralités sur R R (1995, AT&T Bell Laboratories) est un logiciel d analyse statistique et graphique,
Cours 1 I- Généralités sur R II- Les fonctions de R et autres objets III-Les vecteurs IV-Les facteurs I-1 Généralités sur R R (1995, AT&T Bell Laboratories) est un logiciel d analyse statistique et graphique,
Séquence 2. L expression du patrimoine génétique. Sommaire
 Séquence 2 L expression du patrimoine génétique Sommaire 1. La synthèse des protéines 2. Phénotypes, génotypes et environnement Synthèse de la séquence 2 Exercices de la séquence 2 Glossaire des séquences
Séquence 2 L expression du patrimoine génétique Sommaire 1. La synthèse des protéines 2. Phénotypes, génotypes et environnement Synthèse de la séquence 2 Exercices de la séquence 2 Glossaire des séquences
Introduction à MATLAB R
 Introduction à MATLAB R Romain Tavenard 10 septembre 2009 MATLAB R est un environnement de calcul numérique propriétaire orienté vers le calcul matriciel. Il se compose d un langage de programmation, d
Introduction à MATLAB R Romain Tavenard 10 septembre 2009 MATLAB R est un environnement de calcul numérique propriétaire orienté vers le calcul matriciel. Il se compose d un langage de programmation, d
R01 Import de données
 R01 Import de données A partir du package de base, R peut lire les données stockées dans des fichiers texte, à partir notamment des fonctions read.table, read.csv, read.delim, read.csv2, read.delim2, et
R01 Import de données A partir du package de base, R peut lire les données stockées dans des fichiers texte, à partir notamment des fonctions read.table, read.csv, read.delim, read.csv2, read.delim2, et
INTRODUCTION A JAVA. Fichier en langage machine Exécutable
 INTRODUCTION A JAVA JAVA est un langage orienté-objet pur. Il ressemble beaucoup à C++ au niveau de la syntaxe. En revanche, ces deux langages sont très différents dans leur structure (organisation du
INTRODUCTION A JAVA JAVA est un langage orienté-objet pur. Il ressemble beaucoup à C++ au niveau de la syntaxe. En revanche, ces deux langages sont très différents dans leur structure (organisation du
Les concepts de base, l organisation des données
 Chapitre 1 Les concepts de base, l organisation des données Objectif Ce chapitre présente les concepts de base du logiciel R (mode calculatrice, opérateur d affectation, variables, utilisation de fonctions,
Chapitre 1 Les concepts de base, l organisation des données Objectif Ce chapitre présente les concepts de base du logiciel R (mode calculatrice, opérateur d affectation, variables, utilisation de fonctions,
Cours 1 : Introduction Ordinateurs - Langages de haut niveau - Application
 Université de Provence Licence Math-Info Première Année V. Phan Luong Algorithmique et Programmation en Python Cours 1 : Introduction Ordinateurs - Langages de haut niveau - Application 1 Ordinateur Un
Université de Provence Licence Math-Info Première Année V. Phan Luong Algorithmique et Programmation en Python Cours 1 : Introduction Ordinateurs - Langages de haut niveau - Application 1 Ordinateur Un
Gestion des données avec R
 Gestion des données avec R Christophe Lalanne & Bruno Falissard Table des matières 1 Introduction 1 2 Importation de fichiers CSV 1 2.1 Structure du fichier de données...................................
Gestion des données avec R Christophe Lalanne & Bruno Falissard Table des matières 1 Introduction 1 2 Importation de fichiers CSV 1 2.1 Structure du fichier de données...................................
Pourquoi utiliser SharePoint?
 Pourquoi utiliser SharePoint? Partage de Fichiers Accès distant aux informations Mise à jour permanente Gestion électronique de documents (GED) Notifications / Alertes Workflow / Flux de travail Extranet
Pourquoi utiliser SharePoint? Partage de Fichiers Accès distant aux informations Mise à jour permanente Gestion électronique de documents (GED) Notifications / Alertes Workflow / Flux de travail Extranet
Extraction d information des bases de séquences biologiques avec R
 Extraction d information des bases de séquences biologiques avec R 21 novembre 2006 Résumé Le module seqinr fournit des fonctions pour extraire et manipuler des séquences d intérêt (nucléotidiques et protéiques)
Extraction d information des bases de séquences biologiques avec R 21 novembre 2006 Résumé Le module seqinr fournit des fonctions pour extraire et manipuler des séquences d intérêt (nucléotidiques et protéiques)
THEME 1 : L ORDINATEUR ET SON ENVIRONNEMENT. Objectifs
 Architecture Matérielle des Systèmes Informatiques. S1 BTS Informatique de Gestion 1 ère année THEME 1 : L ORDINATEUR ET SON ENVIRONNEMENT Dossier 1 L environnement informatique. Objectifs Enumérer et
Architecture Matérielle des Systèmes Informatiques. S1 BTS Informatique de Gestion 1 ère année THEME 1 : L ORDINATEUR ET SON ENVIRONNEMENT Dossier 1 L environnement informatique. Objectifs Enumérer et
Conventions d écriture et outils de mise au point
 Logiciel de base Première année par alternance Responsable : Christophe Rippert [email protected] Introduction Conventions d écriture et outils de mise au point On va utiliser dans cette
Logiciel de base Première année par alternance Responsable : Christophe Rippert [email protected] Introduction Conventions d écriture et outils de mise au point On va utiliser dans cette
Première session de travail
 Fiche TD avec le logiciel : tdr12 Première session de travail D. Chessel, A.B. Dufour & J.R. Lobry Table des matières 1 Importer des données 2 2 Consulter la documentation 4 3 Le passage des paramètres
Fiche TD avec le logiciel : tdr12 Première session de travail D. Chessel, A.B. Dufour & J.R. Lobry Table des matières 1 Importer des données 2 2 Consulter la documentation 4 3 Le passage des paramètres
Vue d ensemble de la vie microbienne
 Vue d ensemble de la vie microbienne C HAPITRE D EUX I Structure cellulaire et évolution 22 2.1 Les structures cellulaires et virales 22 2.2 L organisation de l ADN dans les cellules microbiennes 24 2.3
Vue d ensemble de la vie microbienne C HAPITRE D EUX I Structure cellulaire et évolution 22 2.1 Les structures cellulaires et virales 22 2.2 L organisation de l ADN dans les cellules microbiennes 24 2.3
Solution de sauvegarde pour flotte nomade
 Solution de sauvegarde pour flotte nomade > PRÉSENTATION D OODRIVE > Les enjeux LA SOLUTION > La solution AdBackup Laptop > Sécurité et options de protection > Monitoring et services > Hébergement (mode
Solution de sauvegarde pour flotte nomade > PRÉSENTATION D OODRIVE > Les enjeux LA SOLUTION > La solution AdBackup Laptop > Sécurité et options de protection > Monitoring et services > Hébergement (mode
Projet d informatique M1BI : Compression et décompression de texte. 1 Généralités sur la compression/décompression de texte
 Projet d informatique M1BI : Compression et décompression de texte Le but de ce projet est de coder un programme réalisant de la compression et décompression de texte. On se proposera de coder deux algorithmes
Projet d informatique M1BI : Compression et décompression de texte Le but de ce projet est de coder un programme réalisant de la compression et décompression de texte. On se proposera de coder deux algorithmes
Cours 3 : Python, les conditions
 Cours 3 : Python, les conditions Conditions Permet d'exécuter des ordres dans certaines conditions : if condition1: code exécuté si condition1 est vraie code exécuté si condition1 est vraie... elif condition2:
Cours 3 : Python, les conditions Conditions Permet d'exécuter des ordres dans certaines conditions : if condition1: code exécuté si condition1 est vraie code exécuté si condition1 est vraie... elif condition2:
1 Introduction - Qu est-ce que le logiciel R?
 Master 1 GSI - Mentions ACCIE et RIM - ULCO, La Citadelle, 2012/2013 Mesures et Analyses Statistiques de Données - Probabilités TP 1 - Initiation au logiciel R 1 Introduction - Qu est-ce que le logiciel
Master 1 GSI - Mentions ACCIE et RIM - ULCO, La Citadelle, 2012/2013 Mesures et Analyses Statistiques de Données - Probabilités TP 1 - Initiation au logiciel R 1 Introduction - Qu est-ce que le logiciel
DIAGNOSTIC BACTERIOLOGIQUE DIRECT D UNE INFECTION
 DIAGNOSTIC BACTERIOLOGIQUE DIRECT D UNE INFECTION Les prélèvements, principales bactéries en cause, interprétation. Maryse ARCHAMBAUD Danielle CLAVE DCEM 1 Laboratoire de Bactériologie-Hygiène Faculté
DIAGNOSTIC BACTERIOLOGIQUE DIRECT D UNE INFECTION Les prélèvements, principales bactéries en cause, interprétation. Maryse ARCHAMBAUD Danielle CLAVE DCEM 1 Laboratoire de Bactériologie-Hygiène Faculté
Plan du cours 2014-2015. Cours théoriques. 29 septembre 2014
 numériques et Institut d Astrophysique et de Géophysique (Bât. B5c) Bureau 0/13 email:[email protected] Tél.: 04-3669771 29 septembre 2014 Plan du cours 2014-2015 Cours théoriques 16-09-2014 numériques pour
numériques et Institut d Astrophysique et de Géophysique (Bât. B5c) Bureau 0/13 email:[email protected] Tél.: 04-3669771 29 septembre 2014 Plan du cours 2014-2015 Cours théoriques 16-09-2014 numériques pour
CAHIER DES CHARGES D IMPLANTATION
 CAHIER DES CHARGES D IMPLANTATION Tableau de diffusion du document Document : Cahier des Charges d Implantation EVRP Version 6 Etabli par DCSI Vérifié par Validé par Destinataires Pour information Création
CAHIER DES CHARGES D IMPLANTATION Tableau de diffusion du document Document : Cahier des Charges d Implantation EVRP Version 6 Etabli par DCSI Vérifié par Validé par Destinataires Pour information Création
LMI 2. Programmation Orientée Objet POO - Cours 9. Said Jabbour. [email protected] www.cril.univ-artois.fr/~jabbour
 LMI 2 Programmation Orientée Objet POO - Cours 9 Said Jabbour [email protected] www.cril.univ-artois.fr/~jabbour CRIL UMR CNRS 8188 Faculté des Sciences - Univ. Artois Février 2011 Les collections
LMI 2 Programmation Orientée Objet POO - Cours 9 Said Jabbour [email protected] www.cril.univ-artois.fr/~jabbour CRIL UMR CNRS 8188 Faculté des Sciences - Univ. Artois Février 2011 Les collections
Contrôle de l'expression génétique :
 Contrôle de l'expression génétique : Les régulations post-transcriptionnelles L'ARNm, simple intermédiaire entre le génome et les protéines? gène protéine L'ARNm, simple intermédiaire entre le génome et
Contrôle de l'expression génétique : Les régulations post-transcriptionnelles L'ARNm, simple intermédiaire entre le génome et les protéines? gène protéine L'ARNm, simple intermédiaire entre le génome et
Table des matières L INTEGRATION DE SAS AVEC JMP. Les échanges de données entre SAS et JMP, en mode déconnecté. Dans JMP
 L INTEGRATION DE SAS AVEC JMP Quelles sont les techniques possibles pour intégrer SAS avec JMP? Comment échanger des données entre SAS et JMP? Comment connecter JMP à SAS? Quels sont les apports d une
L INTEGRATION DE SAS AVEC JMP Quelles sont les techniques possibles pour intégrer SAS avec JMP? Comment échanger des données entre SAS et JMP? Comment connecter JMP à SAS? Quels sont les apports d une
Le langage SQL Rappels
 Le langage SQL Rappels Description du thème : Présentation des principales notions nécessaires pour réaliser des requêtes SQL Mots-clés : Niveau : Bases de données relationnelles, Open Office, champs,
Le langage SQL Rappels Description du thème : Présentation des principales notions nécessaires pour réaliser des requêtes SQL Mots-clés : Niveau : Bases de données relationnelles, Open Office, champs,
IN 102 - Cours 1. 1 Informatique, calculateurs. 2 Un premier programme en C
 IN 102 - Cours 1 Qu on le veuille ou non, les systèmes informatisés sont désormais omniprésents. Même si ne vous destinez pas à l informatique, vous avez de très grandes chances d y être confrontés en
IN 102 - Cours 1 Qu on le veuille ou non, les systèmes informatisés sont désormais omniprésents. Même si ne vous destinez pas à l informatique, vous avez de très grandes chances d y être confrontés en
TD séance n 2c Mise à jour des Systèmes
 1 Gestion des Logiciels 1.1 Introduction sur les logiciels Un logiciel est un programme nécessaire au fonctionnement d'un ordinateur (logiciel système) ou au traitement de données (logiciel applicatif).
1 Gestion des Logiciels 1.1 Introduction sur les logiciels Un logiciel est un programme nécessaire au fonctionnement d'un ordinateur (logiciel système) ou au traitement de données (logiciel applicatif).
Boîte électronique. Découverte des principales fonctions d une messagerie électronique. (et les pièces jointes)
 restosound.mp3 Boîte électronique Découverte des principales fonctions d une messagerie électronique (et les pièces jointes) 1 Rappels Nous avons vu dans l initiation Messagerie 1 les bases du courrier
restosound.mp3 Boîte électronique Découverte des principales fonctions d une messagerie électronique (et les pièces jointes) 1 Rappels Nous avons vu dans l initiation Messagerie 1 les bases du courrier
Séquence 1. Glycémie et diabète. Sommaire
 Séquence 1 Glycémie et diabète Sommaire 1. Prérequis 2. La régulation de la glycémie 3. Le diabète 4. Synthè se 5. Glossaire 6. Exercices Exercices d apprentissage du chapitre 2 Exercices d apprentissage
Séquence 1 Glycémie et diabète Sommaire 1. Prérequis 2. La régulation de la glycémie 3. Le diabète 4. Synthè se 5. Glossaire 6. Exercices Exercices d apprentissage du chapitre 2 Exercices d apprentissage
pas à pas prise en main du logiciel Le Cloud d Orange - Transfert de fichiers sur PC et MAC Le Cloud
 pas à pas prise en main du logiciel Le Cloud d Orange - Transfert de fichiers sur PC et MAC Le Cloud sommaire installer son logiciel PC : 3-9 / MAC : 15-19 définir ses répertoires à surveiller PC : 10-12
pas à pas prise en main du logiciel Le Cloud d Orange - Transfert de fichiers sur PC et MAC Le Cloud sommaire installer son logiciel PC : 3-9 / MAC : 15-19 définir ses répertoires à surveiller PC : 10-12
VidZone, qu est-ce que c est? INSTALLATION DE VidZone
 VidZone, qu est-ce que c est? VidZone, c est le service de musique et vidéo qui vous permet d accéder à des milliers de clips musicaux. VidZone est téléchargeable GRATUITEMENT sur PlayStation Network et
VidZone, qu est-ce que c est? VidZone, c est le service de musique et vidéo qui vous permet d accéder à des milliers de clips musicaux. VidZone est téléchargeable GRATUITEMENT sur PlayStation Network et
1. Structure d'un programme FORTRAN 95
 FORTRAN se caractérise par la nécessité de compiler les scripts, c'est à dire transformer du texte en binaire.(transforme un fichier de texte en.f95 en un executable (non lisible par un éditeur) en.exe.)
FORTRAN se caractérise par la nécessité de compiler les scripts, c'est à dire transformer du texte en binaire.(transforme un fichier de texte en.f95 en un executable (non lisible par un éditeur) en.exe.)
TP n 2 Concepts de la programmation Objets Master 1 mention IL, semestre 2 Le type Abstrait Pile
 TP n 2 Concepts de la programmation Objets Master 1 mention IL, semestre 2 Le type Abstrait Pile Dans ce TP, vous apprendrez à définir le type abstrait Pile, à le programmer en Java à l aide d une interface
TP n 2 Concepts de la programmation Objets Master 1 mention IL, semestre 2 Le type Abstrait Pile Dans ce TP, vous apprendrez à définir le type abstrait Pile, à le programmer en Java à l aide d une interface
Programmation C. Apprendre à développer des programmes simples dans le langage C
 Programmation C Apprendre à développer des programmes simples dans le langage C Notes de cours sont disponibles sur http://astro.u-strasbg.fr/scyon/stusm (attention les majuscules sont importantes) Modalités
Programmation C Apprendre à développer des programmes simples dans le langage C Notes de cours sont disponibles sur http://astro.u-strasbg.fr/scyon/stusm (attention les majuscules sont importantes) Modalités
PARTIE I Compte pour 75 %
 PARTIE I Compte pour 75 % Instructions : Noircissez la lettre correspondant à la bonne réponse sur la feuille de réponse fournie. 1. Dans le diagramme, quelles structures font partie du système nerveux
PARTIE I Compte pour 75 % Instructions : Noircissez la lettre correspondant à la bonne réponse sur la feuille de réponse fournie. 1. Dans le diagramme, quelles structures font partie du système nerveux
AdBackup Laptop. Solution de sauvegarde pour flotte nomade. Société Oodrive www.adbackup-corporatesolutions.com
 AdBackup Laptop Solution de sauvegarde pour flotte nomade Société Oodrive www.adbackup-corporatesolutions.com Sommaire Présentation d Oodrive...3 Carte d identité...3 Références clients...3 Les enjeux...4
AdBackup Laptop Solution de sauvegarde pour flotte nomade Société Oodrive www.adbackup-corporatesolutions.com Sommaire Présentation d Oodrive...3 Carte d identité...3 Références clients...3 Les enjeux...4
CHAPITRE III /Glycémie-Diabète/ Le Diabète
 CHAPITRE III /Glycémie-Diabète/ Le Diabète Problématiques : Comment diagnostique-t-on un diabète chez un patient? Quels sont les mécanismes à l'origine des deux principaux types de diabète? Quels sont
CHAPITRE III /Glycémie-Diabète/ Le Diabète Problématiques : Comment diagnostique-t-on un diabète chez un patient? Quels sont les mécanismes à l'origine des deux principaux types de diabète? Quels sont
MODULE I1. Plan. Introduction. Introduction. Historique. Historique avant 1969. R&T 1ère année. Sylvain MERCHEZ
 MODULE I1 Plan Chapitre 1 Qu'est ce qu'un S.E? Introduction Historique Présentation d'un S.E Les principaux S.E R&T 1ère année Votre environnement Sylvain MERCHEZ Introduction Introduction Rôles et fonctions
MODULE I1 Plan Chapitre 1 Qu'est ce qu'un S.E? Introduction Historique Présentation d'un S.E Les principaux S.E R&T 1ère année Votre environnement Sylvain MERCHEZ Introduction Introduction Rôles et fonctions
CHAPITRE 3 LA SYNTHESE DES PROTEINES
 CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
Sélection du contrôleur
 Démo CoDeSys - 1 - 1. Configuration de l environnement de travail : Lancer le logiciel CoDeSys Fichier Nouveau Lors de la première utilisation, une boîte de dialogue apparaît permettant la sélection du
Démo CoDeSys - 1 - 1. Configuration de l environnement de travail : Lancer le logiciel CoDeSys Fichier Nouveau Lors de la première utilisation, une boîte de dialogue apparaît permettant la sélection du
Info0101 Intro. à l'algorithmique et à la programmation. Cours 3. Le langage Java
 Info0101 Intro. à l'algorithmique et à la programmation Cours 3 Le langage Java Pierre Delisle, Cyril Rabat et Christophe Jaillet Université de Reims Champagne-Ardenne Département de Mathématiques et Informatique
Info0101 Intro. à l'algorithmique et à la programmation Cours 3 Le langage Java Pierre Delisle, Cyril Rabat et Christophe Jaillet Université de Reims Champagne-Ardenne Département de Mathématiques et Informatique
Il est courant de souhaiter conserver à
 Il est courant de souhaiter conserver à la fois Linux et Windows sur un même ordinateur, en particulier pour découvrir Linux, pour garder un filet de sécurité, ou pour continuer à jouer à des jeux récents
Il est courant de souhaiter conserver à la fois Linux et Windows sur un même ordinateur, en particulier pour découvrir Linux, pour garder un filet de sécurité, ou pour continuer à jouer à des jeux récents
Cours 1 : Introduction. Langages objets. but du module. contrôle des connaissances. Pourquoi Java? présentation du module. Présentation de Java
 Langages objets Introduction M2 Pro CCI, Informatique Emmanuel Waller, LRI, Orsay présentation du module logistique 12 blocs de 4h + 1 bloc 2h = 50h 1h15 cours, 45mn exercices table, 2h TD machine page
Langages objets Introduction M2 Pro CCI, Informatique Emmanuel Waller, LRI, Orsay présentation du module logistique 12 blocs de 4h + 1 bloc 2h = 50h 1h15 cours, 45mn exercices table, 2h TD machine page
Fiche technique Schneider
 Fiche technique Schneider Test et mise en service rapide des variateurs ATV11 Gamme : ATV11 Introduction Comment mettre en œuvre le variateur ATV11 pour des applications simples. Comment tester rapidement
Fiche technique Schneider Test et mise en service rapide des variateurs ATV11 Gamme : ATV11 Introduction Comment mettre en œuvre le variateur ATV11 pour des applications simples. Comment tester rapidement
Identification de nouveaux membres dans des familles d'interleukines
 Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
21 mars 2012. Simulations et Méthodes de Monte Carlo. DADI Charles-Abner. Objectifs et intérêt de ce T.E.R. Générer l'aléatoire.
 de 21 mars 2012 () 21 mars 2012 1 / 6 de 1 2 3 4 5 () 21 mars 2012 2 / 6 1 de 2 3 4 5 () 21 mars 2012 3 / 6 1 2 de 3 4 5 () 21 mars 2012 4 / 6 1 2 de 3 4 de 5 () 21 mars 2012 5 / 6 de 1 2 3 4 5 () 21 mars
de 21 mars 2012 () 21 mars 2012 1 / 6 de 1 2 3 4 5 () 21 mars 2012 2 / 6 1 de 2 3 4 5 () 21 mars 2012 3 / 6 1 2 de 3 4 5 () 21 mars 2012 4 / 6 1 2 de 3 4 de 5 () 21 mars 2012 5 / 6 de 1 2 3 4 5 () 21 mars
SSL ET IPSEC. Licence Pro ATC Amel Guetat
 SSL ET IPSEC Licence Pro ATC Amel Guetat LES APPLICATIONS DU CHIFFREMENT Le protocole SSL (Secure Socket Layer) La sécurité réseau avec IPSec (IP Security Protocol) SSL - SECURE SOCKET LAYER Historique
SSL ET IPSEC Licence Pro ATC Amel Guetat LES APPLICATIONS DU CHIFFREMENT Le protocole SSL (Secure Socket Layer) La sécurité réseau avec IPSec (IP Security Protocol) SSL - SECURE SOCKET LAYER Historique
ET 24 : Modèle de comportement d un système Boucles de programmation avec Labview.
 ET 24 : Modèle de comportement d un système Boucles de programmation avec Labview. Sciences et Technologies de l Industrie et du Développement Durable Formation des enseignants parcours : ET24 Modèle de
ET 24 : Modèle de comportement d un système Boucles de programmation avec Labview. Sciences et Technologies de l Industrie et du Développement Durable Formation des enseignants parcours : ET24 Modèle de
Cours d initiation à la programmation en C++ Johann Cuenin
 Cours d initiation à la programmation en C++ Johann Cuenin 11 octobre 2014 2 Table des matières 1 Introduction 5 2 Bases de la programmation en C++ 7 3 Les types composés 9 3.1 Les tableaux.............................
Cours d initiation à la programmation en C++ Johann Cuenin 11 octobre 2014 2 Table des matières 1 Introduction 5 2 Bases de la programmation en C++ 7 3 Les types composés 9 3.1 Les tableaux.............................
Introduction à R. Florence Yerly. Dept. de mathématiques, Université de Fribourg (CH) SP 2011
 Dept. de mathématiques, Université de Fribourg (CH) SP 2011 Qu est ce que R? Un logiciel de statistiques libre et gratuit ; Un logiciel multi-plateforme (UNIX, Windows MacOS X) R permet de faire des calculs
Dept. de mathématiques, Université de Fribourg (CH) SP 2011 Qu est ce que R? Un logiciel de statistiques libre et gratuit ; Un logiciel multi-plateforme (UNIX, Windows MacOS X) R permet de faire des calculs
Contrôle Parental Numericable. Guide d installation et d utilisation
 Contrôle Parental Numericable Guide d installation et d utilisation Version 12.3 pour OS X Copyright 2012 Xooloo. Tous droits réservés. Table des matières 1. Introduction Dénomination et caractéristiques
Contrôle Parental Numericable Guide d installation et d utilisation Version 12.3 pour OS X Copyright 2012 Xooloo. Tous droits réservés. Table des matières 1. Introduction Dénomination et caractéristiques
La salle des marchés. Version 13.7.0 et supérieures. Manuel Entreprises - Spécifique CRE
 Solution de dématérialisation des procédures de marchés publics La salle des marchés Version 13.7.0 et supérieures Manuel Entreprises - Spécifique CRE Répondre sur la plateforme de réponse aux appels d
Solution de dématérialisation des procédures de marchés publics La salle des marchés Version 13.7.0 et supérieures Manuel Entreprises - Spécifique CRE Répondre sur la plateforme de réponse aux appels d
Bases moléculaires des mutations Marc Jeanpierre
 Bases moléculaires des mutations Marc Jeanpierre Chaque enfant qui naît hérite de 10 à 30 nouvelles mutations ponctuelles. L essentiel des ces mutations sont heureusement des variations neutres de séquence
Bases moléculaires des mutations Marc Jeanpierre Chaque enfant qui naît hérite de 10 à 30 nouvelles mutations ponctuelles. L essentiel des ces mutations sont heureusement des variations neutres de séquence
as Architecture des Systèmes d Information
 Plan Plan Programmation - Introduction - Nicolas Malandain March 14, 2005 Introduction à Java 1 Introduction Présentation Caractéristiques Le langage Java 2 Types et Variables Types simples Types complexes
Plan Plan Programmation - Introduction - Nicolas Malandain March 14, 2005 Introduction à Java 1 Introduction Présentation Caractéristiques Le langage Java 2 Types et Variables Types simples Types complexes
Ricco Rakotomalala http://eric.univ-lyon2.fr/~ricco/cours/cours_programmation_r.html. R.R. Université Lyon 2
 Ricco Rakotomalala http://eric.univ-lyon2.fr/~ricco/cours/cours_programmation_r.html 1 Plan de présentation 1. L écosystème Hadoop 2. Principe de programmation MapReduce 3. Programmation des fonctions
Ricco Rakotomalala http://eric.univ-lyon2.fr/~ricco/cours/cours_programmation_r.html 1 Plan de présentation 1. L écosystème Hadoop 2. Principe de programmation MapReduce 3. Programmation des fonctions
Architecture des ordinateurs
 Architecture des ordinateurs Cours 7 17 décembre 2012 Archi 1/1 Fonctions, suite et fin Archi 2/1 Rappel Forme générale d un sous-programme (fonction) : etiquette sousprogramme: push ebp ; empile la valeur
Architecture des ordinateurs Cours 7 17 décembre 2012 Archi 1/1 Fonctions, suite et fin Archi 2/1 Rappel Forme générale d un sous-programme (fonction) : etiquette sousprogramme: push ebp ; empile la valeur
Questionnaire après (exemple)
 Questionnaire après (exemple) Questionnaire après formation destiné aux collaborateurs de (nom de l'établissement) Bonjour, Comme nous vous l avions annoncé, voici un deuxième questionnaire qui a pour
Questionnaire après (exemple) Questionnaire après formation destiné aux collaborateurs de (nom de l'établissement) Bonjour, Comme nous vous l avions annoncé, voici un deuxième questionnaire qui a pour
Manuel utilisateur. Version 1.6b
 Manuel utilisateur Version 1.6b Table des matières Table des matières... 2 1. Introduction... 3 a. But de ce document... 3 b. Objet de ce document... 3 c. Remarques et commentaires... 3 2. Premiers pas
Manuel utilisateur Version 1.6b Table des matières Table des matières... 2 1. Introduction... 3 a. But de ce document... 3 b. Objet de ce document... 3 c. Remarques et commentaires... 3 2. Premiers pas
Analyse de sécurité de logiciels système par typage statique
 Contexte Modélisation Expérimentation Conclusion Analyse de sécurité de logiciels système par typage statique Application au noyau Linux Étienne Millon UPMC/LIP6 Airbus Group Innovations Sous la direction
Contexte Modélisation Expérimentation Conclusion Analyse de sécurité de logiciels système par typage statique Application au noyau Linux Étienne Millon UPMC/LIP6 Airbus Group Innovations Sous la direction
Programmation en Java IUT GEII (MC-II1) 1
 Programmation en Java IUT GEII (MC-II1) 1 Christophe BLANC - Paul CHECCHIN IUT Montluçon Université Blaise Pascal Novembre 2009 Christophe BLANC - Paul CHECCHIN Programmation en Java IUT GEII (MC-II1)
Programmation en Java IUT GEII (MC-II1) 1 Christophe BLANC - Paul CHECCHIN IUT Montluçon Université Blaise Pascal Novembre 2009 Christophe BLANC - Paul CHECCHIN Programmation en Java IUT GEII (MC-II1)
CHAPITRE V SYSTEMES DIFFERENTIELS LINEAIRES A COEFFICIENTS CONSTANTS DU PREMIER ORDRE. EQUATIONS DIFFERENTIELLES.
 CHAPITRE V SYSTEMES DIFFERENTIELS LINEAIRES A COEFFICIENTS CONSTANTS DU PREMIER ORDRE EQUATIONS DIFFERENTIELLES Le but de ce chapitre est la résolution des deux types de systèmes différentiels linéaires
CHAPITRE V SYSTEMES DIFFERENTIELS LINEAIRES A COEFFICIENTS CONSTANTS DU PREMIER ORDRE EQUATIONS DIFFERENTIELLES Le but de ce chapitre est la résolution des deux types de systèmes différentiels linéaires
WINDOWS Remote Desktop & Application publishing facile!
 Secure Cloud & Solutions Accès BOYD CLOUD acces informatiques & BYOD sécurisé MYRIAD-Connect facilite votre travail en tous lieux et à tous moments comme si vous étiez au bureau. Conçu pour vous simplifier
Secure Cloud & Solutions Accès BOYD CLOUD acces informatiques & BYOD sécurisé MYRIAD-Connect facilite votre travail en tous lieux et à tous moments comme si vous étiez au bureau. Conçu pour vous simplifier
www.netexplorer.fr [email protected]
 www.netexplorer.fr 05 61 61 20 10 [email protected] Sommaire Sécurité applicative... 3 Authentification... 3 Chiffrement... 4 Traçabilité... 4 Audits... 5 Sécurité infrastructure... 6 Datacenters...
www.netexplorer.fr 05 61 61 20 10 [email protected] Sommaire Sécurité applicative... 3 Authentification... 3 Chiffrement... 4 Traçabilité... 4 Audits... 5 Sécurité infrastructure... 6 Datacenters...
Conferencing Services. Web Meeting. Guide de démarrage rapide V5_FR
 Web Meeting Guide de démarrage rapide V5_FR Table des matières 1 INFORMATION PRODUIT... 3 1.1 GENERALITES... 3 1.2 SYSTEMES D EXPLOITATION ET LANGUES PRIS EN CHARGE... 3 1.3 FONCTIONS... 3 2 RESERVER UNE
Web Meeting Guide de démarrage rapide V5_FR Table des matières 1 INFORMATION PRODUIT... 3 1.1 GENERALITES... 3 1.2 SYSTEMES D EXPLOITATION ET LANGUES PRIS EN CHARGE... 3 1.3 FONCTIONS... 3 2 RESERVER UNE
Séquencer une application
 Séquencer une application Pour ma part, j'utilise Vmware workstation pour créer les packages. Je configure une VM sous Windows Xp Pro Sp2 : 1. Mise du Pc dans le domaine 2. Installation du séquenceur 3.
Séquencer une application Pour ma part, j'utilise Vmware workstation pour créer les packages. Je configure une VM sous Windows Xp Pro Sp2 : 1. Mise du Pc dans le domaine 2. Installation du séquenceur 3.
MICROBIOLOGIE. 1. Strep A et Urine Slide. 2. Coloration de Gram 3. Virologie (HCV, HBV, HIV)
 MICROBIOLOGIE 1. Strep A et Urine Slide (page 1) 2. Coloration de Gram (page 1) 3. Virologie (HCV, HBV, HIV) (page 1) 4. UKNEQAS Microbiology (page 2) 5. QCMD (page 4) Les programmes 1, 2 et 3 sont organisés
MICROBIOLOGIE 1. Strep A et Urine Slide (page 1) 2. Coloration de Gram (page 1) 3. Virologie (HCV, HBV, HIV) (page 1) 4. UKNEQAS Microbiology (page 2) 5. QCMD (page 4) Les programmes 1, 2 et 3 sont organisés
SoMachine. Solution logicielle pour votre architecture Machine Atelier de découverte. SoMachine
 Solution logicielle pour votre architecture Machine Atelier de découverte SoMachine Objectif de la manipulation Le programme à réaliser va permettre de gérer le remplissage et la vidange d une cuve en
Solution logicielle pour votre architecture Machine Atelier de découverte SoMachine Objectif de la manipulation Le programme à réaliser va permettre de gérer le remplissage et la vidange d une cuve en
Programmation linéaire
 1 Programmation linéaire 1. Le problème, un exemple. 2. Le cas b = 0 3. Théorème de dualité 4. L algorithme du simplexe 5. Problèmes équivalents 6. Complexité de l Algorithme 2 Position du problème Soit
1 Programmation linéaire 1. Le problème, un exemple. 2. Le cas b = 0 3. Théorème de dualité 4. L algorithme du simplexe 5. Problèmes équivalents 6. Complexité de l Algorithme 2 Position du problème Soit
26 Centre de Sécurité et de
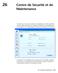 26 Centre de Sécurité et de Maintenance La fenêtre du Centre de sécurité et de maintenance (CSM) rassemble tous les outils nécessaires au contrôle, à l analyse, à la maintenance, à la sauvegarde et au
26 Centre de Sécurité et de Maintenance La fenêtre du Centre de sécurité et de maintenance (CSM) rassemble tous les outils nécessaires au contrôle, à l analyse, à la maintenance, à la sauvegarde et au
Accès à un coupleur/contrôleur Ethernet via une liaison téléphonique
 Accès à un coupleur/contrôleur Ethernet via une liaison téléphonique, Français Version 1.0.0 23/12/2002 ii General Copyright 2001 by WAGO Kontakttechnik GmbH Tous droits réservés. WAGO Kontakttechnik GmbH
Accès à un coupleur/contrôleur Ethernet via une liaison téléphonique, Français Version 1.0.0 23/12/2002 ii General Copyright 2001 by WAGO Kontakttechnik GmbH Tous droits réservés. WAGO Kontakttechnik GmbH
Plan du cours. Historique du langage http://www.oracle.com/technetwork/java/index.html. Nouveautés de Java 7
 Université Lumière Lyon 2 Faculté de Sciences Economiques et Gestion KHARKIV National University of Economic Introduction au Langage Java Master Informatique 1 ère année Julien Velcin http://mediamining.univ-lyon2.fr/velcin
Université Lumière Lyon 2 Faculté de Sciences Economiques et Gestion KHARKIV National University of Economic Introduction au Langage Java Master Informatique 1 ère année Julien Velcin http://mediamining.univ-lyon2.fr/velcin
Cours 1 : La compilation
 /38 Interprétation des programmes Cours 1 : La compilation Yann Régis-Gianas [email protected] PPS - Université Denis Diderot Paris 7 2/38 Qu est-ce que la compilation? Vous avez tous déjà
/38 Interprétation des programmes Cours 1 : La compilation Yann Régis-Gianas [email protected] PPS - Université Denis Diderot Paris 7 2/38 Qu est-ce que la compilation? Vous avez tous déjà
Introduction aux Machines Virtuelles avec VMKit
 Introduction aux Machines Virtuelles avec Ingénieur :, Responsables : Gaël Thomas, Gilles Müller EQUIPE REGAL LIP6 INRIA (Paris/France) un substrat de machine virtuelle : a substrate for Managed Runtime
Introduction aux Machines Virtuelles avec Ingénieur :, Responsables : Gaël Thomas, Gilles Müller EQUIPE REGAL LIP6 INRIA (Paris/France) un substrat de machine virtuelle : a substrate for Managed Runtime
Insulinothérapie et diabète de type 1
 Insulinothérapie et diabète de type 1 Introduction: la molécule d insuline L instauration de l insulinothérapie Dispositif d administration de l insuline Les propriétés de l insuline Insuline et schémas
Insulinothérapie et diabète de type 1 Introduction: la molécule d insuline L instauration de l insulinothérapie Dispositif d administration de l insuline Les propriétés de l insuline Insuline et schémas
Cours intensif Java. 1er cours: de C à Java. Enrica DUCHI LIAFA, Paris 7. Septembre 2009. [email protected]
 . Cours intensif Java 1er cours: de C à Java Septembre 2009 Enrica DUCHI LIAFA, Paris 7 [email protected] LANGAGES DE PROGRAMMATION Pour exécuter un algorithme sur un ordinateur il faut le
. Cours intensif Java 1er cours: de C à Java Septembre 2009 Enrica DUCHI LIAFA, Paris 7 [email protected] LANGAGES DE PROGRAMMATION Pour exécuter un algorithme sur un ordinateur il faut le
Bases de données des mutations
 Bases de données des mutations CFMDB CFTR2 CFTR-France / Registre Corinne THEZE, Corinne BAREIL Laboratoire de génétique moléculaire Montpellier Atelier Muco, Lille, 25-27 septembre 2014 Accès libre http://www.genet.sickkids.on.ca/app
Bases de données des mutations CFMDB CFTR2 CFTR-France / Registre Corinne THEZE, Corinne BAREIL Laboratoire de génétique moléculaire Montpellier Atelier Muco, Lille, 25-27 septembre 2014 Accès libre http://www.genet.sickkids.on.ca/app
Structurer ses données : les tableaux. Introduction à la programmation
 Structurer ses données : les tableaux Introduction à la programmation Plan du document Introduction Introduire la notion de type Types primitifs Types composés Tableaux de type primitif Page 2 Notion de
Structurer ses données : les tableaux Introduction à la programmation Plan du document Introduction Introduire la notion de type Types primitifs Types composés Tableaux de type primitif Page 2 Notion de
Les débuts de la génétique
 HPITRE 9 DES DÉBTS DE L ÉNÉTIQE X ENJEX TELS DES BIOTEHNOLOIES 1 Les débuts de la génétique est avec les travaux de regor Mendel vers la fin du XIX e siècle que furent posées les bases de la génétique.
HPITRE 9 DES DÉBTS DE L ÉNÉTIQE X ENJEX TELS DES BIOTEHNOLOIES 1 Les débuts de la génétique est avec les travaux de regor Mendel vers la fin du XIX e siècle que furent posées les bases de la génétique.
Machines virtuelles Cours 1 : Introduction
 Machines virtuelles Cours 1 : Introduction Pierre Letouzey 1 [email protected] PPS - Université Denis Diderot Paris 7 janvier 2012 1. Merci à Y. Régis-Gianas pour les transparents Qu est-ce qu une
Machines virtuelles Cours 1 : Introduction Pierre Letouzey 1 [email protected] PPS - Université Denis Diderot Paris 7 janvier 2012 1. Merci à Y. Régis-Gianas pour les transparents Qu est-ce qu une
Architecture des ordinateurs Introduction à l informatique
 Architecture des ordinateurs Introduction à l informatique 17 septembre 2004 1 2 3 4 5 6 Les interrupteurs... 0V 5V Ce sont des composants électroniques qui laissent pser un courant principal lorsque la
Architecture des ordinateurs Introduction à l informatique 17 septembre 2004 1 2 3 4 5 6 Les interrupteurs... 0V 5V Ce sont des composants électroniques qui laissent pser un courant principal lorsque la
Plan 1/9/2013. Génération et exploitation de données. CEP et applications. Flux de données et notifications. Traitement des flux Implémentation
 Complex Event Processing Traitement de flux de données en temps réel Romain Colle R&D Project Manager Quartet FS Plan Génération et exploitation de données CEP et applications Flux de données et notifications
Complex Event Processing Traitement de flux de données en temps réel Romain Colle R&D Project Manager Quartet FS Plan Génération et exploitation de données CEP et applications Flux de données et notifications
Un exemple de régression logistique sous
 Fiche TD avec le logiciel : tdr341 Un exemple de régression logistique sous A.B. Dufour & A. Viallefont Etude de l apparition ou non d une maladie cardiaque des coronaires 1 Présentation des données Les
Fiche TD avec le logiciel : tdr341 Un exemple de régression logistique sous A.B. Dufour & A. Viallefont Etude de l apparition ou non d une maladie cardiaque des coronaires 1 Présentation des données Les
Manuel. Nero MediaHome. Nero AG
 Manuel Nero MediaHome Nero AG Informations sur le copyright et les marques Le manuel de Nero MediaHome et son contenu sont protégés par copyright et sont la propriété de Nero AG. Tous droits réservés.
Manuel Nero MediaHome Nero AG Informations sur le copyright et les marques Le manuel de Nero MediaHome et son contenu sont protégés par copyright et sont la propriété de Nero AG. Tous droits réservés.
Ce guide détaille pas à pas les étapes d installation et de configuration de la solution jusqu'à la sauvegarde des BDD.
 Le présent guide est le fruit de mon travail en tant que stagiaire au sein d un Hôpital, dont l objectif était l élaboration d une offre d inventaire et d un Helpdesk. Ce guide détaille pas à pas les étapes
Le présent guide est le fruit de mon travail en tant que stagiaire au sein d un Hôpital, dont l objectif était l élaboration d une offre d inventaire et d un Helpdesk. Ce guide détaille pas à pas les étapes
Compte Rendu. Projet Reseaux : Conception Implémentation d un Mini DataCenter
 Compte Rendu Projet Reseaux : Conception Implémentation d un Mini DataCenter Etudiant Encadrant M. Jake MORISON Dr.Olivier RICHARD M. Eric Michel FOTSING Dr Didier DONSEZ Avril 2015 1. SOMMAIRE 1. Sommaire...
Compte Rendu Projet Reseaux : Conception Implémentation d un Mini DataCenter Etudiant Encadrant M. Jake MORISON Dr.Olivier RICHARD M. Eric Michel FOTSING Dr Didier DONSEZ Avril 2015 1. SOMMAIRE 1. Sommaire...
Solution d inventaire automatisé d un parc informatique et de télédistribution OCS INVENTORY NG. EHRHARD Eric - Gestionnaire Parc Informatique
 Solution d inventaire automatisé d un parc informatique et de télédistribution OCS INVENTORY NG EHRHARD Eric - Gestionnaire Parc Informatique 1 Possibilités d OCS Inventory. Informations d'inventaire pertinentes.
Solution d inventaire automatisé d un parc informatique et de télédistribution OCS INVENTORY NG EHRHARD Eric - Gestionnaire Parc Informatique 1 Possibilités d OCS Inventory. Informations d'inventaire pertinentes.
Accéder à ZeCoffre via FTP
 Accéder à ZeCoffre via FTP Table des matières Introduction... 2 Qu est-ce que FTP... 3 Comment se connecter à ZeCoffre via FTP... 4 Connexion FTP depuis Windows / Linux / Mac OS X... 5 Connexion FTP depuis
Accéder à ZeCoffre via FTP Table des matières Introduction... 2 Qu est-ce que FTP... 3 Comment se connecter à ZeCoffre via FTP... 4 Connexion FTP depuis Windows / Linux / Mac OS X... 5 Connexion FTP depuis
Procédure d installation d AMESim 4.3.0
 Procédure d installation d AMESim 4.3.0 B2i Automotive Yahia BOUZID 18/07/2006 SOMMAIRE SOMMAIRE... 2 LISTE DES FIGURES... 3 HISTORIQUE DES MODIFICATIONS... 4 1. INTRODUCTION... 5 1.1. OBJET DU DOCUMENT...
Procédure d installation d AMESim 4.3.0 B2i Automotive Yahia BOUZID 18/07/2006 SOMMAIRE SOMMAIRE... 2 LISTE DES FIGURES... 3 HISTORIQUE DES MODIFICATIONS... 4 1. INTRODUCTION... 5 1.1. OBJET DU DOCUMENT...
Compléments de documentation Scilab : affichage de texte et formatage de nombres
 Université des Sciences et Technologies de Lille U.F.R. de Mathématiques Pures et Appliquées Agrégation externe Année 2002-2003 Compléments de documentation Scilab : affichage de texte et formatage de
Université des Sciences et Technologies de Lille U.F.R. de Mathématiques Pures et Appliquées Agrégation externe Année 2002-2003 Compléments de documentation Scilab : affichage de texte et formatage de
Enregistrer votre dispositif
 Guide de Commencement Rapide: NotifyLink pour Symbian Series 60, 3 rd Edition Votre appareil aura besoin d un des suivants: Connection cellulaire capable de soutenir la transmission des données via votre
Guide de Commencement Rapide: NotifyLink pour Symbian Series 60, 3 rd Edition Votre appareil aura besoin d un des suivants: Connection cellulaire capable de soutenir la transmission des données via votre
Publipostage et étiquettes
 Publipostage et étiquettes avec une base de données Calc Réalisé avec : LibreOffice 4.1.5 par Dominique MILLET Plate-forme / Os : Toutes www.atem-formation.com 05 63 47 98 36 Table des matières 1 Présentation...3
Publipostage et étiquettes avec une base de données Calc Réalisé avec : LibreOffice 4.1.5 par Dominique MILLET Plate-forme / Os : Toutes www.atem-formation.com 05 63 47 98 36 Table des matières 1 Présentation...3
Apps Sage : les 10 étapes pour publier vos données dans le Cloud.
 Apps Sage : les 10 étapes pour publier vos données dans le Cloud. Produits concernés : Sage Customer View et Sage Reports Contexte Depuis Sage Data Manager, vous venez d installer Sage Business Sync (cf
Apps Sage : les 10 étapes pour publier vos données dans le Cloud. Produits concernés : Sage Customer View et Sage Reports Contexte Depuis Sage Data Manager, vous venez d installer Sage Business Sync (cf
