Bioinformatique structurale : Un enjeu de l après génome IN Tech Lyon le 23/10/2003
|
|
|
- Lucille Champagne
- il y a 8 ans
- Total affichages :
Transcription
1 Bioinformatique structurale : Un enjeu de l après génome IN Tech Lyon le 23/10/2003 Gilbert DELEAGE PBIL-IBCP-CNRS UMR 5086 Pôle Bioinformatique Lyonnais Lyon-Gerland 7, passage du Vercors Lyon cedex 07 Tél: +33 (0) fax: +33 (0) mel: g.deleage@ibcp. fr
2 Equipe Bioinformatique et RMN structurales Permanents DELEAGE G. PR1 UCBL, Bioinformatique PENIN F. IR2-CNRS, NMR, Biophysique, Biochimie BETTLER E. MC2, Bioinformatique BLANCHET C. IR2-CNRS, Bioinformatique BOCKMANN A. CR2-CNRS, NMR COMBET C. CR2-CNRS Bioinformatique GEOURJON C. IR2-CNRS, Bioinformatique HUET E. CR2-CNRS, Biologie-Biochimie LAVERGNE J-P CR1-CNRS, Biochimie MONTSERRET R. IE2-CNRS, NMR, Biophysique Post-docs and CDD DORKELD F. (EC), Bioinformatique LECLUSE A. (French ministry) Bioinformatique LACORNE N. (ACI GRID) Bioinformatique MISSEREY S. (Genopôle Rhône-Alpes) Bioinformatique Etudiants BOULANT S. Biochimie GIRAUD N. NMR RATINIER M. Biochimie SAPAY N. Bioinformatique VERNOIS A. GRID computing, Informatique PBIL-IBCP
3 La révolution génomique - Une stratégie revisitée Avant la Bio-informatique Relations Activité Biologique connue Etude Biochimique Structure 3D Séquence Protéine Gène Mutagénèse structureactivité BIO-INFORMATIQUE Bases de données Prédiction des gènes Identification de protéines Prédiction sites/signatures Prédiction de structure Modélisation moléculaire Stockage Classification Intégration Criblage Protéomique Séquences génomiques Séquences Protéiques Prédiction Activités biologiques Etudes Biochimiques Structures 3D Aujourd hui (depuis les programmes de séquençages massifs et la Bio-informatique) Génomique structurale
4 Réalisations bioinformatiques du groupe PBIL-IBCP odatabase : o HCVDB : Base de données de séquences annotées d HCV; o Méthodologies : o MLRC : Prédiction de la structure secondaire des protéines (Guermeur et al., Bioinformatics, 1999) Coll. LIP6 o ProScan : Scan PROSITE avec recherche pondérée par un système original o PattInProt : Algorithme de recherche original de signatures dans les banques. Outil de PROTEOMIQUE o PROCSS : Compatibilité des structures protéiques grâce aux structures secondaires (Geourjon et al., Protein Sci.,2001; Errami et al., Bioinformatics, 2003) o SOPM :Méthode auto-optimisée de prédiction de structure secondaire (Geourjon et al., Protein Eng., 1994) o SOPMA : Méthode auto-optimisée de prédiction de structure secondaire avec alignements (Geourjon et al., Comput Appl Biosci., 1995) o SuMo : Détection de sites 3D dans les protéines (Jambon et al., Proteins, 2003, Brevet international) ologiciels : o AnTheProt : Logiciel intégré d analyse de séquences client/serveur (Deléage et al., Comput Appl Biosci., 1988; Deléage et al., Comput Biol Med., 2001; o AnTheNuc : Analyse de sites de restriction o Bioread : Interface graphique pour extractblast and extractfasta programmes parser. o DicroProt : Analyse des spectres de dichroïsme circulaire des protéines (Deléage G et Geourjon C.Comp. Appl. Biosc., 1993) o MPSA : integrated protein sequence analysis with client/server capabilities (Blanchet et al., Bioinformatics, 2000; o SecTrace : secondary structure plot o Services Web: o NPS@ : Analyse intégrée de séquences sur le Web (Combet et al., Trends Biochem Sci, 2000; Perrière, Combet et al., Nucleic Acids Res., 2003; o Geno3d : Modélisation moléculaire de protéines à grande échelle (Combet et al., Bioinformatics, 2002; o SuMo : protein common 3D sites detection (Jambon et al., Proteins, 2003; oapplications biologiques: o Très nombreuses en collaboration avec des biologistes
5 Portail de bioinformatique : Webiciel - NPS@ NPS@ Partie «protéine et structure» du Pôle BioInformatique Lyonnais Interconnexion de 46 méthodes d analyse de séquences de protéine Récupération automatique des données dans des logiciels clients/serveurs d analyse biologique MPSA, AnTheProt, Clustal X, RasMol, Liens hypertextes sur les données de 17 banques de données internationales (SWISS-PROT, PROSITE, PDB, SCOP, ). Mise à jour automatique des bases de données disponibles sur le serveur (SP, SP-TrEMBL, NRL3D, Nr, PDB, PROSITE, etc.). Références internationales: Expasy, University of California, InfoBioGen, RSCB(PDB), analyses / jour en analyses / jour depuis janvier 2003 France 24% Oceania 2% Europa 36% Africa 1% Combet et al., Trends Biochem Sci, 2000; Perrière et al., Nucleic Acids Res., 2003 America 25% Asia 12%
6 Projets bioinformatique en cours o GRID computing : o GPS@ : Portail d analysesde séquences de protéines sur la Grille (Déploiement de NPS@ sur la grille, EU FP5 DATAGRID) o GriPPS : Services de recherche de signatures sur la grille (Ministère rehcerche ACI-GRID, CNRS-IN2P3, ENSL-LIP) o RUGBI : Prédictions de structures secondaires sur la grille GRID (Ministère rehcerche et CNRS-IN2P3 (V. BRETON), Biopôle Clermont-Limagne, CS, ECP) o e-toile : Projet de grille expérimentale (Ministère rehcerche RNTL) o GiGn : Réseau de recherche français de GrIlle pour la GeNomique (Ministère rehcerche Action IMPG, with V. BRETON) o HealthGrid : Organisation de conférences (HealthGrid Conference, Lyon FR, January 2003) o Bioinformatique structurale et clinique : o CPS@ : Version pour cliuster de NPS@ (PBS, PVM and MPI algorithms) o euhcvdb : Base européenne de séquences HCV (EU FP5 HepCVax QLK2-CT ; EU FP6 virgil) o Collaborarion avec D. KAHN (PRODOM) o Améliorationdes domaines PRODOM grâce aux prédictions de structures o Connexion avec Geno3D ostrucannot Annotation structurale de genome Arabidopsis thaliana : (consortium France, Belgium (P. ROUZE, Gent) and SIB (A. BAIROCH, Geneva)) o MSFold : Modélisation de structure 3D à partir des contraintes de MS. Protéomique structurale, (coll. E. Forest, IBS, Grenoble) o MADPROTS : Modélisation, analyses et serveur Web pour amarrage moléculaire o SYNAPSE : Système expert d annotation de protéines
7 Organisation hiérarchique des protéines Structure primaire = séquence= mot écrit avec un alphabet de 20 lettres MKLDEIARLAGVSRTTASYVINGKAKQYRVSDKTVEKVMAVVREHNYHPNAVAAGLRAGR Structure secondaire Brins β Hélice α Structure secondaire = mot alphabet de 3 à 10 lettres CCHHHHHHHHHHHCCCEEEETTTTEEEEECCCCHHHHHHHHHHHCCHHHHHHHHHGCCCC Structure tertiaire = objet 3D Fonction
8 Relation identité de séquence ressemblance structurale 1AJJ : LDL receptor 1CR8 : Low Density Lipoprotein Receptor Related Protein pdb1ajj.ent P--CSAFEFHC-LSGECIHSSWRCDGGPDCKDKSDEENCA pdb1cr8.ent PGGCHTDEFQCRLDGLCIPLRWRCDGDTDCMDSSDEKSCEGV 42 Identity * * : **:* *.* ** *****..** *.***:.* 100 Ssearch (E=10) Pourcentage de protéines ressemblantes (%) Identité de séquence (%) Nécessité de discriminer dans l intervalle 10-30% d identité
9 Utilité des prédictions de structure secondaire Sov = 81 15,9% identité ; Protéines apparentées (FSSP : RMSD = 2,3A ; Z-score = 19,9) 1auq 1ido 16% identité ; Protéines non apparentées Sov = 9 1ai7-A 1jac-A helix α β sheet Les structures secondaires permettent de discriminer les protéines ayant des structures 3D proches «zone floue» de 10-30% (Geourjon et al., Protein Sci.,2001; Errami et al., Bioinformatics, 2003)
10 Prédiction de structure 3D Modélisation homologique «classique» 2 protéines qui ont plus de 30% d'identité de séquences ont 80% de leurs Cα superposables avec un écart quadratique moyen de 1 Å (RMSD=1Å) Modélisation par analogie (à faible taux d identité) 2 protéines qui ont la même fonction et une topologie «probablement» identique en dépit de l'absence de similarité importante (arguments expérimentaux ou de structures secondaires). Threading (en cours de développement) Une séquence est testée sur une librairie de repliements pour déterminer sa compatibilité structure-séquence probable, méthode d alignement séquencestructures tridimensionnelles. Ab initio (en progression dans CASP5, folding@home Stanford) Structure directement déduite de la séquence à partir de règles empiriques.
11 Modélisation moléculaire via le Serveur Web Geno3D Protein sequence MKLDEIARLAGVSRTTASYVINGKAKQYRVSDKTVEKVMAVVREHNYHPNAVAAGLRAGR Secondary structures Geno3 α Helix Tools Β strands Combet C., Jambon M., Deléage G. et Geourjon, C. (2002) Bioinformatics, 18,
12 3D-Crunch : Modélisation à grande échelle Stratégie Modélisation à faible taux d identité (entre 10-35%) 1,315 protéines de structures 3D connues (pdb 25%) PSI-BLAST sur chaque entrée (max 5 run). Les 5,390 protéines ayant un pourcentage d indentité entre 10 et 35% ont été modélisées Les calculs ont été réalisés au CC-IN 2 P 3 Villeurbanne (France) 7,881 heures de CPU Number of models RMSD (A) Z-score
13 Apport des prédictions de structures secondaires Sans Sov 70,00 60,00 50,00 40,00 Identité de séquences 10-35% 10-20% 20-35% ,00 20,00 10,00 0,00 rmsd>5 5<rmsd>4 4<rmsd>3 3<rmsd>2 2<rmsd 70, ,00 50, Avec Sov >60% 40,00 30,00 20,00 10,00 0,00 Rmsd 1> 5 5<rmsd>4 2 4<rmsd>3 3 3<rmsd>2 4 2<rmsd 5
14 Geno3D : Bilan Bonne fiabilité y compris à bas taux d identité Ne pas introduire d a priori au niveau des «gaps» et insertions dans l alignement. Possibilité d avoir une estimation de la qualité du modèle obtenu. Possibilité de combiner des données provenant de protéines proches (séquences ou fonctions) mais aussi des données expérimentales. Possibilité de modéliser des dimères ou trimères Possibilité de modéliser des protéines par domaines Possibilité d inclure les ligands (géométriquement puis minimisation) Disponible également sur serveur sécurisé Contrat de collaboration avec des industriels Utilisation dans le cadre de modélisation moléculaire à grande échelle Protéome complet Arabidopsis thaliana. Programme GENOPLANTE
15 Problème 1 : séquences non alignables, repliement différent, même fonction, même site actif Subtilisine Protéases à sérine Chymotrypsine
16 Problème 2 : Séquences proches, même repliement, activité différentielle Lectine de cacahuète Arceline (haricot) 2PEL 1IOA 2PELAx0 1IOAAx1 RMSD sur positions conservées = 4,1 Å AETVSFNFNSFSEGNPAINFQGDVTVLSNGNIQLTNLN-----KVNSVGRVLYAMPVRIWSSATGNVASFLTSFSFEMKDIKDYDPADGIIFFIAPEDTQIPAGSIGGGTLGVSDTKGAGHFVGV ATETSFNFPNFHTDDKLI-LQGNATISSKGQLQLTGVGSNELPRVDSLGRAFYSDPIQIKD--SNNVASFNTNFTFIIRAKNQSISAYGLAFALVPVNSPPQKKQEFLGIFNTNNPEPNARTVAV *.****.*.: * :**:.*: *:*::***.:. :*:*:**.:*: *::*. :.***** *.*:* :: ::.* *: * :.* ::. * :...:.:.: *.* Prim.cons. A222SFNF22F222222IN2QG22T22S2G22QLT222SNELP2V2S2GR22Y22P22I22SA22NVASF2T2F2F A2G22F222P G V PELAx0 EFDTYSNSEYNDPPTDHVGIDVNSVDSVKTVPWNSVSGAVVKVTVIYDSSTKTLSVAVTNDNGDITT-IAQVVDLKAKLPERVKFGFSASG--SLGGRQIHLIRSWSFTSTLITTTRRS IOAAx1 VFNTFKNR--IDFDKNFIKPYVN-----ENCDFHKYNGEKTDVQITYDSSNNDLRVFLHFTVSQVKCSVSATVHLEKEVDEWVSVGFSPTSGLTEDTTETHDVLSWSFSSKFRNKLSNILLNNIL *:*:.* *.:.: ** :. ::..*..* : ****.: * * :.::. ::.*.*: :: * *..***.:. :. : * : ****:*.:... Prim.cons. 2F2T22N2EY2D VNSVDSV G2222V222YDSS222L2V S2222V2L22222E2V22GFS222GL H222SWSF2S LLNNIL 26 % d identité entre les séquences
17 Méthodologie SuMo (PHD M. Jambon) Etape 1 : découpage en groupements chimiques aromatic Cα Phényle Hydroxyle hydroxyl Exemple : tyrosine = aromatic, hydroxyl (Jambon et al., Proteins, 2003, Brevet international)
18 Etape 2 : génération d un graphe de triplets de groupements chimiques Sélection des groupements accessibles Connexion des groupements voisins (< 6 Å, par exemple) Définition des triplets de groupements chimiques P3 P P2 P1 : groupement chimique 1 C3 C2 de la protéine C C1 : centre de densité 1 CP: orientation vers l'extérieur
19 Représentation finale des molécules et stockage Graphe des groupements chimiques T4 T2 T1 T4 Graphe des triplets T2 T1 Base de données T3 T3 Utilisation (comparaisons)
20 Principe de la comparaison entre 2 molécules T4 Identification de toutes les paires de triplets similaires Connexion des paires T2 T1 T1/T1' T3 T2/T2' T4/T4' T1' T2' T3' T5' T4' T3/T3' 2 zones de similitude (à affiner)
21 Exemple 1 : protéases à sérine Asp32 His64 Subtilisine Chymotrypsine His57 Asp32 Comparaison subtilisine 1SBC / chymotrypsine 1AFQ Résultat : [Comparison result of (1AFQ,1SBC): Patch number Number of groups: 4 Score = [RMSD = 0.708] [penalty = 0.408] Ser195 Selected pairs of groups: ammonium B HIS ammonium HIS Ser221 aromatic B HIS aromatic HIS Données biochimiques acyl B ASP acyl ASP hydroxyl C SER hydroxyl SER ]
22 Vue de surface des 2 sites catalytiques de protéases Subtilisine (1SBC) Chymotrypsine (1AFQ) His 64 His 57 Asp 32 Ser 221 Asp 102 His195
23 Exemple 2 : lectines de légumineuses Collaboration avec Anne Imberty, CERMAV (Grenoble) Travail sur la famille des lectines de légumineuses Séquences voisines Même repliement
24 Recherche de site à sucre dans la famille des lectines de légumineuses 100% 4 80% 60% 40% % 0% lectines fonctionnelles (avec ou sans ligand) lectines démétallisées arceline non détectés détectés inhibiteurs d'alphaamylase Site recherché : lectine de cacahuète (2PEL) Nombre de protéines analysées = 107 Pourcentage de réussite global = 96 %
25 Illustration : 1DQ1 et 1DQ2 : Concanavaline A Orange : concanavaline A fonctionnelle Bleu : concanavaline A non fonctionnelle (démétallisée) RMSD = 0,9 Å séquences très proches
26 Vue des sites 1DQ1 et 1DQ2 : concanavaline A native et démétallisée Site de fixation du sucre (mannose) Asn 14 Tyr 12 Asp 208 Superposition : Site fonctionnel et site détruit en absence de calcium et de zinc
27 Annotation structurale
28 Etude structure fonction des protéines. Génomique structurale Séquences de génome Détection de phases ouvertes de lecture Analyse comparative Séquences Protéiques Thérapie génique Thérapie protéique Médicaments Agroalimentaire Classification Prédictions Biocristallographie Résonance Magnétique Nucléaire Modélisation Moléculaire Organisation en familles Sélection d un membre représentatif de la famille Détermination de la structure 3D Construction de modèles 3D par homologie Criblage virtuel
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST.
 La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
Big data et sciences du Vivant L'exemple du séquençage haut débit
 Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard [email protected] INRA - MIAT - Plate-forme
Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard [email protected] INRA - MIAT - Plate-forme
Semestre 2 Spécialité «Analyse in silico des complexes macromolécules biologiques-médicaments»
 Master In silico Drug Design Semestre 2 Spécialité «Analyse in silico des complexes macromolécules biologiques-médicaments» 30NU01IS INITIATION A LA PROGRAMMATION (6 ECTS) Responsables : D. MESTIVIER,
Master In silico Drug Design Semestre 2 Spécialité «Analyse in silico des complexes macromolécules biologiques-médicaments» 30NU01IS INITIATION A LA PROGRAMMATION (6 ECTS) Responsables : D. MESTIVIER,
Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant
 Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant Parcours: Master 1 : Bioinformatique et biologie des Systèmes dans le Master
Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant Parcours: Master 1 : Bioinformatique et biologie des Systèmes dans le Master
MASTER (LMD) MANAGEMENT DE PROJET ET INNOVATION EN BIOTECHNOLOGIE
 MASTER (LMD) MANAGEMENT DE PROJET ET INNOVATION EN BIOTECHNOLOGIE RÉSUMÉ DE LA FORMATION Type de diplôme : Master (LMD) Domaine ministériel : Sciences, Technologies, Santé Mention : BIOLOGIE SANTE Spécialité
MASTER (LMD) MANAGEMENT DE PROJET ET INNOVATION EN BIOTECHNOLOGIE RÉSUMÉ DE LA FORMATION Type de diplôme : Master (LMD) Domaine ministériel : Sciences, Technologies, Santé Mention : BIOLOGIE SANTE Spécialité
MABioVis. Bio-informatique et la
 MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
Introduction aux bases de données: application en biologie
 Introduction aux bases de données: application en biologie D. Puthier 1 1 ERM206/Technologies Avancées pour le Génome et la Clinique, http://tagc.univ-mrs.fr/staff/puthier, [email protected] ESIL,
Introduction aux bases de données: application en biologie D. Puthier 1 1 ERM206/Technologies Avancées pour le Génome et la Clinique, http://tagc.univ-mrs.fr/staff/puthier, [email protected] ESIL,
Calcul intensif pour la biologie
 Calcul intensif pour la biologie PPF Bio-informatique et PPF Calcul intensif 14 juin 2011 Calcul intensif... Cluster : ensemble de machines homogènes et localisées, organisées en grappe Grille : infrastructure
Calcul intensif pour la biologie PPF Bio-informatique et PPF Calcul intensif 14 juin 2011 Calcul intensif... Cluster : ensemble de machines homogènes et localisées, organisées en grappe Grille : infrastructure
SysFera. Benjamin Depardon
 SysFera Passage d applications en SaaS Benjamin Depardon CTO@SysFera SysFera Technologie 2001 Création 2010 Spin Off INRIA Direction par un consortium d investisseurs 12 personnes 75% en R&D Implantation
SysFera Passage d applications en SaaS Benjamin Depardon CTO@SysFera SysFera Technologie 2001 Création 2010 Spin Off INRIA Direction par un consortium d investisseurs 12 personnes 75% en R&D Implantation
e-biogenouest CNRS UMR 6074 IRISA-INRIA / Plateforme de Bioinformatique GenOuest [email protected] Programme fédérateur Biogenouest co-financé
 e-biogenouest Coordinateur : Olivier Collin Animateur : Yvan Le Bras CNRS UMR 6074 IRISA-INRIA / Plateforme de Bioinformatique GenOuest [email protected] Programme fédérateur Biogenouest co-financé
e-biogenouest Coordinateur : Olivier Collin Animateur : Yvan Le Bras CNRS UMR 6074 IRISA-INRIA / Plateforme de Bioinformatique GenOuest [email protected] Programme fédérateur Biogenouest co-financé
UE6 - Cycle de vie du médicament : Conception rationnelle
 UE6 - Cycle de vie du médicament : Conception rationnelle Dr. Raphaël Terreux Faculté de Pharmacie (ISPB) Département pédagogique des Sciences Physico-Chimiques et Pharmacie Galénique 8 avenue Rockefeller,
UE6 - Cycle de vie du médicament : Conception rationnelle Dr. Raphaël Terreux Faculté de Pharmacie (ISPB) Département pédagogique des Sciences Physico-Chimiques et Pharmacie Galénique 8 avenue Rockefeller,
Eco-système calcul et données
 Eco-système calcul et données M. Daydé Dr du Comité d'orientation pour le Calcul Intensif (COCIN) Délégué Scientifique INS2I en charge HPC / Grille / Cloud Calcul / données : un enjeu stratégique Calcul
Eco-système calcul et données M. Daydé Dr du Comité d'orientation pour le Calcul Intensif (COCIN) Délégué Scientifique INS2I en charge HPC / Grille / Cloud Calcul / données : un enjeu stratégique Calcul
UTILISATION DE LA PLATEFORME WEB D ANALYSE DE DONNÉES GALAXY
 UTILISATION DE LA PLATEFORME WEB D ANALYSE DE DONNÉES GALAXY Yvan Le Bras [email protected] Cyril Monjeaud, Mathieu Bahin, Claudia Hériveau, Olivier Quenez, Olivier Sallou, Aurélien Roult, Olivier
UTILISATION DE LA PLATEFORME WEB D ANALYSE DE DONNÉES GALAXY Yvan Le Bras [email protected] Cyril Monjeaud, Mathieu Bahin, Claudia Hériveau, Olivier Quenez, Olivier Sallou, Aurélien Roult, Olivier
Formation L.M.D. en instrumentation biomédicale. Mise en œuvre dans une université scientifique et médicale : Claude Bernard Lyon I
 J3eA, Journal sur l enseignement des sciences et technologies de l information et des systèmes, Volume 3, Hors-Série 1, 11 (2004) DOI : http://dx.doi.org/10.1051/bib-j3ea:2004611 EDP Sciences, 2004 Formation
J3eA, Journal sur l enseignement des sciences et technologies de l information et des systèmes, Volume 3, Hors-Série 1, 11 (2004) DOI : http://dx.doi.org/10.1051/bib-j3ea:2004611 EDP Sciences, 2004 Formation
MASTER 2 SCIENCES DU MEDICAMENT
 MASTER 2 SCIENCES DU MEDICAMENT RÉSUMÉ DE LA FORMATION Type de diplôme : Master (LMD) Domaine ministériel : Sciences, Technologies, Santé Présentation La vocation de ce diplôme est d'apporter une formation
MASTER 2 SCIENCES DU MEDICAMENT RÉSUMÉ DE LA FORMATION Type de diplôme : Master (LMD) Domaine ministériel : Sciences, Technologies, Santé Présentation La vocation de ce diplôme est d'apporter une formation
Dans la fonction de maître de formation pratique. Dans la fonction de maître assistant
 LA HAUTE ECOLE FRANCISCO FERRER porte à votre connaissance qu'une réserve de recrutement est constituée pr l'année académique 2015-2016. En cas d'engagement, et pr autant que le budget allé par la Communauté
LA HAUTE ECOLE FRANCISCO FERRER porte à votre connaissance qu'une réserve de recrutement est constituée pr l'année académique 2015-2016. En cas d'engagement, et pr autant que le budget allé par la Communauté
Présentation du Master Ingénierie Informatique et du Master Science Informatique 2007-2008, Année 2 Université Paris-Est Marne-la-Vallée
 Présentation du Master Ingénierie Informatique et du Master Science Informatique 2007-2008, Année 2 Université Paris-Est Marne-la-Vallée Responsable du Master Informatique : Marc Zipstein Responsable de
Présentation du Master Ingénierie Informatique et du Master Science Informatique 2007-2008, Année 2 Université Paris-Est Marne-la-Vallée Responsable du Master Informatique : Marc Zipstein Responsable de
! Séquence et structure des macromolécules. " Séquences protéiques (UniProt) " Séquences nucléotidiques (EMBL / ENA, Genbank, DDBJ)
 Introduction à la Bioinformatique Introduction! Les bases de données jouent un rôle crucial dans l organisation des connaissances biologiques.! Nous proposons ici un tour rapide des principales bases de
Introduction à la Bioinformatique Introduction! Les bases de données jouent un rôle crucial dans l organisation des connaissances biologiques.! Nous proposons ici un tour rapide des principales bases de
Calcul Haute Performance & Données
 Calcul Haute Performance & Données Plan de l exposé CALMIP : news Evolution Matérielle Evolution des systèmes de calcul (CALMIP) Evolution Applicative Modélisation et Calcul haute performance Calcul haute
Calcul Haute Performance & Données Plan de l exposé CALMIP : news Evolution Matérielle Evolution des systèmes de calcul (CALMIP) Evolution Applicative Modélisation et Calcul haute performance Calcul haute
Budget Constrained Resource Allocation for Non-Deterministic Workflows on a IaaS Cloud
 Budget Constrained Resource Allocation for Non-Deterministic Workflows on a IaaS Cloud Eddy Caron, Frédéric Desprez, Adrian Muresan, Frédéric Suter To cite this version: Eddy Caron, Frédéric Desprez, Adrian
Budget Constrained Resource Allocation for Non-Deterministic Workflows on a IaaS Cloud Eddy Caron, Frédéric Desprez, Adrian Muresan, Frédéric Suter To cite this version: Eddy Caron, Frédéric Desprez, Adrian
Prédiction de la structure d une
 Prédiction de la structure d une protéine Soluscience Guillaume Chakroun guillaume [email protected] Copyright c 2004 Guillaume Chakroun TABLE DES MATIÈRES Table des matières 1 Les structures protéiques
Prédiction de la structure d une protéine Soluscience Guillaume Chakroun guillaume [email protected] Copyright c 2004 Guillaume Chakroun TABLE DES MATIÈRES Table des matières 1 Les structures protéiques
Partie Observer : Ondes et matière CHAP 04-ACT/DOC Analyse spectrale : Spectroscopies IR et RMN
 Partie Observer : Ondes et matière CHAP 04-ACT/DOC Analyse spectrale : Spectroscopies IR et RMN Objectifs : Exploiter un spectre infrarouge pour déterminer des groupes caractéristiques Relier un spectre
Partie Observer : Ondes et matière CHAP 04-ACT/DOC Analyse spectrale : Spectroscopies IR et RMN Objectifs : Exploiter un spectre infrarouge pour déterminer des groupes caractéristiques Relier un spectre
Physiopathologie : de la Molécule à l'homme
 Mention Sciences du Vivant Spécialité de Master : Physiopathologie : de la Molécule à l'homme Pourquoi, comment, combien, contourner, greffer, restaurer, inhiber, suppléer Responsables : Dr Gilles Prévost
Mention Sciences du Vivant Spécialité de Master : Physiopathologie : de la Molécule à l'homme Pourquoi, comment, combien, contourner, greffer, restaurer, inhiber, suppléer Responsables : Dr Gilles Prévost
Conception de Médicament
 Conception de Médicament Approche classique HTS Chimie combinatoire Rational Drug Design Ligand based (QSAR) Structure based (ligand et ou macromolec.) 3DQSAR Docking Virtual screening Needle in a Haystack
Conception de Médicament Approche classique HTS Chimie combinatoire Rational Drug Design Ligand based (QSAR) Structure based (ligand et ou macromolec.) 3DQSAR Docking Virtual screening Needle in a Haystack
Les mésocentres HPC àportée de clic des utilisateurs industriels
 Les mésocentres HPC àportée de clic des utilisateurs industriels Université de Reims Champagne-Ardenne (URCA) Centre de Calcul ROMEO Multidisciplinary university more than 22 000 students a wide initial
Les mésocentres HPC àportée de clic des utilisateurs industriels Université de Reims Champagne-Ardenne (URCA) Centre de Calcul ROMEO Multidisciplinary university more than 22 000 students a wide initial
Thierry DELZESCAUX. «biopicsel» group, URA CNRS-CEA 2210 Service MIRCen, I²BM, CEA Fontenay-aux-Roses, France. Thierry.Delzescaux@cea.
 Thierry DELZESCAUX «biopicsel» group, URA CNRS-CEA 2210 Service MIRCen, I²BM, CEA Fontenay-aux-Roses, France [email protected] Thierry DELZESCAUX Ingénieur chercheur CEA Experience Since 2008:
Thierry DELZESCAUX «biopicsel» group, URA CNRS-CEA 2210 Service MIRCen, I²BM, CEA Fontenay-aux-Roses, France [email protected] Thierry DELZESCAUX Ingénieur chercheur CEA Experience Since 2008:
Comment reproduire les résultats de l article : POP-Java : Parallélisme et distribution orienté objet
 Comment reproduire les résultats de l article : POP-Java : Parallélisme et distribution orienté objet Beat Wolf 1, Pierre Kuonen 1, Thomas Dandekar 2 1 icosys, Haute École Spécialisée de Suisse occidentale,
Comment reproduire les résultats de l article : POP-Java : Parallélisme et distribution orienté objet Beat Wolf 1, Pierre Kuonen 1, Thomas Dandekar 2 1 icosys, Haute École Spécialisée de Suisse occidentale,
Identification de nouveaux membres dans des familles d'interleukines
 Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
Laboratoire de Photophysique et de Photochimie Supra- et Macromoléculaires (UMR 8531)
 Unité Mixte du CNRS (UMR8531) Institut de Chimie Directeur : Keitaro NAKATANI (PU ENS Cachan) Courrier électronique : [email protected] http://www.ppsm.ens-cachan.fr Problématique générale :
Unité Mixte du CNRS (UMR8531) Institut de Chimie Directeur : Keitaro NAKATANI (PU ENS Cachan) Courrier électronique : [email protected] http://www.ppsm.ens-cachan.fr Problématique générale :
Tutoriel Cloud IFB - Initiation -
 Tutoriel Cloud IFB - Initiation - Christophe BLANCHET Institut Français de Bioinformatique - IFB French Institute of Bioinformatics - ELIXIR-FR CNRS UMS3601 - Gif-sur-Yvette - FRANCE Ecole Cumulo NumBio
Tutoriel Cloud IFB - Initiation - Christophe BLANCHET Institut Français de Bioinformatique - IFB French Institute of Bioinformatics - ELIXIR-FR CNRS UMS3601 - Gif-sur-Yvette - FRANCE Ecole Cumulo NumBio
Formavie 2010. 2 Différentes versions du format PDB...3. 3 Les champs dans les fichiers PDB...4. 4 Le champ «ATOM»...5. 6 Limites du format PDB...
 Formavie 2010 Les fichiers PDB Les fichiers PDB contiennent les informations qui vont permettre à des logiciels de visualisation moléculaire (ex : RasTop ou Jmol) d afficher les molécules. Un fichier au
Formavie 2010 Les fichiers PDB Les fichiers PDB contiennent les informations qui vont permettre à des logiciels de visualisation moléculaire (ex : RasTop ou Jmol) d afficher les molécules. Un fichier au
CHAPITRE 3 LA SYNTHESE DES PROTEINES
 CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010
 GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010 Analyse de la diversité moléculaire des régions génomiques de 30 gènes du développement méristématique dans une core collection
GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010 Analyse de la diversité moléculaire des régions génomiques de 30 gènes du développement méristématique dans une core collection
Mise en œuvre de la virtualisation à l IGBMC. Guillaume Seith Remy Fritz
 Mise en œuvre de la virtualisation à l IGBMC. Guillaume Seith Remy Fritz Introduction Contexte Objectifs Mise en œuvre Présentation Exploitation Conclusion IGBMC: Institut de génétique et de Biologie Moléculaire
Mise en œuvre de la virtualisation à l IGBMC. Guillaume Seith Remy Fritz Introduction Contexte Objectifs Mise en œuvre Présentation Exploitation Conclusion IGBMC: Institut de génétique et de Biologie Moléculaire
Les Parcours Scientifiques et les Ecoles Doctorales
 Aix 11 Septembre 2006 Les Parcours Scientifiques et les Ecoles Doctorales «Sciences de la Vie» Jean-François DHAINAUT Président du Pôle de Recherche et Enseignement Supérieur «Université Paris-Centre»
Aix 11 Septembre 2006 Les Parcours Scientifiques et les Ecoles Doctorales «Sciences de la Vie» Jean-François DHAINAUT Président du Pôle de Recherche et Enseignement Supérieur «Université Paris-Centre»
La reconnaissance moléculaire: la base du design rationnel Modélisation moléculaire: Introduction Hiver 2006
 La reconnaissance moléculaire: la base du design rationnel En 1890 Emil Fisher a proposé le modèle "serrure et clé" pour expliquer la façon de fonctionner des systèmes biologiques. Un substrat rentre et
La reconnaissance moléculaire: la base du design rationnel En 1890 Emil Fisher a proposé le modèle "serrure et clé" pour expliquer la façon de fonctionner des systèmes biologiques. Un substrat rentre et
Programme consultant expert (PCE)
 Programme consultant expert (PCE) Bénéficiez par le biais du PCE de l expertise d un consultant sénior et/ou d un jeune chercheur expérimenté Ce programme s adresse aux entreprises de la filière chimie
Programme consultant expert (PCE) Bénéficiez par le biais du PCE de l expertise d un consultant sénior et/ou d un jeune chercheur expérimenté Ce programme s adresse aux entreprises de la filière chimie
E-Biothon : Une plate-forme pour accélérer les recherches en biologie, santé et environnement.
 E-Biothon : Une plate-forme pour accélérer les recherches en biologie, santé et environnement. N.Bard, S.Boin, F.Bothorel, P.Collinet, M.Daydé, B. Depardon, F. Desprez, M.Flé, A.Franc, J.-F. Gibrat, D.
E-Biothon : Une plate-forme pour accélérer les recherches en biologie, santé et environnement. N.Bard, S.Boin, F.Bothorel, P.Collinet, M.Daydé, B. Depardon, F. Desprez, M.Flé, A.Franc, J.-F. Gibrat, D.
Panorama des formations en biotechnologie
 Panorama des formations en biotechnologie 5) Les formations en licence, master et doctorat 5-1) Les licences professionnelles Université spécialité(s) AIX-MARSEILLE 1 (en cohabilitation avec Aix Marseille
Panorama des formations en biotechnologie 5) Les formations en licence, master et doctorat 5-1) Les licences professionnelles Université spécialité(s) AIX-MARSEILLE 1 (en cohabilitation avec Aix Marseille
Base de données bibliographiques Pubmed-Medline
 Chapitre 1 ; Domaine 1 ; Documentation ; Champs référentiels 1.1.1, 1.1.2 et 1.1.3 Base de données bibliographiques Pubmed-Medline D r Patrick Deschamps,, 30 mai 2007 PLAN C2i métiers de la santé Introduction
Chapitre 1 ; Domaine 1 ; Documentation ; Champs référentiels 1.1.1, 1.1.2 et 1.1.3 Base de données bibliographiques Pubmed-Medline D r Patrick Deschamps,, 30 mai 2007 PLAN C2i métiers de la santé Introduction
Objectifs. Clustering. Principe. Applications. Applications. Cartes de crédits. Remarques. Biologie, Génomique
 Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
Laboratoire d informatique Gaspard-Monge UMR 8049. Journée Labex Bézout- ANSES
 Laboratoire d informatique Gaspard-Monge UMR 8049 Journée Labex Bézout- ANSES Présentation du laboratoire 150 membres, 71 chercheurs et enseignants-chercheurs, 60 doctorants 4 tutelles : CNRS, École des
Laboratoire d informatique Gaspard-Monge UMR 8049 Journée Labex Bézout- ANSES Présentation du laboratoire 150 membres, 71 chercheurs et enseignants-chercheurs, 60 doctorants 4 tutelles : CNRS, École des
Informatique. epims : un LIMS pour la gestion des données de spectrométrie de masse TECHNOLOGIE APPLIQUÉE
 Véronique DUPIERRIS 1, Damien BARTHE 2, Christophe BRULEY 2 epims : un LIMS pour la gestion des données de spectrométrie de masse Informatique RÉSUMÉ La protéomique constitue aujourd hui un outil de choix
Véronique DUPIERRIS 1, Damien BARTHE 2, Christophe BRULEY 2 epims : un LIMS pour la gestion des données de spectrométrie de masse Informatique RÉSUMÉ La protéomique constitue aujourd hui un outil de choix
Ingénieur R&D en bio-informatique
 Ingénieur R&D en bio-informatique Spécialisé Bases De Données 33 ans, Célibataire. Biologie & Informatique gabriel.chandesris[at]laposte.net {06 56 41 97 37} Use the bipper! http://gabriel.chandesris.free.fr/
Ingénieur R&D en bio-informatique Spécialisé Bases De Données 33 ans, Célibataire. Biologie & Informatique gabriel.chandesris[at]laposte.net {06 56 41 97 37} Use the bipper! http://gabriel.chandesris.free.fr/
Charger une structure Comment ouvrir un fichier avec VMD? Comment charger un fichier PDB directement depuis la Protein Data Bank? Comment charger un
 Charger une structure Comment ouvrir un fichier avec VMD? Comment charger un fichier PDB directement depuis la Protein Data Bank? Comment charger un fichier restrt de AMBER? Comment charger une trajectoire
Charger une structure Comment ouvrir un fichier avec VMD? Comment charger un fichier PDB directement depuis la Protein Data Bank? Comment charger un fichier restrt de AMBER? Comment charger une trajectoire
IN2P3 et PLUME Valorisation de la production de logiciels
 IN2P3 et PLUME Valorisation de la production de logiciels Teresa Gomez-Diaz Laboratoire d informatique Gaspard-Monge PLUME e-séminaire Réseau des informaticiens de l IN2P3 et de l IRFU (RI3) LAL, Orsay,
IN2P3 et PLUME Valorisation de la production de logiciels Teresa Gomez-Diaz Laboratoire d informatique Gaspard-Monge PLUME e-séminaire Réseau des informaticiens de l IN2P3 et de l IRFU (RI3) LAL, Orsay,
Ecole Mohammadia d Ingénieurs Systèmes Répartis Pr. Slimane Bah, ing. PhD G. Informatique Semaine 24
 Ecole Mohammadia d Ingénieurs Systèmes Répartis Pr. Slimane Bah, ing. PhD G. Informatique Semaine 24 1 Semestre 4 : Fev. 2015 Cluster Caractéristiques : Centralisé Fortement couplé Même domaine administratif
Ecole Mohammadia d Ingénieurs Systèmes Répartis Pr. Slimane Bah, ing. PhD G. Informatique Semaine 24 1 Semestre 4 : Fev. 2015 Cluster Caractéristiques : Centralisé Fortement couplé Même domaine administratif
Application à l astrophysique ACTIVITE
 Application à l astrophysique Seconde ACTIVITE I ) But : Le but de l activité est de donner quelques exemples d'utilisations pratiques de l analyse spectrale permettant de connaître un peu mieux les étoiles.
Application à l astrophysique Seconde ACTIVITE I ) But : Le but de l activité est de donner quelques exemples d'utilisations pratiques de l analyse spectrale permettant de connaître un peu mieux les étoiles.
Analyse empirique et modélisation de la dynamique de la topologie de l Internet
 Analyse empirique et modélisation de la dynamique de la topologie de l Internet Sergey Kirgizov Directrice de thèse: Clémence Magnien Complex Networks, LIP6, (UPMC, CNRS) Paris, 12 décembre 2014 Plan 1
Analyse empirique et modélisation de la dynamique de la topologie de l Internet Sergey Kirgizov Directrice de thèse: Clémence Magnien Complex Networks, LIP6, (UPMC, CNRS) Paris, 12 décembre 2014 Plan 1
MAB Solut. vos projets. MABLife Génopole Campus 1 5 rue Henri Desbruères 91030 Evry Cedex. www.mabsolut.com. intervient à chaque étape de
 Mabsolut-DEF-HI:Mise en page 1 17/11/11 17:45 Page1 le département prestataire de services de MABLife de la conception à la validation MAB Solut intervient à chaque étape de vos projets Création d anticorps
Mabsolut-DEF-HI:Mise en page 1 17/11/11 17:45 Page1 le département prestataire de services de MABLife de la conception à la validation MAB Solut intervient à chaque étape de vos projets Création d anticorps
Possibilités offertes après la L2?
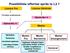 Possibilités offertes après la L2? Licence Pro Licence Générale Formation professionnel Diplôme Bac +3 Diplôme Bac +3 Master Vie Active : Technicien Assistant Ingénieur Laboratoire Public et Privé Master
Possibilités offertes après la L2? Licence Pro Licence Générale Formation professionnel Diplôme Bac +3 Diplôme Bac +3 Master Vie Active : Technicien Assistant Ingénieur Laboratoire Public et Privé Master
Notre métier : Vous accompagner dans votre Projet
 Notre métier : Vous accompagner dans votre Projet Formation initiale en alternance (scolaire ou apprentissage) Formation continue FORMATION INITIALE > Bac Pro Bio Industries de Transformation Accéder x
Notre métier : Vous accompagner dans votre Projet Formation initiale en alternance (scolaire ou apprentissage) Formation continue FORMATION INITIALE > Bac Pro Bio Industries de Transformation Accéder x
IMMUNOLOGIE. La spécificité des immunoglobulines et des récepteurs T. Informations scientifiques
 IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
July 1, 2013. Stéphan Clémençon (Télécom ParisTech) Mastère Big Data July 1, 2013 1 / 15
 Mastère Spécialisé Big Data Stéphan Clémençon Télécom ParisTech July 1, 2013 Stéphan Clémençon (Télécom ParisTech) Mastère Big Data July 1, 2013 1 / 15 Agenda Contexte et Opportunité Les grandes lignes
Mastère Spécialisé Big Data Stéphan Clémençon Télécom ParisTech July 1, 2013 Stéphan Clémençon (Télécom ParisTech) Mastère Big Data July 1, 2013 1 / 15 Agenda Contexte et Opportunité Les grandes lignes
MaiMoSiNE Maison de la Modélisation et de la Simulation : Nano Sciences et Environnement
 Mercredi 9 juillet 24 MaiMoSiNE Maison de la Modélisation et de la Simulation : Nano Sciences et Environnement Université de Grenoble et CNRS Présentation à la Journée de l ARC6 L organisation Institut
Mercredi 9 juillet 24 MaiMoSiNE Maison de la Modélisation et de la Simulation : Nano Sciences et Environnement Université de Grenoble et CNRS Présentation à la Journée de l ARC6 L organisation Institut
Master Développement et Immunologie
 Master Développement et Immunologie MASTER 2 PROFESSIONNALISANT EN IMMUNOLOGIE Le Master 2 Professionnalisant en Immunologie propose aux étudiants des filières scientifiques et médicales une formation
Master Développement et Immunologie MASTER 2 PROFESSIONNALISANT EN IMMUNOLOGIE Le Master 2 Professionnalisant en Immunologie propose aux étudiants des filières scientifiques et médicales une formation
ANALYSE SPECTRALE. monochromateur
 ht ANALYSE SPECTRALE Une espèce chimique est susceptible d interagir avec un rayonnement électromagnétique. L étude de l intensité du rayonnement (absorbé ou réémis) en fonction des longueurs d ode s appelle
ht ANALYSE SPECTRALE Une espèce chimique est susceptible d interagir avec un rayonnement électromagnétique. L étude de l intensité du rayonnement (absorbé ou réémis) en fonction des longueurs d ode s appelle
Les lières. MSc in Electronics and Information Technology Engineering. Ingénieur civil. en informatique. MSc in Architectural Engineering
 Ingénieur civil Ingénieur civil Les lières MSc in Electronics and Information Technology Engineering MSc in Architectural Engineering MSc in Civil Engineering MSc in Electromechanical Engineering MSc
Ingénieur civil Ingénieur civil Les lières MSc in Electronics and Information Technology Engineering MSc in Architectural Engineering MSc in Civil Engineering MSc in Electromechanical Engineering MSc
Recherche d'images par le contenu Application au monitoring Télévisuel à l'institut national de l'audiovisuel
 Recherche d'images par le contenu Application au monitoring Télévisuel à l'institut national de l'audiovisuel Alexis Joly [email protected] INRIA - IMEDIA Alexis Joly cours monitoring p. 1 Plan de l'exposé
Recherche d'images par le contenu Application au monitoring Télévisuel à l'institut national de l'audiovisuel Alexis Joly [email protected] INRIA - IMEDIA Alexis Joly cours monitoring p. 1 Plan de l'exposé
3A-IIC - Parallélisme & Grid GRID : Définitions. GRID : Définitions. Stéphane Vialle. [email protected] http://www.metz.supelec.
 3A-IIC - Parallélisme & Grid Stéphane Vialle [email protected] http://www.metz.supelec.fr/~vialle Principes et Objectifs Evolution Leçons du passé Composition d une Grille Exemple d utilisation
3A-IIC - Parallélisme & Grid Stéphane Vialle [email protected] http://www.metz.supelec.fr/~vialle Principes et Objectifs Evolution Leçons du passé Composition d une Grille Exemple d utilisation
Marie Curie Actions Marie Curie Career Integration Grant (CIG) Call: FP7-People-2012-CIG
 Marie Curie Actions Marie Curie Career Integration Grant (CIG) Call: FP7-People-2012-CIG Genomerge : role of genome merger and redundancy in plant evolution and adaptation Mathieu ROUSSEAU- GUEUTIN UMR-
Marie Curie Actions Marie Curie Career Integration Grant (CIG) Call: FP7-People-2012-CIG Genomerge : role of genome merger and redundancy in plant evolution and adaptation Mathieu ROUSSEAU- GUEUTIN UMR-
TRS: Sélection des sous-graphes représentants par l intermédiaire des attributs topologiques et K-medoïdes
 TRS: Sélection des sous-graphes représentants par l intermédiaire des attributs topologiques et K-medoïdes Mohamed Moussaoui,Wajdi Dhifli,Sami Zghal,Engelbert Mephu Nguifo FSJEG, Université de Jendouba,
TRS: Sélection des sous-graphes représentants par l intermédiaire des attributs topologiques et K-medoïdes Mohamed Moussaoui,Wajdi Dhifli,Sami Zghal,Engelbert Mephu Nguifo FSJEG, Université de Jendouba,
Sciences Humaines et Sociales. Informatique et applications. VIGNERON Vincent [email protected] STIC Traitement du signal et des images
 Sujets de thèse Ecole Doctorale "Sciences et Ingénierie" 2012-2013 Sujet de thèse Unité de recherche Nom de l'encadrant Discipline principale Discipline secondaire Navigation topologique basée sur des
Sujets de thèse Ecole Doctorale "Sciences et Ingénierie" 2012-2013 Sujet de thèse Unité de recherche Nom de l'encadrant Discipline principale Discipline secondaire Navigation topologique basée sur des
Modélisation prédictive et incertitudes. P. Pernot. Laboratoire de Chimie Physique, CNRS/U-PSUD, Orsay [email protected]
 Modélisation prédictive et incertitudes P. Pernot Laboratoire de Chimie Physique, CNRS/U-PSUD, Orsay [email protected] Le concept de Mesure Virtuelle mesure virtuelle résultat d un modèle visant
Modélisation prédictive et incertitudes P. Pernot Laboratoire de Chimie Physique, CNRS/U-PSUD, Orsay [email protected] Le concept de Mesure Virtuelle mesure virtuelle résultat d un modèle visant
Big Data et Graphes : Quelques pistes de recherche
 Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci http://liris.cnrs.fr/hamamache.kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de
Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci http://liris.cnrs.fr/hamamache.kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de
Résonance Magnétique Nucléaire : RMN
 21 Résonance Magnétique Nucléaire : RMN Salle de TP de Génie Analytique Ce document résume les principaux aspects de la RMN nécessaires à la réalisation des TP de Génie Analytique de 2ème année d IUT de
21 Résonance Magnétique Nucléaire : RMN Salle de TP de Génie Analytique Ce document résume les principaux aspects de la RMN nécessaires à la réalisation des TP de Génie Analytique de 2ème année d IUT de
FACULTÉ DES SCIENCES FACULTY OF SCIENCES OFFRE DE FORMATION CONDITIONS D ADMISSION COURSE OFFERING ADMISSION REQUIREMENTS
 FACULTÉ DES SCIENCES FACULTY OF SCIENCES OFFRE DE FORMATION CONDITIONS D ADMISSION COURSE OFFERING ADMISSION REQUIREMENTS La Faculté des Sciences de l USEK Présentation Essentiellement orientée vers les
FACULTÉ DES SCIENCES FACULTY OF SCIENCES OFFRE DE FORMATION CONDITIONS D ADMISSION COURSE OFFERING ADMISSION REQUIREMENTS La Faculté des Sciences de l USEK Présentation Essentiellement orientée vers les
Compléments - Chapitre 5 Spectroscopie
 ompléments - hapitre 5 Spectroscopie Spectroscopie par résonance magnétique nucléaire (RMN 13 ) Tandis que la spectroscopie RMN 1 H fournit des données sur la disposition des atomes d'hydrogène dans une
ompléments - hapitre 5 Spectroscopie Spectroscopie par résonance magnétique nucléaire (RMN 13 ) Tandis que la spectroscopie RMN 1 H fournit des données sur la disposition des atomes d'hydrogène dans une
Dr Pascale Vergne-Salle Service de Rhumatologie, CHU de Limoges. Membre enseignant chercheur EA 4021
 Dr Pascale Vergne-Salle Service de Rhumatologie, CHU de Limoges Membre enseignant chercheur EA 4021 Rhumatisme inflammatoire chronique Prévalence des SA ies en Europe = 1 à 2% Age moy : 26 ans, ratio homme/femme:
Dr Pascale Vergne-Salle Service de Rhumatologie, CHU de Limoges Membre enseignant chercheur EA 4021 Rhumatisme inflammatoire chronique Prévalence des SA ies en Europe = 1 à 2% Age moy : 26 ans, ratio homme/femme:
Mise en place d une plateforme de gestion de matériels biologiques : quels avantages pour les chercheurs?
 Mise en place d une plateforme de gestion de matériels biologiques : quels avantages pour les chercheurs? Dr Xavier Manival, Laboratoire IMoPA, CR, CNRS Françoise Tisserand-Bedri, Documentaliste, Inist-CNRS
Mise en place d une plateforme de gestion de matériels biologiques : quels avantages pour les chercheurs? Dr Xavier Manival, Laboratoire IMoPA, CR, CNRS Françoise Tisserand-Bedri, Documentaliste, Inist-CNRS
TerrOïko : JEU en collaboration avec la SEEM
 TerrOïko : JEU en collaboration avec la SEEM Journée EcoInnovation et Biodiversité 21 mai 2014 - Contact: Catherine de Roincé, [email protected] Historique d une start-up issue de la recherche
TerrOïko : JEU en collaboration avec la SEEM Journée EcoInnovation et Biodiversité 21 mai 2014 - Contact: Catherine de Roincé, [email protected] Historique d une start-up issue de la recherche
RESIF Une infrastructure de recherche pour l'observation des déformations de la terre site web : http://www.resif.fr
 RESIF Une infrastructure de recherche pour l'observation des déformations de la terre site web : http://www.resif.fr Catherine Péquegnat Univ. Grenoble Alpes, ISTerre, F-38041 Grenoble, France CNRS, ISTerre,
RESIF Une infrastructure de recherche pour l'observation des déformations de la terre site web : http://www.resif.fr Catherine Péquegnat Univ. Grenoble Alpes, ISTerre, F-38041 Grenoble, France CNRS, ISTerre,
Ordonnancement sous contraintes de Qualité de Service dans les Clouds
 Ordonnancement sous contraintes de Qualité de Service dans les Clouds GUÉROUT Tom DA COSTA Georges (SEPIA) MONTEIL Thierry (SARA) 05/12/2014 1 Contexte CLOUD COMPUTING Contexte : Environnement de Cloud
Ordonnancement sous contraintes de Qualité de Service dans les Clouds GUÉROUT Tom DA COSTA Georges (SEPIA) MONTEIL Thierry (SARA) 05/12/2014 1 Contexte CLOUD COMPUTING Contexte : Environnement de Cloud
23. Interprétation clinique des mesures de l effet traitement
 23. Interprétation clinique des mesures de l effet traitement 23.1. Critères de jugement binaires Plusieurs mesures (indices) sont utilisables pour quantifier l effet traitement lors de l utilisation d
23. Interprétation clinique des mesures de l effet traitement 23.1. Critères de jugement binaires Plusieurs mesures (indices) sont utilisables pour quantifier l effet traitement lors de l utilisation d
Perl Orienté Objet BioPerl There is more than one way to do it
 Perl Orienté Objet BioPerl There is more than one way to do it Bérénice Batut, [email protected] DUT Génie Biologique Option Bioinformatique Année 2014-2015 Perl Orienté Objet - BioPerl Rappels
Perl Orienté Objet BioPerl There is more than one way to do it Bérénice Batut, [email protected] DUT Génie Biologique Option Bioinformatique Année 2014-2015 Perl Orienté Objet - BioPerl Rappels
A GRASPxELS approach for the Job Shop with generic time-lags and new statistical determination of the parameters
 A GRASPxELS approach for the Job Shop with generic time-lags and new statistical determination of the parameters Présenté par : Equipe de travail : Laboratoire : Maxime CHASSAING Philippe LACOMME, Nikolay
A GRASPxELS approach for the Job Shop with generic time-lags and new statistical determination of the parameters Présenté par : Equipe de travail : Laboratoire : Maxime CHASSAING Philippe LACOMME, Nikolay
Spécialisation 3A AgroSup Dijon IAA Microbiologie Industrielle et Biotechnologie (MIB)
 Spécialisation 3A AgroSup Dijon IAA Microbiologie Industrielle et Biotechnologie (MIB) Responsable : Jean-François Cavin (Pr. Microbiologie Biotechnologie) Tel 03 80 77 40 72, Fax 03 80 77 23 84 [email protected]
Spécialisation 3A AgroSup Dijon IAA Microbiologie Industrielle et Biotechnologie (MIB) Responsable : Jean-François Cavin (Pr. Microbiologie Biotechnologie) Tel 03 80 77 40 72, Fax 03 80 77 23 84 [email protected]
Alphonse Carlier, Intelligence Économique et Knowledge Management, AFNOR Éditions, 2012.
 1 Du même auteur chez le même éditeur Alphonse Carlier, Intelligence Économique et Knowledge Management, AFNOR Éditions, 2012. AFNOR 2013 Couverture : création AFNOR Éditions Crédit photo 2011 Fotolia
1 Du même auteur chez le même éditeur Alphonse Carlier, Intelligence Économique et Knowledge Management, AFNOR Éditions, 2012. AFNOR 2013 Couverture : création AFNOR Éditions Crédit photo 2011 Fotolia
EVALUATION DE LA QUALITE DES SONDAGES EN LIGNE : CAS D UN SONDAGE D OPINION AU BURKINA FASO
 EVALUATION DE LA QUALITE DES SONDAGES EN LIGNE : CAS D UN SONDAGE D OPINION AU BURKINA FASO Auteur Baguinébié Bazongo 1 Ingénieur Statisticien Economiste Chef de l Unité de recherche à l Institut national
EVALUATION DE LA QUALITE DES SONDAGES EN LIGNE : CAS D UN SONDAGE D OPINION AU BURKINA FASO Auteur Baguinébié Bazongo 1 Ingénieur Statisticien Economiste Chef de l Unité de recherche à l Institut national
présentée DEVANT L UNIVERSITÉ DE RENNES 1 pour obtenir le grade de : DOCTEUR DE L UNIVERSITÉ DE RENNES 1 PAR Emilie GUÉRIN TITRE DE LA THÈSE :
 N Ordre de la Thèse 3282 THÈSE présentée DEVANT L UNIVERSITÉ DE RENNES 1 pour obtenir le grade de : DOCTEUR DE L UNIVERSITÉ DE RENNES 1 Mention : BIOLOGIE PAR Emilie GUÉRIN Équipe d accueil : École Doctorale
N Ordre de la Thèse 3282 THÈSE présentée DEVANT L UNIVERSITÉ DE RENNES 1 pour obtenir le grade de : DOCTEUR DE L UNIVERSITÉ DE RENNES 1 Mention : BIOLOGIE PAR Emilie GUÉRIN Équipe d accueil : École Doctorale
ARCHEOVISION. Centre de Ressources Numériques 3D. UMR 5607 du CNRS. R. Vergnieux IR-CNRS
 ARCHEOVISION Centre de Ressources Numériques 3D R. Vergnieux IR-CNRS UMR 5607 du CNRS Archéovision CNRS Caroline Delevoie (AI cnrs Coordination - 2D) Bruno Dutailly (IE cnrs Dev. 3D) Robert Vergnieux (IR
ARCHEOVISION Centre de Ressources Numériques 3D R. Vergnieux IR-CNRS UMR 5607 du CNRS Archéovision CNRS Caroline Delevoie (AI cnrs Coordination - 2D) Bruno Dutailly (IE cnrs Dev. 3D) Robert Vergnieux (IR
Vers une approche Adaptative pour la Découverte et la Composition Dynamique des Services
 69 Vers une approche Adaptative pour la Découverte et la Composition Dynamique des Services M. Bakhouya, J. Gaber et A. Koukam Laboratoire Systèmes et Transports SeT Université de Technologie de Belfort-Montbéliard
69 Vers une approche Adaptative pour la Découverte et la Composition Dynamique des Services M. Bakhouya, J. Gaber et A. Koukam Laboratoire Systèmes et Transports SeT Université de Technologie de Belfort-Montbéliard
DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION
 DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION PRINCIPES DE BASE SUR LES DONNEES ET LE CALCUL HAUTE PERFORMANCE Lois de Gray sur l ingénierie des données 1 : Les calculs scientifiques traitent des volumes considérables
DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION PRINCIPES DE BASE SUR LES DONNEES ET LE CALCUL HAUTE PERFORMANCE Lois de Gray sur l ingénierie des données 1 : Les calculs scientifiques traitent des volumes considérables
Evaluation des performances de programmes parallèles haut niveau à base de squelettes
 Evaluation des performances de programmes parallèles haut niveau à base de squelettes Enhancing the Performance Predictability of Grid Applications with Patterns and Process Algebras A. Benoit, M. Cole,
Evaluation des performances de programmes parallèles haut niveau à base de squelettes Enhancing the Performance Predictability of Grid Applications with Patterns and Process Algebras A. Benoit, M. Cole,
à l intelligence économique
 Délégation interministérielle à l intelligence économique Délégation interministérielle à l intelligence économique Avril 2014 Kit de présentation Qu est-ce-que c est? DIESE-Lab est un logiciel évaluant
Délégation interministérielle à l intelligence économique Délégation interministérielle à l intelligence économique Avril 2014 Kit de présentation Qu est-ce-que c est? DIESE-Lab est un logiciel évaluant
ASA-Advanced Solutions Accelerator. Solution pour la gestion des données des laboratoires et des plateformes de service
 ASA-Advanced Solutions Accelerator Partenaire informatique des Laboratoires de Recherche 100lims Solution pour la gestion des données des laboratoires et des plateformes de service Parce que vous cherchez
ASA-Advanced Solutions Accelerator Partenaire informatique des Laboratoires de Recherche 100lims Solution pour la gestion des données des laboratoires et des plateformes de service Parce que vous cherchez
MYRIAD. l ADN isolé n est à présent plus brevetable!
 MYRIAD La Cour Suprême des Etats-Unis revient sur plus de 30 ans de pratique : l ADN isolé n est à présent plus brevetable! Mauvaise passe pour les inventions en biotechnologies sur le territoire américain.
MYRIAD La Cour Suprême des Etats-Unis revient sur plus de 30 ans de pratique : l ADN isolé n est à présent plus brevetable! Mauvaise passe pour les inventions en biotechnologies sur le territoire américain.
E-BIOGENOUEST, VERS UN ENVIRONNEMENT VIRTUEL DE RECHERCHE (VRE) ORIENTÉ SCIENCES DE LA VIE? Intervenant(s) : Yvan Le Bras, Olivier Collin
 E-BIOGENOUEST, VERS UN ENVIRONNEMENT VIRTUEL DE RECHERCHE (VRE) ORIENTÉ SCIENCES DE LA VIE? Intervenant(s) : Yvan Le Bras, Olivier Collin E-BIOGENOUEST Programme fédérateur Biogenouest co-financé par les
E-BIOGENOUEST, VERS UN ENVIRONNEMENT VIRTUEL DE RECHERCHE (VRE) ORIENTÉ SCIENCES DE LA VIE? Intervenant(s) : Yvan Le Bras, Olivier Collin E-BIOGENOUEST Programme fédérateur Biogenouest co-financé par les
Analyse des données de séquençage massif par des méthodes phylogénétiques
 Analyse des données de séquençage massif par des méthodes phylogénétiques Roux S., Taib N., Mangot J.F., Hugoni M., Mary I., Ravet V., Bronner G., Enault F., Debroas D. Équipe Microbiologie de l'environnement
Analyse des données de séquençage massif par des méthodes phylogénétiques Roux S., Taib N., Mangot J.F., Hugoni M., Mary I., Ravet V., Bronner G., Enault F., Debroas D. Équipe Microbiologie de l'environnement
Introduction au Data-Mining
 Introduction au Data-Mining Alain Rakotomamonjy - Gilles Gasso. INSA Rouen -Département ASI Laboratoire PSI Introduction au Data-Mining p. 1/25 Data-Mining : Kèkecé? Traduction : Fouille de données. Terme
Introduction au Data-Mining Alain Rakotomamonjy - Gilles Gasso. INSA Rouen -Département ASI Laboratoire PSI Introduction au Data-Mining p. 1/25 Data-Mining : Kèkecé? Traduction : Fouille de données. Terme
Dossier justificatif des travaux de R&D déclarés au titre du CIR
 Dossier justificatif des travaux de R&D déclarés au titre du CIR I PRÉSENTATION DE LA SOCIETE Présenter l activité de la société en quelques lignes ou au moyen d une plaquette publicitaire et replacer
Dossier justificatif des travaux de R&D déclarés au titre du CIR I PRÉSENTATION DE LA SOCIETE Présenter l activité de la société en quelques lignes ou au moyen d une plaquette publicitaire et replacer
AA-SO5 KIDA/GSOV/VAMDC
 AA-SO5 Centres de traitement et d archivage de données KIDA - 2 mars 2015 AA-SO5 KIDA/GSOV/VAMDC Contexte général L observation des molécules dans le milieu interstellaire, mais aussi dans les atmosphères
AA-SO5 Centres de traitement et d archivage de données KIDA - 2 mars 2015 AA-SO5 KIDA/GSOV/VAMDC Contexte général L observation des molécules dans le milieu interstellaire, mais aussi dans les atmosphères
Journée SITG, Genève 15 octobre 2013. Nicolas Lachance-Bernard M.ATDR Doctorant, Laboratoire de systèmes d information géographique
 Monitorint spatio-temporel intégré de la mobilité urbaine Monitoring spatio-temporel de l ADN urbain Une réponse aux défis, problèmes, enjeux et risques des milieux urbains Nicolas Lachance-Bernard M.ATDR
Monitorint spatio-temporel intégré de la mobilité urbaine Monitoring spatio-temporel de l ADN urbain Une réponse aux défis, problèmes, enjeux et risques des milieux urbains Nicolas Lachance-Bernard M.ATDR
Les environnements de calcul distribué
 2 e Atelier CRAG, 3 au 8 Décembre 2012 Par Blaise Omer YENKE IUT, Université de Ngaoundéré, Cameroun. 4 décembre 2012 1 / 32 Calcul haute performance (HPC) High-performance computing (HPC) : utilisation
2 e Atelier CRAG, 3 au 8 Décembre 2012 Par Blaise Omer YENKE IUT, Université de Ngaoundéré, Cameroun. 4 décembre 2012 1 / 32 Calcul haute performance (HPC) High-performance computing (HPC) : utilisation
SYLLABUS MASTER. Mention BIOCHIMIE BIOTECHNOLOGIES
 SYLLABUS MASTER Mention BIOCHIMIE BIOTECHNOLOGIES Année universitaire 2011/2012 Le Master de Biochimie-BioTechnologies propose une association de formations professionnelles et de recherche sur les aspects
SYLLABUS MASTER Mention BIOCHIMIE BIOTECHNOLOGIES Année universitaire 2011/2012 Le Master de Biochimie-BioTechnologies propose une association de formations professionnelles et de recherche sur les aspects
Les OGM. 5 décembre 2008. Nicole Mounier
 Les OGM 5 décembre 2008 Nicole Mounier Université Claude Bernard Lyon 1 CGMC, bâtiment Gregor Mendel 43, boulevard du 11 Novembre 1918 69622 Villeurbanne Cedex OGM Organismes Génétiquement Modifiés Transfert
Les OGM 5 décembre 2008 Nicole Mounier Université Claude Bernard Lyon 1 CGMC, bâtiment Gregor Mendel 43, boulevard du 11 Novembre 1918 69622 Villeurbanne Cedex OGM Organismes Génétiquement Modifiés Transfert
Comment réaliser physiquement un ordinateur quantique. Yves LEROYER
 Comment réaliser physiquement un ordinateur quantique Yves LEROYER Enjeu: réaliser physiquement -un système quantique à deux états 0 > ou 1 > -une porte à un qubitconduisant à l état générique α 0 > +
Comment réaliser physiquement un ordinateur quantique Yves LEROYER Enjeu: réaliser physiquement -un système quantique à deux états 0 > ou 1 > -une porte à un qubitconduisant à l état générique α 0 > +
Solutions logicielles de gestion énergétique coopérante smart building in smart grid : Exemple CANOPEA. Xavier Brunotte info@vesta-system.
 Solutions logicielles de gestion énergétique coopérante smart building in smart grid : Exemple CANOPEA Xavier Brunotte [email protected] Agenda Vesta-System et principe de son gestionnaire de son gestionnaire
Solutions logicielles de gestion énergétique coopérante smart building in smart grid : Exemple CANOPEA Xavier Brunotte [email protected] Agenda Vesta-System et principe de son gestionnaire de son gestionnaire
Détection et prise en charge de la résistance aux antirétroviraux
 Détection et prise en charge de la résistance aux antirétroviraux Jean Ruelle, PhD AIDS Reference Laboratory, UCLouvain, Bruxelles Corata 2011, Namur, 10 juin 2011 Laboratoires de référence SIDA (Belgique)
Détection et prise en charge de la résistance aux antirétroviraux Jean Ruelle, PhD AIDS Reference Laboratory, UCLouvain, Bruxelles Corata 2011, Namur, 10 juin 2011 Laboratoires de référence SIDA (Belgique)
