Classification phylogénétique du vivant. Classification cladistique vs phénétique
|
|
|
- Michel Marois
- il y a 9 ans
- Total affichages :
Transcription
1 Classification phylogénétique du vivant Classification cladistique vs phénétique
2
3 phénétique Quantification des similitudes Ressemblance générale chiffrée => distance Particulièrement adapté aux données moléculaires ou chaque position dans la séquence correspond à un caractère
4
5 Cinq étapes de l analyse phylogénétique 1. Choix du jeu de données Une bonne connaissance des séquences que l on analyse S assurer de la validité du jeu de donnée (qualité des séquences et cohérence du JDD) 2. Alignement des séquences Obtenir un bon alignement Tester différents méthodes et revenir à la main sur les résultats 3. Détermination du modèle de substitution 4. Construction des arbres 5. Evaluation des arbres
6 Les éléments d une phylogénie Les caractères Le jeu de données Les banques Les alignements Les arbres
7 Description et codage des états Présence absence : +/-; 0/1; a/b Etats multiples : Les 20 acides aminés Les 4 nucléotides A,T,C,G Nb de répétitions en tandem (microsatellites) Morphologie (a, b, c, d,..)
8 Morphologie vs. Données moléculaires Les vautours du vieux et du nouveau monde semblent être étroitement liés sur la base de leur morphologie Les données moléculaires indiquent que les vautours du vieux monde sont liées à des oiseaux de proie (faucons, éperviers, etc), tandis que les vautours du Nouveau Monde sont plus étroitement liés à des cigognes C est un exemple de convergence évolutive
9 Le jeu de données Au début les modes de classifications des espèces étaient: Les comparaisons morphologiques Les comparaisons comportementales Les répartitions géographiques Aujourd hui les phylogénies sont obtenues à partir: des séquences moléculaires (phylogénie moléculaire) : ADN, ARN, Protéines, Codons des caractères discrets (présence, absence, 0, 1) des fréquences des gènes des traits quantitatifs des sites de restriction, RFLP des microsatellites, SNP
10 Données moléculaires Les séquences d ADN présentent beaucoup d avantages face aux caractères de taxonomie morphologiques: L état des caractères peut être déterminé sans ambiguïté Un grand nombre de caractère peuvent être pris en compte pour chaque individu Inconvénients: Peu d états donc possibilité de mutations silencieuses (homoplasie) Arbre de gène vs Arbre de génome (cad arbre de espèces) Alignements de qualité difficile à obtenir
11 Choix du marqueur il faut choisir le marqueur moléculaire approprié au groupe taxonomique étudié. Critères du choix d'un marqueur: universalité structure conservée absence de transfert génétique taux d évolution approprié absence de biais sélectif Quelques exemples: phylogénie de bactéries (16S rdna) phylogénie d eucaryotes (18S rdna, actine, EF1, RPB1 (RNA polymerase)) phylogénie de plantes (rbcl(ribulose carboxylase),18s rdna) Phylogénie d animaux niveau phylum, classe, ordre : (18S rdna, génome mt) niveau famille : (RAG2 (recombination activating gene 2), 12S, 16S mt) niveau genre : (ITS, protéines mt) niveau intra-spécifique : (D-Loop, introns)
12 Eviter les séquences incomplètes Eviter les xénologues (transfert latéraux) Eviter les séquences recombinantes (2 ancêtres) Eviter les grandes familles complexes (répétitions et nombres de domaines importants) Ajouter un groupe externe (outgroup) ADN ou protéines? Quand cela est possible travailler préférentiellement avec des alignement de protéines en particulier lorsque les séquences d ADN diffèrent de plus 70%. Si les séquences protéiques sont trop proches revenir à l ADN.
13 Banques de données
14 Alignement L alignement est une étape cruciale qui permet de choisir les sites qui seront utilisés dans les analyses phylogénétiques. But : S assurer que chacun des sites choisis est homologue Pour s assurer de l homologie d un site: la structure primaire des séquences (ordre des nucléotides) la structure secondaire des séquences (gènes ribosomiques) la séquence en acides aminés (gènes codant pour des protéines)
15 Les résidus (nucléotides, acides-aminés) sont superposés de façon à maximiser la similarité entre les séquences. Mutations : Substitution (mismatch) Insertion Délétion Insertions ou délétions : indels(gap)
16 Pour le biologiste, généralement, le bon alignement est celui qui représente le scénario évolutif le plus probable
17 Exemple: identité = 1 mismatch= 0 gap = -1 Score = 10-4 = 6
18 Transition vs transversion Transition: A <-> G T<-> C Transversions : autres substitutions p(transition) > p(transversion)
19 Code génétique Asp (GAC, GAU) Tyr (UAC, UAU) : 1 mutation Asp(GAC, GAU) Cys(UGC, UGU) : 2 mutations Asp(GAC, GAU) Trp(UGG) : 3 mutations Propriétés physico-chimiques des acidesaminés (acidité, hydrophobicité, encombrement stérique, etc.)
20 Les arbres Les méthodes cladistiques et phénétique construisent un arbre (dendrogramme) Cladogramme - un dendrogramme exprimant les relations phylogénétiques entre taxa et construit à partir de l'analyse cladistique; phénogramme- un dendrogramme obtenu par méthodes de distance où les relations entre taxa expriment des degrés de similitude globale; phylogramme- un dendrogramme dont la longueur des branches est proportionnelle au nombre de changements évolutifs
21 Il existe 3 approches : L'approche cladistique cherche en particulier à déterminer les caractères propres à une branche, qui «signent» un apparentement. L'approche phénétique, une classification basée uniquement sur des mesures de distance entre taxons (évaluées par exemple en comptant les différences de séquences d'adn) sans chercher à faire une interprétation phylogénétique. L approche probabiliste qui construit des arbres phylogénétiques en utilisant des modèles d évolution des caractères (le plus souvent moléculaires, mais pas obligatoirement).
22 Phénétique vs cladistique L'approche phénétique (taxonomie numérique) se veut complètement objective. C'est une approche très quantitative dans laquelle tous les traits (qu'ils soient homologues ou non) sont traités également. Cette méthode se révèle peu pertinente lorsqu'on l'applique aux caractères morphologiques en raison des analogies : convergence évolutives. Elle s applique préférentiellement sur des caractères moléculaires où le nombre de caractères pris en compte est important La cladistique hiérarchise les caractères comparés. Ne sont en fait regroupés dans un même taxon que les êtres vivants qui partagent des caractères homologues: partage d une ascendance commune. Les homologies sont en fait vues comme des innovations évolutives partagées : synapomorphies
23 Méthodologie
24 Choix de la méthode d analyse phylogénétique
25 Méthode de maximum de parcimonie Hypothèses de base Les séquences en présence ont évolué à partir d'une séquence ancestrale commune grâce à un processus de mutation-sélection. Les différents sites (site=position occupée par un acide aminé ou une base) de la séquence évoluent indépendamment les uns des autres : la séquence peut être considérée comme une suite de caractères non ordonnés. Les lignées se différencient les unes des autres d'une façon autonome. La vitesse d'évolution est lente et constante au cours du temps
26 Cette méthode : ne prend en considération que les sites informatifs. ne fait pas de corrections pour les substitutions multiples. ne donne aucune information sur la longueur des branches. est connue pour être très sensible au biais des codons Méthode Reconstruire les événements évolutifs pour une phylogénie donnée Chercher parmi toutes les phylogénies possibles celle qui minimise le nombre d événements évolutifs
27 homoplasie Homoplasie = se dit de sites présentant des états moléculaires identiques résultant d événements évolutifs différents, tel que des évènements de convergence ou de réversion => La présence d homoplasie conduit à sousestimer le nombre total de mutations s étant produites au cours du temps
28 Tester les arbres Te s t e r l e s a r b r e s => tester la robustesse des nœuds => évaluer le rapport entre homoplasie et homologie Calcul de l indice de cohérence
29 Pb : il faut tester tous les arbres et les évalués un par un Le nombre d arbre augmente de manière exponentielle lorsque l on ajoute des taxons 10 taxons => 2 millions d arbres..
30 Méthode recherche arbre plus parcimonieux Exhaustive on obtient un score pour chaque arbre possible. On est sûr de trouver le meilleur arbre. Pratique seulement pour des petits jeux de données (<10 taxa). Heuristique on utilise des algorithmes pour parcourir l ensemble de l espace des arbres mais pas chaque arbre Stochastique recherche aléatoire dans l espace des arbres. Algorithmes comme le simulated annealing ou les algorithmes génetiques
31 Approche heuristique Principe : Partir d un arbre initial Petits réarrangements de branches successifs => Exploration des arbres voisins Si un des voisins est «meilleur» on le conserve On répète cette opération jusqu à ce qu on trouve un arbre pour lequel aucun réarrangement ne donne de meilleur arbre
32 Distances évolutives La notion de distance découle naturellement de celle de similitude : Plus la similitude entre deux séquences est forte, plus la distance entre elles δ est faible Elles reflètent la quantité d évolution survenue pendant un temps Elles ne sont pas une simple fonction de temps ex: deux séquences qui sont issues d un groupe frère dans une phylogénie sont séparées par le même intervalle de temps t, mais peuvent avoir subit une quantité d évolution différente
33 De façon basique, les séquences d ADN ne sont pas très informatives sur l histoire évolutive. Pour chaque site, le nombre maximal de différences est 1. Il existe seulement 4 états. Donc, si il y a plus d une substitution, nous perdons la substitution passée. Les substitutions multiples cachent l histoire évolutive entre les séquences.
34 Distances évolutives
35
36 Méthodes de distance Principe La configuration de l'arbre traduit avant tout le degré de similarité, sans nécessairement tenter de refléter l'évolution moléculaire sous-jacente (l'histoire) aux phénotypes observés. Données initiales Matrice de distances entre séquences prises deux à deux Compression de l'information en une seule valeur (=distance) : tous les sites sont traités de manière équivalente (perte d'information). Hypothèses de base Les distances ont été mesurées indépendamment. Les distances s'additionnent. Méthode Ces programmes procèdent par regroupement successifs (clusterisation),depuis la paire des séquences les plus proches aux plus éloignées. Résultat Un seul arbre sans racine Avantages-Inconvénients Ces méthodes sont rapides et donnent de bons résultats pour des séquences proches. Mais les sites sont traités de manière équivalente : ces méthodes ne sont pas applicables aux séquences très divergentes
37 Méthode UPGMA UPGMA (Unweight Pair Group Method with Arithmetic mean) C est un algorithme itératif de clustering, à chaque étape on crée un nouveau cluster regroupant deux clusters proches et on rajoute un nœud à l arbre. Les branches évoluent à la même vitesse (taux de mutation identiques sur les différentes branches (lignée)) => hypothèse d une horloge moléculaire
38 Ex de matrice
39 =Connecter phoque et otarie dans l arbre => D phoque-noeud= D otarie-nœud = D phoque-otarie/2=24/2 =12
40 Calculer la distance entre le nouveau groupe (ij) et tous les autres groupes en utilisant Dij,k= ni x Dik/ (ni+ nj) + nj x Djk/ (ni+ nj) Calcul de la distance entre le nouveau groupe phoque-otarie et tous les autres groupes Pour le chien : (50x1 + 48x1)/(1+1) = 49
41 Étape similiaire pour proposer un nouveau regroupement
42
43 BILAN UPGMA Méthode extrêmement simple de reconstruire des arbres phylogénétiques Est très critiquée dans la littérature en phylogénie à cause de l hypothèse de l égalité des taux d évolution entre les lignées. N est presque plus utilisée Peut être réaliste si on étudie des espèces très proches Donne des résultats faux si les distances de la matrice n obéissent pas au critère d horloge moléculaire
44 Méthode NJ NEIGHBOR-JOINING (Saitou et Nei, 1987 ) Cette méthode tente de corriger la méthode UPGMA afin d'autoriser un taux de mutation différent sur les branches Pour cela, la matrice de distances est corrigée afin de prendre en compte la divergence moyenne de chacune des séquences avec les autres.
45 Méthode maximum de vraissemblance
46 Méthode maximum de vraissemblance Idée de base Introduit par Edwards et Cavalli-Sforza (1964) pour des données de fréquences de gènes Application à des données moléculaires par Neyman (1971) Élargissement par Kashyap et Subas (1974) et Felsenstein (1981) Étant donné un modèle d évolution, on peut estimer une phylogénie avec des méthodes statistiques comme le maximum de vraisemblance (ou des méthodes bayesiennes). La vraisemblance d'un arbre estime la probabilité d observer des données (séquences + modèle de l'évolution) selon l'hypothèse qu'il véhicule (topologie + longueurs des branches). On choisit l arbre qui maximise la vraisemblance (qui a la plus forte probabilité d avoir conduit aux données
47 Intérêts des différentes méthodes Distance Méthode très rapide Correction des distances possibles selon les modèles d évolution Avec corrections peut être proche de la performance de MV Parcimonie Utilisée avec données morphologiques et fossiles Principe plus simple et critère à fondement biologique Peut converger avec MV si les données ne sont pas trop divergentes Maximum de vraisemblance Méthode flexible, robuste et consistante Tient compte de la complexité du processus évolutif Tient compte de la longueur des branches pour estimer la vraisemblance
48 Distance Sous estime les substitutions et la longueur des branches Problème d attraction des branches longues et courtes Perte d information dans les distances Parcimonie Sous estime les substitutions et la longueur des branches Pas basée sur un modèle d évolution Problème d attraction des branches longues et courtes Utilise seulement les sites informatifs Très affectée par les convergences et réversions des caractères Maximum de vraisemblance Méthode coûteuse (lente) Dépendant du bon choix de modèle d évolution Peut tomber dans des régions de maxima locaux
MABioVis. Bio-informatique et la
 MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
CHAPITRE 3 LA SYNTHESE DES PROTEINES
 CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST.
 La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
Objectifs. Clustering. Principe. Applications. Applications. Cartes de crédits. Remarques. Biologie, Génomique
 Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
La reconnaissance moléculaire: la base du design rationnel Modélisation moléculaire: Introduction Hiver 2006
 La reconnaissance moléculaire: la base du design rationnel En 1890 Emil Fisher a proposé le modèle "serrure et clé" pour expliquer la façon de fonctionner des systèmes biologiques. Un substrat rentre et
La reconnaissance moléculaire: la base du design rationnel En 1890 Emil Fisher a proposé le modèle "serrure et clé" pour expliquer la façon de fonctionner des systèmes biologiques. Un substrat rentre et
Gènes Diffusion - EPIC 2010
 Gènes Diffusion - EPIC 2010 1. Contexte. 2. Notion de génétique animale. 3. Profil de l équipe plateforme. 4. Type et gestion des données biologiques. 5. Environnement Matériel et Logiciel. 6. Analyses
Gènes Diffusion - EPIC 2010 1. Contexte. 2. Notion de génétique animale. 3. Profil de l équipe plateforme. 4. Type et gestion des données biologiques. 5. Environnement Matériel et Logiciel. 6. Analyses
Génétique et génomique Pierre Martin
 Génétique et génomique Pierre Martin Principe de la sélections Repérage des animaux intéressants X Accouplements Programmés Sélection des meilleurs mâles pour la diffusion Index diffusés Indexation simultanée
Génétique et génomique Pierre Martin Principe de la sélections Repérage des animaux intéressants X Accouplements Programmés Sélection des meilleurs mâles pour la diffusion Index diffusés Indexation simultanée
Détection des duplications en tandem au niveau nucléique à l'aide de la théorie des flots
 Université Toulouse 3 Paul Sabatier(UT3 Paul Sabatier) Informatique Spécialité Bioinformatique Eric AUDEMARD lundi 28 novembre 2011 Détection des duplications en tandem au niveau nucléique à l'aide de
Université Toulouse 3 Paul Sabatier(UT3 Paul Sabatier) Informatique Spécialité Bioinformatique Eric AUDEMARD lundi 28 novembre 2011 Détection des duplications en tandem au niveau nucléique à l'aide de
ALGORITHME GENETIQUE ET MODELE DE SIMULATION POUR L'ORDONNANCEMENT D'UN ATELIER DISCONTINU DE CHIMIE
 ALGORITHME GENETIQUE ET MODELE DE SIMULATION POUR L'ORDONNANCEMENT D'UN ATELIER DISCONTINU DE CHIMIE P. Baudet, C. Azzaro-Pantel, S. Domenech et L. Pibouleau Laboratoire de Génie Chimique - URA 192 du
ALGORITHME GENETIQUE ET MODELE DE SIMULATION POUR L'ORDONNANCEMENT D'UN ATELIER DISCONTINU DE CHIMIE P. Baudet, C. Azzaro-Pantel, S. Domenech et L. Pibouleau Laboratoire de Génie Chimique - URA 192 du
Identification de nouveaux membres dans des familles d'interleukines
 Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
Identification de nouveaux membres dans des familles d'interleukines Nicolas Beaume Jérôme Mickolajczak Gérard Ramstein Yannick Jacques 1ère partie : Définition de la problématique Les familles de gènes
Big data et sciences du Vivant L'exemple du séquençage haut débit
 Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard [email protected] INRA - MIAT - Plate-forme
Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard [email protected] INRA - MIAT - Plate-forme
Jean-Philippe Préaux http://www.i2m.univ-amu.fr/~preaux
 Colonies de fourmis Comment procèdent les colonies de fourmi pour déterminer un chemin presque géodésique de la fourmilière à un stock de nourriture? Les premières fourmis se déplacent au hasard. Les fourmis
Colonies de fourmis Comment procèdent les colonies de fourmi pour déterminer un chemin presque géodésique de la fourmilière à un stock de nourriture? Les premières fourmis se déplacent au hasard. Les fourmis
Séquence 6. Mais ces espèces pour autant ne sont pas identiques et parfois d ailleurs ne se ressemblent pas vraiment.
 Sommaire Séquence 6 Nous avons vu dans les séances précédentes qu au cours des temps géologiques des espèces différentes se sont succédé, leur apparition et leur disparition étant le résultat de modifications
Sommaire Séquence 6 Nous avons vu dans les séances précédentes qu au cours des temps géologiques des espèces différentes se sont succédé, leur apparition et leur disparition étant le résultat de modifications
Travaux dirigés de Microbiologie Master I Sciences des Génomes et des Organismes Janvier 2015
 Andrew Tolonen [email protected] Travaux dirigés de Microbiologie Master I Sciences des Génomes et des Organismes Janvier 2015 A- Généralités I- La vie sur terre telle que nous la connaissons ne
Andrew Tolonen [email protected] Travaux dirigés de Microbiologie Master I Sciences des Génomes et des Organismes Janvier 2015 A- Généralités I- La vie sur terre telle que nous la connaissons ne
Formavie 2010. 2 Différentes versions du format PDB...3. 3 Les champs dans les fichiers PDB...4. 4 Le champ «ATOM»...5. 6 Limites du format PDB...
 Formavie 2010 Les fichiers PDB Les fichiers PDB contiennent les informations qui vont permettre à des logiciels de visualisation moléculaire (ex : RasTop ou Jmol) d afficher les molécules. Un fichier au
Formavie 2010 Les fichiers PDB Les fichiers PDB contiennent les informations qui vont permettre à des logiciels de visualisation moléculaire (ex : RasTop ou Jmol) d afficher les molécules. Un fichier au
Projet SINF2275 «Data mining and decision making» Projet classification et credit scoring
 Projet SINF2275 «Data mining and decision making» Projet classification et credit scoring Année académique 2006-2007 Professeurs : Marco Saerens Adresse : Université catholique de Louvain Information Systems
Projet SINF2275 «Data mining and decision making» Projet classification et credit scoring Année académique 2006-2007 Professeurs : Marco Saerens Adresse : Université catholique de Louvain Information Systems
Dr E. CHEVRET UE2.1 2013-2014. Aperçu général sur l architecture et les fonctions cellulaires
 Aperçu général sur l architecture et les fonctions cellulaires I. Introduction II. Les microscopes 1. Le microscope optique 2. Le microscope à fluorescence 3. Le microscope confocal 4. Le microscope électronique
Aperçu général sur l architecture et les fonctions cellulaires I. Introduction II. Les microscopes 1. Le microscope optique 2. Le microscope à fluorescence 3. Le microscope confocal 4. Le microscope électronique
Analyse de la variance Comparaison de plusieurs moyennes
 Analyse de la variance Comparaison de plusieurs moyennes Biostatistique Pr. Nicolas MEYER Laboratoire de Biostatistique et Informatique Médicale Fac. de Médecine de Strasbourg Mars 2011 Plan 1 Introduction
Analyse de la variance Comparaison de plusieurs moyennes Biostatistique Pr. Nicolas MEYER Laboratoire de Biostatistique et Informatique Médicale Fac. de Médecine de Strasbourg Mars 2011 Plan 1 Introduction
Analyse des données de séquençage massif par des méthodes phylogénétiques
 Analyse des données de séquençage massif par des méthodes phylogénétiques Roux S., Taib N., Mangot J.F., Hugoni M., Mary I., Ravet V., Bronner G., Enault F., Debroas D. Équipe Microbiologie de l'environnement
Analyse des données de séquençage massif par des méthodes phylogénétiques Roux S., Taib N., Mangot J.F., Hugoni M., Mary I., Ravet V., Bronner G., Enault F., Debroas D. Équipe Microbiologie de l'environnement
23. Interprétation clinique des mesures de l effet traitement
 23. Interprétation clinique des mesures de l effet traitement 23.1. Critères de jugement binaires Plusieurs mesures (indices) sont utilisables pour quantifier l effet traitement lors de l utilisation d
23. Interprétation clinique des mesures de l effet traitement 23.1. Critères de jugement binaires Plusieurs mesures (indices) sont utilisables pour quantifier l effet traitement lors de l utilisation d
La Classification des espèces
 La Classification des espèces Professeur Patrice Francour [email protected] http://www.unice.fr/leml/francour_internet/ EA 3156 «Gestion de la Biodiversité». Université de Nice-Sophia Antipolis. Parc Valrose.
La Classification des espèces Professeur Patrice Francour [email protected] http://www.unice.fr/leml/francour_internet/ EA 3156 «Gestion de la Biodiversité». Université de Nice-Sophia Antipolis. Parc Valrose.
Document d orientation sur les allégations issues d essais de non-infériorité
 Document d orientation sur les allégations issues d essais de non-infériorité Février 2013 1 Liste de contrôle des essais de non-infériorité N o Liste de contrôle (les clients peuvent se servir de cette
Document d orientation sur les allégations issues d essais de non-infériorité Février 2013 1 Liste de contrôle des essais de non-infériorité N o Liste de contrôle (les clients peuvent se servir de cette
Ebauche Rapport finale
 Ebauche Rapport finale Sommaire : 1 - Introduction au C.D.N. 2 - Définition de la problématique 3 - Etat de l'art : Présentatio de 3 Topologies streaming p2p 1) INTRODUCTION au C.D.N. La croissance rapide
Ebauche Rapport finale Sommaire : 1 - Introduction au C.D.N. 2 - Définition de la problématique 3 - Etat de l'art : Présentatio de 3 Topologies streaming p2p 1) INTRODUCTION au C.D.N. La croissance rapide
Raisonnement probabiliste
 Plan Raisonnement probabiliste IFT-17587 Concepts avancés pour systèmes intelligents Luc Lamontagne Réseaux bayésiens Inférence dans les réseaux bayésiens Inférence exacte Inférence approximative 1 2 Contexte
Plan Raisonnement probabiliste IFT-17587 Concepts avancés pour systèmes intelligents Luc Lamontagne Réseaux bayésiens Inférence dans les réseaux bayésiens Inférence exacte Inférence approximative 1 2 Contexte
L apprentissage automatique
 L apprentissage automatique L apprentissage automatique L'apprentissage automatique fait référence au développement, à l analyse et à l implémentation de méthodes qui permettent à une machine d évoluer
L apprentissage automatique L apprentissage automatique L'apprentissage automatique fait référence au développement, à l analyse et à l implémentation de méthodes qui permettent à une machine d évoluer
Déroulement d un projet en DATA MINING, préparation et analyse des données. Walid AYADI
 1 Déroulement d un projet en DATA MINING, préparation et analyse des données Walid AYADI 2 Les étapes d un projet Choix du sujet - Définition des objectifs Inventaire des données existantes Collecte, nettoyage
1 Déroulement d un projet en DATA MINING, préparation et analyse des données Walid AYADI 2 Les étapes d un projet Choix du sujet - Définition des objectifs Inventaire des données existantes Collecte, nettoyage
à moyen Risque moyen Risq à élevé Risque élevé Risq e Risque faible à moyen Risq Risque moyen à élevé Risq
 e élevé Risque faible Risq à moyen Risque moyen Risq à élevé Risque élevé Risq e Risque faible à moyen Risq Risque moyen à élevé Risq L e s I n d i c e s F u n d a t a é Risque Les Indices de faible risque
e élevé Risque faible Risq à moyen Risque moyen Risq à élevé Risque élevé Risq e Risque faible à moyen Risq Risque moyen à élevé Risq L e s I n d i c e s F u n d a t a é Risque Les Indices de faible risque
Introduction au Data-Mining
 Introduction au Data-Mining Alain Rakotomamonjy - Gilles Gasso. INSA Rouen -Département ASI Laboratoire PSI Introduction au Data-Mining p. 1/25 Data-Mining : Kèkecé? Traduction : Fouille de données. Terme
Introduction au Data-Mining Alain Rakotomamonjy - Gilles Gasso. INSA Rouen -Département ASI Laboratoire PSI Introduction au Data-Mining p. 1/25 Data-Mining : Kèkecé? Traduction : Fouille de données. Terme
La classification automatique de données quantitatives
 La classification automatique de données quantitatives 1 Introduction Parmi les méthodes de statistique exploratoire multidimensionnelle, dont l objectif est d extraire d une masse de données des informations
La classification automatique de données quantitatives 1 Introduction Parmi les méthodes de statistique exploratoire multidimensionnelle, dont l objectif est d extraire d une masse de données des informations
LA MITOSE CUEEP - USTL DÉPARTEMENT SCIENCES BAHIJA DELATTRE
 Biologie LA MITOSE CUEEP - USTL DÉPARTEMENT SCIENCES BAHIJA DELATTRE Février 2006 I. L'INTRODUCTION Chaque cellule d'un organisme supérieur provient de la multiplication d'une cellule préexistante (cellule
Biologie LA MITOSE CUEEP - USTL DÉPARTEMENT SCIENCES BAHIJA DELATTRE Février 2006 I. L'INTRODUCTION Chaque cellule d'un organisme supérieur provient de la multiplication d'une cellule préexistante (cellule
Conférence technique internationale de la FAO
 Décembre 2009 ABDC-10/7.2 F Conférence technique internationale de la FAO Biotechnologies agricoles dans les pays en développement: choix et perspectives pour les cultures, les forêts, l élevage, les pêches
Décembre 2009 ABDC-10/7.2 F Conférence technique internationale de la FAO Biotechnologies agricoles dans les pays en développement: choix et perspectives pour les cultures, les forêts, l élevage, les pêches
GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010
 GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010 Analyse de la diversité moléculaire des régions génomiques de 30 gènes du développement méristématique dans une core collection
GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010 Analyse de la diversité moléculaire des régions génomiques de 30 gènes du développement méristématique dans une core collection
Séquence 2. L expression du patrimoine génétique. Sommaire
 Séquence 2 L expression du patrimoine génétique Sommaire 1. La synthèse des protéines 2. Phénotypes, génotypes et environnement Synthèse de la séquence 2 Exercices de la séquence 2 Glossaire des séquences
Séquence 2 L expression du patrimoine génétique Sommaire 1. La synthèse des protéines 2. Phénotypes, génotypes et environnement Synthèse de la séquence 2 Exercices de la séquence 2 Glossaire des séquences
Objectifs : piloter l organisation à travers des indicateurs (regroupés dans un tableau de bord), et informer des résultats la hiérarchie.
 C HAPI TRE 8 Tableau de bord et reporting Objectifs : piloter l organisation à travers des indicateurs (regroupés dans un tableau de bord), et informer des résultats la hiérarchie. 1 Principes A Le tableau
C HAPI TRE 8 Tableau de bord et reporting Objectifs : piloter l organisation à travers des indicateurs (regroupés dans un tableau de bord), et informer des résultats la hiérarchie. 1 Principes A Le tableau
UE6 - Cycle de vie du médicament : Conception rationnelle
 UE6 - Cycle de vie du médicament : Conception rationnelle Dr. Raphaël Terreux Faculté de Pharmacie (ISPB) Département pédagogique des Sciences Physico-Chimiques et Pharmacie Galénique 8 avenue Rockefeller,
UE6 - Cycle de vie du médicament : Conception rationnelle Dr. Raphaël Terreux Faculté de Pharmacie (ISPB) Département pédagogique des Sciences Physico-Chimiques et Pharmacie Galénique 8 avenue Rockefeller,
 Optimisation Combinatoire et Colonies de Fourmis Nicolas Monmarche April 21, 1999 Sommaire Inspiration biologiques Ant Colony Optimization Applications TSP QAP Flow Shop Problemes dynamiques 1 Historique
Optimisation Combinatoire et Colonies de Fourmis Nicolas Monmarche April 21, 1999 Sommaire Inspiration biologiques Ant Colony Optimization Applications TSP QAP Flow Shop Problemes dynamiques 1 Historique
Analyse stochastique de la CRM à ordre partiel dans le cadre des essais cliniques de phase I
 Analyse stochastique de la CRM à ordre partiel dans le cadre des essais cliniques de phase I Roxane Duroux 1 Cadre de l étude Cette étude s inscrit dans le cadre de recherche de doses pour des essais cliniques
Analyse stochastique de la CRM à ordre partiel dans le cadre des essais cliniques de phase I Roxane Duroux 1 Cadre de l étude Cette étude s inscrit dans le cadre de recherche de doses pour des essais cliniques
Modélisation multi-agents - Agents réactifs
 Modélisation multi-agents - Agents réactifs Syma cursus CSI / SCIA Julien Saunier - [email protected] Sources www-lih.univlehavre.fr/~olivier/enseignement/masterrecherche/cours/ support/algofourmis.pdf
Modélisation multi-agents - Agents réactifs Syma cursus CSI / SCIA Julien Saunier - [email protected] Sources www-lih.univlehavre.fr/~olivier/enseignement/masterrecherche/cours/ support/algofourmis.pdf
Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant
 Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant Parcours: Master 1 : Bioinformatique et biologie des Systèmes dans le Master
Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant Parcours: Master 1 : Bioinformatique et biologie des Systèmes dans le Master
chargement d amplitude variable à partir de mesures Application à l approche fiabiliste de la tolérance aux dommages Modélisation stochastique d un d
 Laboratoire de Mécanique et Ingénieriesnieries EA 3867 - FR TIMS / CNRS 2856 ER MPS Modélisation stochastique d un d chargement d amplitude variable à partir de mesures Application à l approche fiabiliste
Laboratoire de Mécanique et Ingénieriesnieries EA 3867 - FR TIMS / CNRS 2856 ER MPS Modélisation stochastique d un d chargement d amplitude variable à partir de mesures Application à l approche fiabiliste
1 les caractères des êtres humains.
 Quelques rappels des classes précédentes ACTIVITÉ livre pages 8 et 9 : apprendre le bilan de la page 9 Les êtres vivants sont répartis en espèces. Chaque être vivant est formé de cellules. schéma d une
Quelques rappels des classes précédentes ACTIVITÉ livre pages 8 et 9 : apprendre le bilan de la page 9 Les êtres vivants sont répartis en espèces. Chaque être vivant est formé de cellules. schéma d une
VI. Tests non paramétriques sur un échantillon
 VI. Tests non paramétriques sur un échantillon Le modèle n est pas un modèle paramétrique «TESTS du CHI-DEUX» : VI.1. Test d ajustement à une loi donnée VI.. Test d indépendance de deux facteurs 96 Différentes
VI. Tests non paramétriques sur un échantillon Le modèle n est pas un modèle paramétrique «TESTS du CHI-DEUX» : VI.1. Test d ajustement à une loi donnée VI.. Test d indépendance de deux facteurs 96 Différentes
Bases moléculaires des mutations Marc Jeanpierre
 Bases moléculaires des mutations Marc Jeanpierre Chaque enfant qui naît hérite de 10 à 30 nouvelles mutations ponctuelles. L essentiel des ces mutations sont heureusement des variations neutres de séquence
Bases moléculaires des mutations Marc Jeanpierre Chaque enfant qui naît hérite de 10 à 30 nouvelles mutations ponctuelles. L essentiel des ces mutations sont heureusement des variations neutres de séquence
Structure quantique cohérente et incohérente de l eau liquide
 Structure quantique cohérente et incohérente de l eau liquide Prof. Marc HENRY Chimie Moléculaire du Solide Institut Le Bel, 4, Rue Blaise Pascal 67070 Strasbourg Cedex, France Tél: 03.68.85.15.00 e-mail:
Structure quantique cohérente et incohérente de l eau liquide Prof. Marc HENRY Chimie Moléculaire du Solide Institut Le Bel, 4, Rue Blaise Pascal 67070 Strasbourg Cedex, France Tél: 03.68.85.15.00 e-mail:
Résolution d équations non linéaires
 Analyse Numérique Résolution d équations non linéaires Said EL HAJJI et Touria GHEMIRES Université Mohammed V - Agdal. Faculté des Sciences Département de Mathématiques. Laboratoire de Mathématiques, Informatique
Analyse Numérique Résolution d équations non linéaires Said EL HAJJI et Touria GHEMIRES Université Mohammed V - Agdal. Faculté des Sciences Département de Mathématiques. Laboratoire de Mathématiques, Informatique
6. Hachage. Accès aux données d'une table avec un temps constant Utilisation d'une fonction pour le calcul d'adresses
 6. Hachage Accès aux données d'une table avec un temps constant Utilisation d'une fonction pour le calcul d'adresses PLAN Définition Fonctions de Hachage Méthodes de résolution de collisions Estimation
6. Hachage Accès aux données d'une table avec un temps constant Utilisation d'une fonction pour le calcul d'adresses PLAN Définition Fonctions de Hachage Méthodes de résolution de collisions Estimation
Optimisation multi-critère pour l allocation de ressources sur Clouds distribués avec prise en compte de l énergie
 Optimisation multi-critère pour l allocation de ressources sur Clouds distribués avec prise en compte de l énergie 1 Présenté par: Yacine KESSACI Encadrement : N. MELAB E-G. TALBI 31/05/2011 Plan 2 Motivation
Optimisation multi-critère pour l allocation de ressources sur Clouds distribués avec prise en compte de l énergie 1 Présenté par: Yacine KESSACI Encadrement : N. MELAB E-G. TALBI 31/05/2011 Plan 2 Motivation
UEO11 COURS/TD 1. nombres entiers et réels codés en mémoire centrale. Caractères alphabétiques et caractères spéciaux.
 UEO11 COURS/TD 1 Contenu du semestre Cours et TDs sont intégrés L objectif de ce cours équivalent a 6h de cours, 10h de TD et 8h de TP est le suivant : - initiation à l algorithmique - notions de bases
UEO11 COURS/TD 1 Contenu du semestre Cours et TDs sont intégrés L objectif de ce cours équivalent a 6h de cours, 10h de TD et 8h de TP est le suivant : - initiation à l algorithmique - notions de bases
Les OGM. 5 décembre 2008. Nicole Mounier
 Les OGM 5 décembre 2008 Nicole Mounier Université Claude Bernard Lyon 1 CGMC, bâtiment Gregor Mendel 43, boulevard du 11 Novembre 1918 69622 Villeurbanne Cedex OGM Organismes Génétiquement Modifiés Transfert
Les OGM 5 décembre 2008 Nicole Mounier Université Claude Bernard Lyon 1 CGMC, bâtiment Gregor Mendel 43, boulevard du 11 Novembre 1918 69622 Villeurbanne Cedex OGM Organismes Génétiquement Modifiés Transfert
Communications collectives et ordonnancement en régime permanent pour plates-formes hétérogènes
 Loris MARCHAL Laboratoire de l Informatique du Parallélisme Équipe Graal Communications collectives et ordonnancement en régime permanent pour plates-formes hétérogènes Thèse réalisée sous la direction
Loris MARCHAL Laboratoire de l Informatique du Parallélisme Équipe Graal Communications collectives et ordonnancement en régime permanent pour plates-formes hétérogènes Thèse réalisée sous la direction
3A-IIC - Parallélisme & Grid GRID : Définitions. GRID : Définitions. Stéphane Vialle. [email protected] http://www.metz.supelec.
 3A-IIC - Parallélisme & Grid Stéphane Vialle [email protected] http://www.metz.supelec.fr/~vialle Principes et Objectifs Evolution Leçons du passé Composition d une Grille Exemple d utilisation
3A-IIC - Parallélisme & Grid Stéphane Vialle [email protected] http://www.metz.supelec.fr/~vialle Principes et Objectifs Evolution Leçons du passé Composition d une Grille Exemple d utilisation
MASTER SIS PRO : logique et sécurité DÉTECTION D INTRUSIONS. Odile PAPINI, LSIS. Université de Toulon et du Var. papini@univ-tln.
 MASTER SIS PRO : logique et sécurité DÉTECTION D INTRUSIONS Odile PAPINI, LSIS. Université de Toulon et du Var. [email protected] Plan Introduction Généralités sur les systèmes de détection d intrusion
MASTER SIS PRO : logique et sécurité DÉTECTION D INTRUSIONS Odile PAPINI, LSIS. Université de Toulon et du Var. [email protected] Plan Introduction Généralités sur les systèmes de détection d intrusion
INF6304 Interfaces Intelligentes
 INF6304 Interfaces Intelligentes filtres collaboratifs 1/42 INF6304 Interfaces Intelligentes Systèmes de recommandations, Approches filtres collaboratifs Michel C. Desmarais Génie informatique et génie
INF6304 Interfaces Intelligentes filtres collaboratifs 1/42 INF6304 Interfaces Intelligentes Systèmes de recommandations, Approches filtres collaboratifs Michel C. Desmarais Génie informatique et génie
Base de données bibliographiques Pubmed-Medline
 Chapitre 1 ; Domaine 1 ; Documentation ; Champs référentiels 1.1.1, 1.1.2 et 1.1.3 Base de données bibliographiques Pubmed-Medline D r Patrick Deschamps,, 30 mai 2007 PLAN C2i métiers de la santé Introduction
Chapitre 1 ; Domaine 1 ; Documentation ; Champs référentiels 1.1.1, 1.1.2 et 1.1.3 Base de données bibliographiques Pubmed-Medline D r Patrick Deschamps,, 30 mai 2007 PLAN C2i métiers de la santé Introduction
Les algorithmes de fouille de données
 Février 2005 Les algorithmes de fouille de données DATAMINING Techniques appliquées à la vente, aux services client, interdictions. Cycle C Informatique Remerciements Je remercie les personnes, les universités
Février 2005 Les algorithmes de fouille de données DATAMINING Techniques appliquées à la vente, aux services client, interdictions. Cycle C Informatique Remerciements Je remercie les personnes, les universités
Présentation du Master Ingénierie Informatique et du Master Science Informatique 2007-2008, Année 2 Université Paris-Est Marne-la-Vallée
 Présentation du Master Ingénierie Informatique et du Master Science Informatique 2007-2008, Année 2 Université Paris-Est Marne-la-Vallée Responsable du Master Informatique : Marc Zipstein Responsable de
Présentation du Master Ingénierie Informatique et du Master Science Informatique 2007-2008, Année 2 Université Paris-Est Marne-la-Vallée Responsable du Master Informatique : Marc Zipstein Responsable de
Réseau sur. Médicaments. l Innocuité et l Efficacité des. Document d orientation pour la présentation de requêtes au RIEM
 Réseau sur l Innocuité et l Efficacité des Médicaments Document d orientation pour la présentation de requêtes au RIEM Instituts de recherche en santé du Canada Canadian Institutes of Health Research Version
Réseau sur l Innocuité et l Efficacité des Médicaments Document d orientation pour la présentation de requêtes au RIEM Instituts de recherche en santé du Canada Canadian Institutes of Health Research Version
Complexité et auto-organisation chez les insectes sociaux. Complexité et auto-organisation chez les insectes sociaux
 Complexité et auto-organisation chez les insectes sociaux Guy Theraulaz Centre de Recherches sur la Cognition Animale CNRS, UMR 5169, Toulouse, France Marathon des Sciences XXIII ème Festival d Astronomie,
Complexité et auto-organisation chez les insectes sociaux Guy Theraulaz Centre de Recherches sur la Cognition Animale CNRS, UMR 5169, Toulouse, France Marathon des Sciences XXIII ème Festival d Astronomie,
CONSTRUIRE UNE QUESTION ET ELABORER UN QUESTIONNAIRE?
 Déchets : outils et exemples pour agir Fiche méthode n 3 www.optigede.ademe.fr CONSTRUIRE UNE ET ELABORER UN NAIRE? Origine et objectif de la fiche : Les retours d expérience des collectivités locales
Déchets : outils et exemples pour agir Fiche méthode n 3 www.optigede.ademe.fr CONSTRUIRE UNE ET ELABORER UN NAIRE? Origine et objectif de la fiche : Les retours d expérience des collectivités locales
VI- Expression du génome
 VI- Expression du génome VI-1.- EXPRESSION DU GÉNOME- PRINCIPES GÉNÉRAUX DOGME CENTRAL Les gènes et l information génétique sont conservés sous forme d acides nucléiques La perpétuation à l identique de
VI- Expression du génome VI-1.- EXPRESSION DU GÉNOME- PRINCIPES GÉNÉRAUX DOGME CENTRAL Les gènes et l information génétique sont conservés sous forme d acides nucléiques La perpétuation à l identique de
Modèles à Événements Discrets. Réseaux de Petri Stochastiques
 Modèles à Événements Discrets Réseaux de Petri Stochastiques Table des matières 1 Chaînes de Markov Définition formelle Idée générale Discrete Time Markov Chains Continuous Time Markov Chains Propriétés
Modèles à Événements Discrets Réseaux de Petri Stochastiques Table des matières 1 Chaînes de Markov Définition formelle Idée générale Discrete Time Markov Chains Continuous Time Markov Chains Propriétés
LES CARTES À POINTS : POUR UNE MEILLEURE PERCEPTION
 LES CARTES À POINTS : POUR UNE MEILLEURE PERCEPTION DES NOMBRES par Jean-Luc BREGEON professeur formateur à l IUFM d Auvergne LE PROBLÈME DE LA REPRÉSENTATION DES NOMBRES On ne conçoit pas un premier enseignement
LES CARTES À POINTS : POUR UNE MEILLEURE PERCEPTION DES NOMBRES par Jean-Luc BREGEON professeur formateur à l IUFM d Auvergne LE PROBLÈME DE LA REPRÉSENTATION DES NOMBRES On ne conçoit pas un premier enseignement
Actualités sur la sélection des pondeuses Prospections futures. Dr. Matthias Schmutz, Lohmann Tierzucht
 Actualités sur la sélection des pondeuses Prospections futures Dr. Matthias Schmutz, Lohmann Tierzucht Alimentation et démographie mondiale Augmentation annuelle de 80 millions Croissance surtout dans
Actualités sur la sélection des pondeuses Prospections futures Dr. Matthias Schmutz, Lohmann Tierzucht Alimentation et démographie mondiale Augmentation annuelle de 80 millions Croissance surtout dans
Analyse dialectométrique des parlers berbères de Kabylie
 Saïd GUERRAB Analyse dialectométrique des parlers berbères de Kabylie Résumé de la thèse (pour affichage) Il est difficile de parler du berbère sans parler de la variation. Il y a d abord une variation
Saïd GUERRAB Analyse dialectométrique des parlers berbères de Kabylie Résumé de la thèse (pour affichage) Il est difficile de parler du berbère sans parler de la variation. Il y a d abord une variation
Continuité et dérivabilité d une fonction
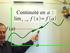 DERNIÈRE IMPRESSIN LE 7 novembre 014 à 10:3 Continuité et dérivabilité d une fonction Table des matières 1 Continuité d une fonction 1.1 Limite finie en un point.......................... 1. Continuité
DERNIÈRE IMPRESSIN LE 7 novembre 014 à 10:3 Continuité et dérivabilité d une fonction Table des matières 1 Continuité d une fonction 1.1 Limite finie en un point.......................... 1. Continuité
Cours 3 : Python, les conditions
 Cours 3 : Python, les conditions Conditions Permet d'exécuter des ordres dans certaines conditions : if condition1: code exécuté si condition1 est vraie code exécuté si condition1 est vraie... elif condition2:
Cours 3 : Python, les conditions Conditions Permet d'exécuter des ordres dans certaines conditions : if condition1: code exécuté si condition1 est vraie code exécuté si condition1 est vraie... elif condition2:
L axe 5 du Cancéropole Nord Ouest
 L axe 5 du Cancéropole Nord Ouest Cancers, Individu id & Société L état des lieux d un pari Le Rapport Cordier 1 Biomarqueurs prédictifs 2 L axe 5 du Cancéropole Nord Ouest Cancers, Individu & Société
L axe 5 du Cancéropole Nord Ouest Cancers, Individu id & Société L état des lieux d un pari Le Rapport Cordier 1 Biomarqueurs prédictifs 2 L axe 5 du Cancéropole Nord Ouest Cancers, Individu & Société
Chapitre 7. Récurrences
 Chapitre 7 Récurrences 333 Plan 1. Introduction 2. Applications 3. Classification des récurrences 4. Résolution de récurrences 5. Résumé et comparaisons Lectures conseillées : I MCS, chapitre 20. I Rosen,
Chapitre 7 Récurrences 333 Plan 1. Introduction 2. Applications 3. Classification des récurrences 4. Résolution de récurrences 5. Résumé et comparaisons Lectures conseillées : I MCS, chapitre 20. I Rosen,
Introduction des. comptes d épargne libre d impôt
 Introduction des comptes d épargne libre d impôt Comptes d épargne libre d impôt Une nouvelle façon d épargner Les comptes d épargne libre d impôt ont été introduits par le gouvernement fédéral dans le
Introduction des comptes d épargne libre d impôt Comptes d épargne libre d impôt Une nouvelle façon d épargner Les comptes d épargne libre d impôt ont été introduits par le gouvernement fédéral dans le
Sauvegarde collaborative entre pairs Ludovic Courtès LAAS-CNRS
 Sauvegarde collaborative entre pairs 1 Sauvegarde collaborative entre pairs Ludovic Courtès LAAS-CNRS Sauvegarde collaborative entre pairs 2 Introduction Pourquoi pair à pair? Utilisation de ressources
Sauvegarde collaborative entre pairs 1 Sauvegarde collaborative entre pairs Ludovic Courtès LAAS-CNRS Sauvegarde collaborative entre pairs 2 Introduction Pourquoi pair à pair? Utilisation de ressources
Génomique Comparative et intégrative
 Génomique Comparative et intégrative Introduction : Le big data : on peut traiter des données massives à présent, l'objectif à présent est d'éviter les transferts de données trop longs. On a tout à portée
Génomique Comparative et intégrative Introduction : Le big data : on peut traiter des données massives à présent, l'objectif à présent est d'éviter les transferts de données trop longs. On a tout à portée
Cours (7) de statistiques à distance, élaboré par Zarrouk Fayçal, ISSEP Ksar-Said, 2011-2012 LES STATISTIQUES INFERENTIELLES
 LES STATISTIQUES INFERENTIELLES (test de Student) L inférence statistique est la partie des statistiques qui, contrairement à la statistique descriptive, ne se contente pas de décrire des observations,
LES STATISTIQUES INFERENTIELLES (test de Student) L inférence statistique est la partie des statistiques qui, contrairement à la statistique descriptive, ne se contente pas de décrire des observations,
Christophe CANDILLIER Cours de DataMining mars 2004 Page 1
 Christophe CANDILLIER Cours de DataMining mars 2004 age 1 1. Introduction 2. rocessus du DataMining 3. Analyse des données en DataMining 4. Analyse en Ligne OLA 5. Logiciels 6. Bibliographie Christophe
Christophe CANDILLIER Cours de DataMining mars 2004 age 1 1. Introduction 2. rocessus du DataMining 3. Analyse des données en DataMining 4. Analyse en Ligne OLA 5. Logiciels 6. Bibliographie Christophe
Les débuts de la génétique
 HPITRE 9 DES DÉBTS DE L ÉNÉTIQE X ENJEX TELS DES BIOTEHNOLOIES 1 Les débuts de la génétique est avec les travaux de regor Mendel vers la fin du XIX e siècle que furent posées les bases de la génétique.
HPITRE 9 DES DÉBTS DE L ÉNÉTIQE X ENJEX TELS DES BIOTEHNOLOIES 1 Les débuts de la génétique est avec les travaux de regor Mendel vers la fin du XIX e siècle que furent posées les bases de la génétique.
Semestre 2 Spécialité «Analyse in silico des complexes macromolécules biologiques-médicaments»
 Master In silico Drug Design Semestre 2 Spécialité «Analyse in silico des complexes macromolécules biologiques-médicaments» 30NU01IS INITIATION A LA PROGRAMMATION (6 ECTS) Responsables : D. MESTIVIER,
Master In silico Drug Design Semestre 2 Spécialité «Analyse in silico des complexes macromolécules biologiques-médicaments» 30NU01IS INITIATION A LA PROGRAMMATION (6 ECTS) Responsables : D. MESTIVIER,
Big Data et Graphes : Quelques pistes de recherche
 Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de Lyon/Université Claude Bernard Lyon 1/Université
Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de Lyon/Université Claude Bernard Lyon 1/Université
«Cours Statistique et logiciel R»
 «Cours Statistique et logiciel R» Rémy Drouilhet (1), Adeline Leclercq-Samson (1), Frédérique Letué (1), Laurence Viry (2) (1) Laboratoire Jean Kuntzmann, Dép. Probabilites et Statistique, (2) Laboratoire
«Cours Statistique et logiciel R» Rémy Drouilhet (1), Adeline Leclercq-Samson (1), Frédérique Letué (1), Laurence Viry (2) (1) Laboratoire Jean Kuntzmann, Dép. Probabilites et Statistique, (2) Laboratoire
IMMUNOLOGIE. La spécificité des immunoglobulines et des récepteurs T. Informations scientifiques
 IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
WEBINAIRE SUR LE SUIVI DE TENDANCES
 WEBINAIRE SUR LE SUIVI DE TENDANCES Le 16/02/2012 à 21H Présenté par Gilles SANTACREU (Boursikoter.com) En partenariat avec CMC Markets 1 Gilles SANTACREU, 43 ans - Webmaster et fondateur du site Boursikoter.com
WEBINAIRE SUR LE SUIVI DE TENDANCES Le 16/02/2012 à 21H Présenté par Gilles SANTACREU (Boursikoter.com) En partenariat avec CMC Markets 1 Gilles SANTACREU, 43 ans - Webmaster et fondateur du site Boursikoter.com
Mensuration officielle Plan de conservation et d archivage de données et de documents (PCA)
 Directive du 23 juin 2014 (état au 29 janvier 2015) Mensuration officielle Plan de conservation et d archivage de données et de documents (PCA) Editeur Groupe de travail «Archivage de données de la MO»
Directive du 23 juin 2014 (état au 29 janvier 2015) Mensuration officielle Plan de conservation et d archivage de données et de documents (PCA) Editeur Groupe de travail «Archivage de données de la MO»
Gestion de projets et de portefeuilles pour l entreprise innovante
 LIVRE BLANC Novembre 2010 Gestion de projets et de portefeuilles pour l entreprise innovante accélérer le taux de rendement de l innovation James Ramsay Consultant principal, Gouvernance de la zone Europe,
LIVRE BLANC Novembre 2010 Gestion de projets et de portefeuilles pour l entreprise innovante accélérer le taux de rendement de l innovation James Ramsay Consultant principal, Gouvernance de la zone Europe,
SysFera. Benjamin Depardon
 SysFera Passage d applications en SaaS Benjamin Depardon CTO@SysFera SysFera Technologie 2001 Création 2010 Spin Off INRIA Direction par un consortium d investisseurs 12 personnes 75% en R&D Implantation
SysFera Passage d applications en SaaS Benjamin Depardon CTO@SysFera SysFera Technologie 2001 Création 2010 Spin Off INRIA Direction par un consortium d investisseurs 12 personnes 75% en R&D Implantation
Modélisation du comportement habituel de la personne en smarthome
 Modélisation du comportement habituel de la personne en smarthome Arnaud Paris, Selma Arbaoui, Nathalie Cislo, Adnen El-Amraoui, Nacim Ramdani Université d Orléans, INSA-CVL, Laboratoire PRISME 26 mai
Modélisation du comportement habituel de la personne en smarthome Arnaud Paris, Selma Arbaoui, Nathalie Cislo, Adnen El-Amraoui, Nacim Ramdani Université d Orléans, INSA-CVL, Laboratoire PRISME 26 mai
Asynchronisme : Cadres continu et discret
 N d ordre : 151 Année 2006 HABILITATION À DIRIGER DES RECHERCHES UNIVERSITÉ DE FRANCHE-COMTÉ Spécialité Informatique présentée par Sylvain CONTASSOT-VIVIER Docteur en Informatique Sujet Asynchronisme :
N d ordre : 151 Année 2006 HABILITATION À DIRIGER DES RECHERCHES UNIVERSITÉ DE FRANCHE-COMTÉ Spécialité Informatique présentée par Sylvain CONTASSOT-VIVIER Docteur en Informatique Sujet Asynchronisme :
Souad EL Bernoussi. Groupe d Analyse Numérique et Optimisation Rabat http ://www.fsr.ac.ma/ano/
 Recherche opérationnelle Les démonstrations et les exemples seront traités en cours Souad EL Bernoussi Groupe d Analyse Numérique et Optimisation Rabat http ://www.fsr.ac.ma/ano/ Table des matières 1 Programmation
Recherche opérationnelle Les démonstrations et les exemples seront traités en cours Souad EL Bernoussi Groupe d Analyse Numérique et Optimisation Rabat http ://www.fsr.ac.ma/ano/ Table des matières 1 Programmation
DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION
 DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION PRINCIPES DE BASE SUR LES DONNEES ET LE CALCUL HAUTE PERFORMANCE Lois de Gray sur l ingénierie des données 1 : Les calculs scientifiques traitent des volumes considérables
DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION PRINCIPES DE BASE SUR LES DONNEES ET LE CALCUL HAUTE PERFORMANCE Lois de Gray sur l ingénierie des données 1 : Les calculs scientifiques traitent des volumes considérables
Univers Vivant Révision. Notions STE
 Univers Vivant Révision Notions STE Chap. 13) L Écologie 1) a) Qu est-ce que l empreinte écologique? L empreinte écologique correspond à la surface terrestre et aquatique totale nécessaire à un individu,
Univers Vivant Révision Notions STE Chap. 13) L Écologie 1) a) Qu est-ce que l empreinte écologique? L empreinte écologique correspond à la surface terrestre et aquatique totale nécessaire à un individu,
Big Data et Graphes : Quelques pistes de recherche
 Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci http://liris.cnrs.fr/hamamache.kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de
Big Data et Graphes : Quelques pistes de recherche Hamamache Kheddouci http://liris.cnrs.fr/hamamache.kheddouci Laboratoire d'informatique en Image et Systèmes d'information LIRIS UMR 5205 CNRS/INSA de
Programmation linéaire
 Programmation linéaire DIDIER MAQUIN Ecole Nationale Supérieure d Electricité et de Mécanique Institut National Polytechnique de Lorraine Mathématiques discrètes cours de 2ème année Programmation linéaire
Programmation linéaire DIDIER MAQUIN Ecole Nationale Supérieure d Electricité et de Mécanique Institut National Polytechnique de Lorraine Mathématiques discrètes cours de 2ème année Programmation linéaire
Evaluation des performances de programmes parallèles haut niveau à base de squelettes
 Evaluation des performances de programmes parallèles haut niveau à base de squelettes Enhancing the Performance Predictability of Grid Applications with Patterns and Process Algebras A. Benoit, M. Cole,
Evaluation des performances de programmes parallèles haut niveau à base de squelettes Enhancing the Performance Predictability of Grid Applications with Patterns and Process Algebras A. Benoit, M. Cole,
Limitations of the Playstation 3 for High Performance Cluster Computing
 Introduction Plan Limitations of the Playstation 3 for High Performance Cluster Computing July 2007 Introduction Plan Introduction Intérêts de la PS3 : rapide et puissante bon marché L utiliser pour faire
Introduction Plan Limitations of the Playstation 3 for High Performance Cluster Computing July 2007 Introduction Plan Introduction Intérêts de la PS3 : rapide et puissante bon marché L utiliser pour faire
Cours des Méthodes de Résolution Exactes Heuristiques et Métaheuristiques
 Université Mohammed V, Faculté des Sciences de Rabat Laboratoire de Recherche Mathématiques, Informatique et Applications Cours des Méthodes de Résolution Exactes Heuristiques et Métaheuristiques MASTER
Université Mohammed V, Faculté des Sciences de Rabat Laboratoire de Recherche Mathématiques, Informatique et Applications Cours des Méthodes de Résolution Exactes Heuristiques et Métaheuristiques MASTER
Classer le vivant. Module de formation pour les professeurs des écoles
 Classer le vivant Module de formation pour les professeurs des écoles L'objectif de cette formation est de permettre aux stagiaires de construire les connaissances et les méthodes permettant de classer
Classer le vivant Module de formation pour les professeurs des écoles L'objectif de cette formation est de permettre aux stagiaires de construire les connaissances et les méthodes permettant de classer
Plus courts chemins, programmation dynamique
 1 Plus courts chemins, programmation dynamique 1. Plus courts chemins à partir d un sommet 2. Plus courts chemins entre tous les sommets 3. Semi-anneau 4. Programmation dynamique 5. Applications à la bio-informatique
1 Plus courts chemins, programmation dynamique 1. Plus courts chemins à partir d un sommet 2. Plus courts chemins entre tous les sommets 3. Semi-anneau 4. Programmation dynamique 5. Applications à la bio-informatique
Arbres binaires de décision
 1 Arbres binaires de décision Résumé Arbres binaires de décision Méthodes de construction d arbres binaires de décision, modélisant une discrimination (classification trees) ou une régression (regression
1 Arbres binaires de décision Résumé Arbres binaires de décision Méthodes de construction d arbres binaires de décision, modélisant une discrimination (classification trees) ou une régression (regression
Algorithmes de recherche
 Algorithmes de recherche 1 Résolution de problèmes par recherche On représente un problème par un espace d'états (arbre/graphe). Chaque état est une conguration possible du problème. Résoudre le problème
Algorithmes de recherche 1 Résolution de problèmes par recherche On représente un problème par un espace d'états (arbre/graphe). Chaque état est une conguration possible du problème. Résoudre le problème
Evaluation du LIDAR et de solutions innovantes pour la chaîne d approvisionnement du bois : les résultats du projet européen FlexWood
 Evaluation du LIDAR et de solutions innovantes pour la chaîne d approvisionnement du bois : les résultats du projet européen FlexWood Le projet de recherche européen FlexWood («Flexible wood supply chain»)
Evaluation du LIDAR et de solutions innovantes pour la chaîne d approvisionnement du bois : les résultats du projet européen FlexWood Le projet de recherche européen FlexWood («Flexible wood supply chain»)
