Introduction : l ère de la génomique
|
|
|
- Lucile Lecompte
- il y a 10 ans
- Total affichages :
Transcription
1 Introduction : l ère de la génomique En 1995, pour la première fois, la séquence complète du génome d une cellule vivante a été déterminée. Il s agissait d Haemophilus influenzae, une bactérie responsable d infections bronco-pulmonaires chez les jeunes enfants, dont le génome est composé d un seul chromosome circulaire long de paires de bases et comportant 1738 gènes. Au cours des 5 dernières années, les progrès ont été tout à fait spectaculaires : aujourd hui (printemps 2002), les séquences d une centaine de génomes complets sont connues, provenant de domaines très différents du Vivant : bactéries, archaebactéries, champignons, invertébrés, insectes, plantes. Cet effort a trouvé son point culminant au début de l année 2001 avec la publication de la séquence «brute» du génome humain. À l heure actuelle, cet effort se poursuit encore avec un grand nombre d autres génomes en cours d étude : bactéries, souris, poissons, plantes cultivées... La biologie moléculaire est donc entrée depuis 1995 dans l ère de la génomique : on dispose maintenant de l information génétique exhaustive sur un nombre croissant d organismes vivants et il est aujourd hui possible d aborder de manière globale un certain nombre de problèmes complexes dont on n avait jusqu à présent qu une connaissance fragmentaire : voies métaboliques, interaction de la cellule avec l extérieur, mécanismes globaux de régulation et de contrôle. Une nouvelle discipline est également née de la connaissance de ces séquences complètes de chromosomes : la génomique comparée. Il est maintenant possible de comparer deux organismes vivants à l échelle de leur génome, de déterminer les gènes qu ils ont en commun ou qui leur sont propres. Ce type d analyse est très prometteur dans le contexte de l identification sélective de gènes correspondant à des cibles thérapeutiques : en comparant par exemple une bactérie pathogène et une proche cousine non-pathogène, on peut essayer 9
2 10 Introduction de repérer les gènes impliqués dans la virulence de la souche infectieuse. L accélération du séquençage, permise en particulier par la robotisation et la parallélisation des méthodes d analyse, nécessite un soutien de plus en plus important de l outil informatique. Dans un premier stade, celui-ci est indispensable pour permettre l assemblage du gigantesque «puzzle» que constituent les milliers ou millions de fragments de génome issus des automates de séquençage. Ensuite l informatique est un outil incontournable pour extraire et analyser l information contenue dans ces gigabases (1 Gbase = 10 9 nucléotides) de séquence. Le volume des données à traiter est considérable, aujourd hui (printemps 2002) les banques de séquences rassemblent plus de nucléotides et leur taille augmente exponentiellement avec un temps de doublement de l ordre de 15 à 18 mois. Il est clairement impossible de caractériser expérimentalement tous les gènes contenus dans ces séquences et c est pourquoi l analyse in silicio (grâce au silicium des microprocesseurs) doit venir au secours des biologistes pour compléter et guider les approches in vitro et in vivo. La bioinformatique, discipline récente, traite des différents aspects de ce nouveau champ de la connaissance et s appuie bien sûr à la fois sur les concepts de la biologie et de l informatique, et sur des outils issus de la chimie et de la physique.
3 Chapitre 1 Séquençage et Génome 1.1 Le séquençage automatique La méthode des didésoxyribonucléotides, inventée il y a une vingtaine d année dans le laboratoire de Fred Sanger à Cambridge en Grande-Bretagne, est aujourd hui universellement employée pour séquencer l ADN. Elle repose sur l allongement par l ADN polymérase d un brin à partir d une amorce, en utilisant un autre brin d ADN comme matrice. Cet allongement est réalisé en présence des quatre désoxyribonucléotides triphosphate (datp, dttp, dgtp, dctp), monomères utilisés par la polymérase, et d un analogue didésoxyribonucléotide (ddntp, cf figure 1) qui joue le rôle de terminateur de chaîne. Du fait de l incorporation spécifique de l analogue par la polymérase, on obtient un mélange de fragments qui se terminent sélectivement aux positions correspondant au nucléotide choisi (dans l exemple ci-dessous, les A). Le principe de la méthode de séquençage est illustré sur la figure 2 : On effectue ainsi quatre réactions en parallèle, chacune avec l un des quatre ddntp, et l on sépare les fragments obtenus par électrophorèse. Afin de pouvoir identifier les fragments d ADN synthétisés par la polymérase et en particulier pour pouvoir les distinguer de l ADN matrice, on les marque avec un traceur fluorescent. Celui-ci est accroché à l une de ses deux extrémités, soit en 5, sur l amorce de séquençage, soit en 3 sur le didésoxyribonucléotide terminateur. Les séquenceurs automatiques modernes utilisent un système de détection in situ pendant l électrophorèse. Le faisceau d un laser émettant dans la bande d absorption du fluorophore traverse le gel (cf. figure 3). Pendant la migration, 11
4 12 Chapitre 1. Séquençage et Génome Dans le didésoxyribonucléotide (ddntp), le remplacement du groupement 3 -OH par un 3 -H empêche la formation d une liaison phosphodiester du côté 3. Ces nucléotides modifiés peuvent toutefois être incorporés par l ADN polymérase car ils possèdent un côté 5 -triphosphate normal. Les règles d appariement A-T et G-C sont respectées lors de l incorporation des ddntp. Ainsi le ddatp sera incorporé lorsqu on trouvera en regard un T sur le brin matrice. Fig. 1.1 : Structure des didésoxyribonucléotides. lorsqu une bande d ADN passe devant le faisceau, un signal de fluorescence est émis. Celui-ci est capté par une photodiode située en regard du gel. Le signal est amplifié puis transmis à l ordinateur de contrôle et analysé par un logiciel spécialisé. Dans des conditions favorables, cette technique permet de lire jusqu à 1000 nucléotides par fragment séquencé. En routine, la moyenne est de l ordre 500 à 800 nucléotides par expérience. Deux méthodologies cohabitent actuellement, reposant sur l utilisation d un fluorophore unique ou bien sur celle de quatre traceurs fluorescents possédant des spectres d émission distincts. Dans le premier cas, les quatre mélanges correspondant à chacun des quatre ddntp sont déposés sur des puits distincts du gel. L analyse se fait sur la migration comparée des fragments dans les quatre pistes résultantes. Dans l autre système, on utilise un fluorophore différent dans chacune des quatre réactions de séquençage. Une solution consiste
5 Le séquençage automatique 13 Fig. 1.2 : Principe du séquençage par la méthode de Sanger. à utiliser des ddntp modifiés chacun par un traceur spécifique. Après avoir effectué les quatre réactions de polymérisation, on les mélange et on dépose dans le même puit sur le gel. La reconnaissance des nucléotides se fait alors sur la base des propriétés d émission du traceur qui passe devant le faisceau laser, au moyen de filtres colorés sélectifs. L analyse est alors effectuée sur une seule piste du gel. La technique à quatre fluorophores est un peu plus onéreuse, car elle nécessite une chimie un peu plus diversifiée. En revanche, elle présente l avantage d être bien adaptée aux plus hauts débits, car plus d échantillons peuvent être analysés sur le même gel. Dans les séquenceurs de dernière génération, le gel de polyacrylamide rectangulaire classique est remplacé par un capillaire réutilisable (le principe de la séparation et de la détection reste le même). Cette technique permet de réduire la durée des expériences de quelques heures
6 14 Chapitre 1. Séquençage et Génome Fig. 1.3 : Séquençage automatique sur des séquenceurs 1 et 4 fluorophores. Les échantillons déposés dans les puits en haut sont séparés par une électrophorèse dans un gel de polyacrylamide-urée. La séquence 5 -CAATCCCGGATGTTT se lit de bas en haut. à quelques dizaines de minutes et de minimiser le temps passé par l opérateur pour sa préparation. Les modèles multi-capillaires les plus performants peuvent, en principe, traiter jusqu à 1000 échantillons par jour, soit un débit théorique de 0,5 Mbases de séquence brute par jour et par machine. Les centres de séquençage massif possèdent aujourd hui plusieurs dizaines de ces machines (figure 4). Les réactions de séquençage peuvent également être réalisées par des robots qui réalisent automatiquement pipetages, mélanges et incubations et minimisent les risques d erreur humaine. La préparation des matrices d ADN reste l étape la plus lourde à automatiser, même si de nombreux progrès ont été accomplis. 1.2 Stratégies de séquençage La méthodologie de séquençage décrite ci-dessus occulte deux difficultés importantes que l on doit prendre en compte lorsqu on attaque un programme de séquençage de grande envergure : On ne peut séquencer que des morceaux d environ 500 à 1000 nucléotides de long.
7 Stratégies de séquençage 15 Fig. 1.4: Séquenceur multicapillaire haut de gamme permettant le séquençage simultané de 96 échantillons. Un système d injection automatique permet d effectuer plusieurs séparations consécutives sans intervention humaine (capplied Biosystems). C T T 280 T G T A T G C G C C 290 A T T G T A G C A C 300 G T G T G T A G C C 310 C T G G T C G T A A 320 G G G C C A T G A T 330 G A C Fig. 1.5 : Exemple de profil de séquençage. L intensité du signal détecté par les photodiodes est tracée en fonction du temps de séparation. Chaque couleur est associée à l une des quatre réactions (A, G, C ou T).
8 16 Chapitre 1. Séquençage et Génome Il faut une amorce de séquençage complémentaire à la matrice pour que l ADN polymérase puisse démarrer la synthèse. Ces deux obstacles peuvent heureusement être simultanément surmontés en fragmentant l ADN à séquencer en morceaux de taille compatible avec le séquençage ( 10 3 paires de bases) et en insérant ensuite ceux-ci dans un vecteur donné (plasmide, virus...). Cet ADN vecteur est choisi suivant plusieurs critères : Il est capable de se répliquer de manière autonome dans une cellule hôte facile à manipuler (en général E. coli). Il porte un ou plusieurs gènes marqueurs permettant de sélectionner les cellules qui le contiennent (par exemple : résistance à un antibiotique). Sa séquence nucléotidique est connue. Il contient des sites pour des endonucléases de restriction pour permettre le clonage (insertion) de fragments d ADN étrangers. Dans la pratique, on utilise en général des petits plasmides bactériens. Après avoir fragmenté et ligaturé l ADN à séquencer dans le vecteur choisi, on peut propager celui-ci dans les cellules hôtes. On isole alors des lignées de cellules clonales (issues d une seule cellule initiale, par divisions successives) contenant chacune un type de vecteur recombinant avec un même fragment d ADN inséré. En collectant un grand nombre de ces lignées clonales, on constitue donc une bibliothèque des morceaux de l ADN à étudier (figure 7). Pour déterminer la séquence de l ADN de l un de ces morceaux, on cultive la lignée de cellules correspondante, on en extrait l ADN qui peut alors être séquencé par la méthode des didésoxyribonucléotides. Pour les amorces, on profite de ce que la séquence du vecteur est connue pour utiliser des amorces oligonucléotidiques situées de part et d autre du site de clonage (cf figure 6). Ces amorces sont indépendantes de la séquence insérée dans le vecteur et peuvent être utilisées pour tous les fragments à séquencer. On parle d amorces universelles. Ces amorces étant constantes, il est très simple d y incorporer les traceurs fluorescents désirés lors de la synthèse chimique de l oligonucléotide. On peut alors utiliser ces amorces fluorescentes pour la très grande majorité des séquençages réalisés.
9 Stratégies de fragmentation 17 Fig. 1.6 : Séquençage dans un vecteur à partir d amorces universelles. 1.3 Stratégies de fragmentation Lorsqu on entame le séquençage d un ADN de taille importante, et afortiori celui d un génome complet, nous venons de voir qu il est indispensable de découper celui-ci en fragments de taille compatible avec le séquençage. Ceci pose à nouveau deux questions : Quelle stratégie de découpage employer? Comment reconstituer la séquence complète à partir des morceaux? Ces deux questions sont intimement liées, car la méthode de reconstruction va dépendre précisément de comment la fragmentation a été opérée. Deux approches différentes ont principalement été utilisées : la fragmentation aléatoire et la segmentation après cartographie. Fragmentation aléatoire Dans la fragmentation aléatoire, on découpe directement l ensemble de l ADN à séquencer en petits morceaux de taille optimisée pour le séquençage ( 1000 paires de bases). Pour cela, on peut soit utiliser une enzyme de restriction à haute fréquence de coupure (1 site tous les pb), en effectuant une digestion très partielle (10 ou 20%) pour générer au hasard des fragments de 1000 à 2000 pb, soit effectuer un cassage par ultrasons. Les contraintes mécaniques induites par les vibrations ultrasonores dans une solution d ADN sont en effet suffisantes pour induire des ruptures de la longue chaîne phosphodiester. La méthode mécanique est plus aléatoire que la méthode enzymatique, mais nécessite une étape supplémentaire de réparation des extrémités d ADN produites. En effet, lors du traitement, la cassure ne se produit en général pas exactement au même niveau sur les deux brins d ADN. Il faut alors rogner les
10 18 Chapitre 1. Séquençage et Génome Fig. 1.7 : Stratégie de construction d une banque d ADN. extrémités simple brins qui dépassent, afin de pouvoir insérer les fragments dans un site de clonage à bords francs, choisi sur le vecteur de séquençage. Le postulat de base de la méthode aléatoire, dite «shotgun» (fusil à canon scié) est que si on analyse suffisamment de clones, on finira par couvrir toute la séquence de l ADN de départ. Si on fait l approximation que la fragmentation
11 Stratégies de fragmentation 19 et le clonage sont des processus réellement aléatoires et que la taille de l ADN séquencé est suffisamment grande devant celle de chacun des clones individuels, ce qui est en général le cas pour un génome complet, alors la probabilité qu un nucléotide de l ADN étudié ne soit pas couvert par le séquençage aléatoire suit une loi de type poissonnien : p 0 = e N/L où N est le nombre total de nucléotides séquencés sur l ensemble des clones et L la longueur totale de l ADN étudié. On appelle N/L le taux de couverture, c est la mesure du taux de redondance des données. Pour obtenir un taux de séquençage de 99%, c est-à-dire p 0 =0, 01, il faut donc séquencer un nombre de clones correspondant à l équivalent de 4,6 fois (log 0, 01 4, 6) la longueur de l ADN étudié. Dans le cas d un génome ou d un très grand ADN, il est donc pratiquement inévitable qu il reste des trous dans la séquence, qu il faudra combler par une autre approche que la méthode aléatoire «shotgun». On peut également évaluer statistiquement la longueur et le nombre moyen de ces «trous» : Longueur totale des trous = Le N/L Longueur moyenne de chaque trou = L n/n Nombredetrous=N/n e N/L où n est la longueur moyenne de chaque fragment séquencé ( 500 nucléotides). Voici à titre d exemple ce que cela donne pour un génome bactérien (L 10 6 pb) et un génome d organisme supérieur (mammifère, plante... ; L 10 9 pb) avec un taux de couverture d un facteur 6 (une valeur moyenne pour ce type de projet), ce qui donne 99,75% de nucléotides séquencés : Bactérie (1 Mpb) Mammifère (1 Gpb) Nombre de séquences Nombre de trous restants Taille moyenne des trous Table 1 La stratégie aléatoire pose deux problèmes principaux : Il est impossible de couvrir la totalité d un génome par cette méthode sans augmenter de manière très sensible le nombre de clones à séquencer : pour couvrir avec quasi-certitude notre génome bactérien, il faudrait que p , soit un taux de couverture d au moins 14 fois. Dans la pratique, il est plus économique de se contenter d un taux de couverture entre 4 et 6 et de boucher ensuite les quelques dizaines de trous restants par des méthodes ad hoc (cf plus bas).
12 20 Chapitre 1. Séquençage et Génome L assemblage du puzzle que constitue l ensemble des fragments peut nécessiter la comparaison systématique 2 à 2 de toutes les séquences obtenues. Avec k séquences, cela représente k (k-1)/2 comparaisons, soit environ 10 8 pour un génome bactérien, et pour un génome de mammifère. Si la première valeur est à la portée des ordinateurs actuels, la seconde représente encore un défi informatique assez formidable. Segmentation après cartographie Dans le cas des génomes de très grande taille, la complexité de reconstruction devient une difficulté sérieuse, c est pourquoi dans ces cas-là, certaines équipes ont eu recours à une approche à deux niveaux. On reprend le principe de la méthode «shotgun», mais pour réduire le nombre de clones nécessaires pour couvrir le génome, on réalise une banque d ADN dans des vecteurs acceptant des fragments de beaucoup plus grande taille. Il en existe trois types principaux : Les cosmides, hybrides entre plasmides bactériens et bactériophages, ils se propagent dans E. coli et acceptent jusqu à kpb d ADN inséré. Les BACs (bacterial artificial chromosome) sont des plasmides de très grande taille, construits à partir de l origine de réplication du chromosome bactérien. Ils peuvent accueillir des fragments insérés de longueur kpb. Les YACs (yeast artificial chromosome) sont des analogues des précédents, mais dérivés du chromosome d une cellule d eucaryote inférieur, la levure de bière. Ces vecteurs acceptent des fragments de l ordre de 1000 kpb. Contrairement aux deux précédents, qui utilisent E. coli comme hôte, les YACs doivent être maintenus dans des cellules de levure. À partir d un génome donné, ces vecteurs permettent de réaliser des banques d ADN pratiquement exhaustives, en utilisant un nombre de clones beaucoup plus raisonnable que les plasmides destinés au séquençage. Ainsi, le Généthon, à Evry, et le CEPH (Centre d étude du polymorphisme humain) ont réalisé une banque du génome humain composée de YACs avec des fragments insérés dont la longueur moyenne était de l ordre de 1 Mpb. Le taux de couverture de cette banque est donc de pratiquement 10 fois (le génome humain comporte environ 3, pb). Ces banques de clones de grande taille ne sont pas directement exploitables pour la séquence. L idée est donc de construire une carte positionnant les YACs, BACs ou cosmides les uns par rapports aux autres, sur les chro-
13 Stratégies de fragmentation 21 mosomes composant le génome étudié. Un tel ordonnancement des YACs du Généthon/CEPH a été réalisé en Cette carte présente deux avantages : 1. Combinée avec une carte génétique, elle permet d isoler des gènes associés à des maladies génétiques, en les localisant sur un YAC donné. Ceci constituait d ailleurs l objectif principal du Généthon dans ce projet. 2. Elle peut servir de cadre pour un programme de séquençage. Une fois les YACs positionnés, il suffit de les séquencer de manière ordonnée, en utilisant la stratégie aléatoire pour chacun d eux. La complexité de la reconstruction est alors analogue à celle d un génome bactérien, puisque leur taille est de l ordre de la Méga paire de bases. Étant donné le taux de couverture (10 x) de la banque de YACs, il «suffit» d en séquencer 3000 à 4000 pour couvrir l ensemble du génome humain. Cette approche est une transposition directe de la stratégie «diviser pour régner» (divide and conquer), classique en informatique. Fig. 1.8 : Stratégie de séquençage en deux étapes avec cartographie intermédiaire. L étape de cartographie ou d ordonnancement de la banque primaire (cosmides, BAC ou YAC) est assez lourde. Elle s effectue par une combinaison de techniques, comme la comparaison des profils de digestion par des enzymes de restriction, l hybridation ADN-ADN ou l identification de marqueurs génétiques. Ainsi, par exemple, deux YACs qui se chevauchent auront des profils de restriction qui contiendront des fragments communs. La recherche systéma-
14 22 Chapitre 1. Séquençage et Génome tique des similitudes entre profils sur l ensemble de la banque de YAC permet d identifier des candidats. Fig. 1.9: Exemple de comparaison de profils de digestion par une enzyme de restriction de deux clones YAC chevauchants. On peut ensuite vérifier le chevauchement de deux YAC par hybridation ADN-ADN. Après séparation des fragments de restriction de l ADN du YAC 1 par électrophorèse, on transfère ceux-ci du gel sur une membrane, où on les immobilise de manière covalente. On sépare alors les deux brins d ADN par un traitement dénaturant (ph alcalin), et l on incube la membrane en présence de l ADN dénaturé du YAC 2, marqué par un traceur (radioactif, fluorescent...). Si les deux YACs ont effectivement des séquences communes, l ADN marqué du YAC 2 se réapparie localement avec celui du YAC 1 (on parle d hybridation entre les deux YAC). On observe alors la fixation du traceur sur la membrane et donc la révélation des fragments d ADN communs aux deux YACs. Dans le cas où la coïncidence des profils de coupure par les enzymes de restriction serait fortuite et ne correspondrait pas à un chevauchement, il n y a pas d hybridation et donc pas de fixation du traceur. Cette analyse, conduite de manière systématique permet de reconstituer une carte du génome. 1.4 Assemblagedeséquence La méthode d assemblage des fragments séquencés requiert dans un premier temps l identification des chevauchements possibles. Il s agit de repérer les clones partageant des séquences communes. Si deux clones se recouvrent, on les réunit pour former ce que l on appelle un contig, termequiestdevenu consacré pour définir un ensemble de fragments qui sont reliés entre eux
15 Assemblage de séquence 23 par des recouvrements de séquences identiques (ou très similaires, aux erreurs de séquençage près). Les phases ultérieures consistent, pas à pas, à comparer chaque nouveau fragment avec les contigs déjà définis (cf figure 10). Ces comparaisons doivent tenir compte des deux orientations relatives possibles des deux séquences. Si le fragment chevauche un contig, on complète celui-ci, sinon on crée un nouveau contig composé de ce seul fragment. Fig : Assemblage itératif des contigs. Les flèches indiquent l orientation des brins d ADN séquencés. Lorsqu un fragment chevauche simultanément deux contigs, on fusionne ces deux contigs et le fragment. À tout moment du développement d un projet de séquençage important, les données comprennent un ensemble de plusieurs contigs. Idéalement, à la fin du projet, il n en reste plus qu un seul, couvrant toute la séquence. En même temps qu on assemble un contig, on définit ce que l on appelle une séquence consensus associée, résultant de l alignement des divers fragments
16 24 Chapitre 1. Séquençage et Génome assemblés au sein du contig. Cette séquence consensus recense les positions où toutes les séquences lues sont en accord et celles pour lesquelles il existe des divergences ou des ambiguïtés associées aux erreurs d interprétation des données (nucléotides non-lus, ou mal interprétés). Ces ambiguïtés devront ensuite être résolues par une analyse plus approfondie des données, voire même par un complément de séquençage. Cette étape de contrôle est indispensable mais longue, car elle est au moins partiellement manuelle. Recherche des chevauchements Lerepéragedeschevauchementspeutsefaireenalignant2à2chaque nouvelle séquence ajoutée à chaque contig déjà assemblé, au moyen des algorithmes décrits dans le chapitre Comparaisons de séquences. Si le score d alignement est supérieur à un seuil donné, les deux séquences sont considérées comme chevauchantes. Cette méthode par «force brute» est toutefois assez coûteuse en temps de calcul, car les algorithmes d alignement sont O(n.m), où n et m sont les longueurs des 2 séquences comparées. S il y a k fragments à assembler, cette méthode est O(k 2 ). Ceci est tout à fait prohibitif dans le cas d un très grand génome où k> Pour simplifier le problème, on peut remarquer que les séquences chevauchantes sont en général identiques ou presque (à quelques rares erreurs près) sur toute la région commune. À partir de cette observation, une stratégie plus efficace a donc été proposée initialement par Roger Staden à Cambridge en 1982 et perfectionnée depuis. Elle consiste à créer une table des 4 n n-uplets de nucléotides possibles (n étant de l ordre de 6 à 12). Pour chaque entrée de la table, on compile la liste des fragments qui contiennent le n-uplet correspondant. La construction de cette table se fait en temps linéaire O(k). Si deux fragments se chevauchent, ils auront un grand nombre de n-uplets communs (tous ceux qui correspondent à la région commune). À partir de ce critère, on peut donc identifier les candidats au chevauchement en regardant simplement dans la table les fragments possédant plusieurs n-uplets communs. Il est alors possible de vérifier le recouvrement en appliquant une méthode d alignement classique. La différence avec la méthode par «force brute» est que le recours à l algorithme d alignement n est fait que dans les cas de recouvrement très probable. Le coût de cette méthode est donc approximativement linéaire en fonction du nombre de gels à analyser. La stratégie heuristique adoptée par Staden peut cependant être mise en défaut dans plusieurs cas : soit parce que la qualité des données de séquence est insuffisante, ce qui peut faire échouer la stratégie de repérage des chevauchements, soit parce que la séquence analysée contient plusieurs répétitions d un motif donné, ce qui peut introduire des erreurs de raccordement dans la
17 Comblement des «trous» 25 construction des contigs, au niveau de ces répétitions. Plusieurs autres méthodes évitent cet écueil en analysant l ensemble des recouvrements possibles, avec des critères permettant d évaluer la qualité des chevauchements (scores d alignement). On construit alors un graphe de l ensemble des connectivités possibles entre fragments. Il faut alors trouver le meilleur chemin dans ce graphe (chemin de coût minimal). Ces méthodes, basées sur l algorithme de Dijkstra, garantissent que l alignement obtenu est un optimum global, mais sont très sensiblement plus coûteuses en temps de calcul. 1.5 Comblement des «trous» Dans un projet de séquençage aléatoire «shotgun» à grande échelle, nous avons vu qu il est pratiquement inévitable qu il reste des régions d ADN non couvertes par les clones. Deux problèmes se posent donc à ce stade : Positionner les contigs disjoints les uns par rapport aux autres. Ceci revient à ordonner et à orienter les différents contigs sur le génome séquencé. Compléterlaséquencedechaquetrou. Cette phase du projet peut s avérer assez laborieuse, car une combinaison de méthodes ad hoc doit en général être mise en œuvre pour combler la totalité des trous. Parmi celles-ci, les trois principales sont l intégration des données d une carte génétique, la PCR par-dessus les trous et l utilisation d une autre banque d ADN, contenant en général des plus grands fragments (cosmides, BACs, YACs...). L intégration des cartes génétiques Bien souvent, pour de nombreux organismes modèles (Escherichia coli, levure, drosophile, souris...), il existe une carte génétique indiquant la position relative de divers loci sur le ou les chromosomes. Cette carte, obtenue par les méthodes classiques de mesure des fréquences de co-transmission de plusieurs marqueurs génétiques, donne une indication précise sur l ordonnancement des gènes associés, et une idée qualitative des distances qui les séparent (mesurées en centimorgans par les généticiens). Dans le cas du colibacille E. coli, par exemple, plus d un millier de loci génétiques avaient été identifiés et cartographiés avant que le séquençage systématique du génome ne soit entrepris. Un certain nombre de ces gènes ont souvent été clonés et séquencés de manière ponctuelle. L identification sur la séquence réalisée d un gène associé à une
18 26 Chapitre 1. Séquençage et Génome fonction connue et localisée sur le génome permet alors de placer de manière absolue le contig qui le contient. L amplification PCR des régions manquantes Lorsquelenombredecontigsetdoncdetrousn estpastropgrand,on peut essayer de combler certains de ces derniers par amplification PCR. Dans le cas d un génome bactérien, nous avons vu (Table 1) que le nombre de trous attendus est de l ordre de quelques dizaines. On synthétise alors des oligonucléotides dont la séquence correspond à celles des extrémités 3 des contigs obtenus. On tente alors d amplifier l ADN chromosomique en utilisant toutes les combinaisons de paires de ces oligonucléotides comme amorces. L amplification PCR ne donne de résultat positif que si les deux amorces correspondent à des séquences prises sur deux brins complémentaires et séparées de moins de quelques milliers de nucléotides. En d autres termes, lorsque les extrémités de deux contigs sont séparées par un trou de taille modeste ( 5 kpb), la PCR permettra d amplifier le segment d ADN chromosomique correspondant au trou manquant. Dans le cas d un résultat positif, la méthode permet donc non seulement de positionner deux contigs voisins, mais elle permet de déterminer la séquence manquante par séquençage du fragment d ADN obtenu par PCR. Fig. 1.11: Exemple simplifié de comblement de trous par PCR, avec deux contigs et deux trous sur un chromosome circulaire. Les amplifications PCR de l ADN chromosomique par les amorces 1 et 3, d une part, et 2 et 4, d autre part, donneront un résultat positif, alors que les autres combinaisons échoueront (1 & 4 et 2 & 3). En séquençant les fragments d ADN obtenus par PCR, on pourra ainsi mener le projet àsonterme.
19 Obstacles à la reconstruction 27 La méthode PCR est très séduisante, car elle répond à la fois au problème de l ordonnancement des contigs et à celui de la détermination des séquences manquantes. Elle se heurte malheureusement à des difficultés dans plusieurs cas, en particulier lorsque les trous sont trop grands pour que l amplification PCR donne un résultat. Dans d autres cas, des répétitions de séquence à l extrémité de divers contigs peuvent conduire à des ambiguïtés de raccordement du fait de l observation de faux positifs lors de la PCR. Enfin, lorsque le nombre de trous à combler est trop grand, cette méthode devient difficilement praticable. En effet, s il y a N contigs, donc 2N amorces PCR, cela représente 2N(N-1) amplifications PCR à effectuer. Si on reprend les données de la table 1, pour un génome bactérien, cela représente quelques centaines à quelques milliers de réactions, ce qui est accessible aux automates actuels. Pour un génome mammifère, cela représenterait plus d un milliard de réactions de PCR, ce qui est encore irréaliste. 1.6 Obstacles à la reconstruction Séquences répétées La plupart des génomes contiennent un certain nombre de séquences répétées. Chez les bactéries ou les eucaryotes inférieurs comme la levure, une fraction très élevée de l ADN chromosomique est «utile», c est-à-dire qu elle correspond à des régions codantes, transcrites en ARN et traduites en protéines. Malgré cela certaines séquences sont répétées plusieurs fois, à différents endroits du génome. Par exemple, les gènes codant pour les ARN ribosomiques sont en général présents à de multiples copies rigoureusement identiques, sur plusieurs milliers de paires de bases de long. On en dénombre ainsi sept chez Escherichia coli, et plus d une centaine chez la levure de bière Saccharomyces cerevisiae. Chez les eucaryotes supérieurs, où seulement une fraction minoritaire du génome est effectivement codante (2% environ chez l homme), on a identifié de nombreuses séquences répétées, dont l origine et la fonction précise sont parfois encore obscures. Celles-ci représentent une fraction tout à fait significative du génome. Ces séquences répétées sont des nuisances pour le séquençage génomique. Elles compliquent considérablement la tâche d alignement et de reconstruction du génome. Lorsqu un clone contient une telle séquence répétée, il va en effet donner des alignements potentiels avec tous les autres clones qui contiennent d autres copies de celle-ci. Ainsi, en plus des alignements avec ses voisins réels, on risque d obtenir des alignements indésirables. Les ambiguïtés inévitables
20 28 Chapitre 1. Séquençage et Génome qui en résultent risquent d induire des erreurs d aiguillage au moment de la reconstruction. Ces problèmes sont d autant plus difficiles que les séquences répétées sont longues et que leur taux d identité est élevé. Les «inclonables» Les méthodes de séquençage imposent systématiquement le clonage de fragments d ADN dans un vecteur de séquençage. Ces vecteurs recombinants doivent ensuite être introduits dans un hôte pour constituer la banque d ADN à séquencer. Il arrive que certains fragments de séquence exogènes ne puissent être propagés de manière stable dans le vecteur. Ceci peut résulter de la toxicité de certaines séquences pour l organisme hôte dans lequel est introduit le vecteur. De manière fortuite, certaines séquences d ADN peuvent par exemple conduire à l expression d un ARN ou d une protéine toxique pour l hôte, ou bien titrer et séquestrer un facteur essentiel de celui-ci. Le résultat est que la cellule portant ces vecteurs meurt. Une autre possibilité est que la séquence insérée dans le vecteur interfère avec la réplication de ce dernier. Dans ce dernier cas, celui-ci ne pouvant plus se transmettre aux deux cellules filles lors de la division cellulaire, il ne pourra se perpétuer dans la descendance de la cellule hôte. Il disparaît donc rapidement au fil des divisions cellulaires. Dans les deux cas, le fragment correspondant de l ADN génomique n est pas représenté dans la banque, il est dit «inclonable». Le résultat de cette difficulté technique, c est un biais d échantillonnage qui entraînera de manière inévitable un trou lors de la reconstruction des contigs. Si le trou est de taille raisonnable, l amplification par PCR permettra cependant de le combler, car il est possible de séquencer le fragment amplifié sans qu il soit nécessaire de le cloner. 1.7 Utilisation d une banque complémentaire de «grands 1»clones Pour vérifier que l ordonnancement des clones au sein des contigs est correct, ainsi que pour aider à combler les derniers trous par PCR, en particulier dans le cas des très grands génomes, il est pratiquement inévitable d avoir recours à une banque additionnelle contenant des fragments d ADN de plus grande taille (typiquement 20 à 100 kpb dans des cosmides ou BACs). Pour 1 Il s agit là d un abus de langage simplificateur. Ce ne sont pas les clones qui sont grands, mais les fragments d ADN insérés dans les vecteurs portés par les lignées clonales composant la banque.
21 Utilisation d une banque complémentaire de «grands» clones 29 couvrir l ensemble du génome, le nombre des clones requis pour constituer cette banque sera bien sûr beaucoup plus faible, puisqu ils portent des fragments d ADN 10 à 100 fois plus grands que ceux de la banque construite dans le vecteur de séquençage. Fig. 1.12: Utilisation des séquences extrêmes d un «grand clone» pour vérifier et guider l assemblage des contigs et combler les trous. On utilise le fait que la taille des fragments d ADN insérés dans ces vecteurs BACs ou cosmides peut être estimée très simplement par électrophorèse. On séquence ensuite les 500 nucléotides situés à chaque extrémité du grand fragment. On recherche alors ces deux séquences dans les contigs obtenus avec la première banque de clones. Si les deux extrémités sont trouvées dans le même contig, on peut alors vérifier que la distance entre celles-ci, dans le contig, est compatible avec la taille de l ADN inséré, mesurée par électrophorèse. Si les deux séquences appartiennent à deux contigs disjoints, cela permet de les positionner l un par rapport à l autre et d estimer la taille du trou qui les sépare, pour ensuite pouvoir effectuer son comblement. Par la vérification de la cohérence des distances lors de l assemblage, cette stratégie permet de s affranchir en grande partie des difficultés liées aux séquences répétées. Ces distances entre extrémités des grands clones peuvent même être directement introduites sous forme de contraintes pour l algorithme de reconstruction. Elle permet également de simplifier considérablement les problèmes de couverture des trous entre contigs par PCR, en réduisant le nombre de combinaisons d amorces à essayer. Enfin, dans les cas très défavorables où les trous sont trop grands pour être accessibles à la PCR, on peut utiliser le «grand» clone qui traverse la région manquante pour essayer
22 30 Chapitre 1. Séquençage et Génome de le séquencer directement. On utilise son ADN comme matrice et l on en synthétise des amorces de séquençage ad hoc, complémentaires des séquences nucléotidiques de part et d autre du trou. La seule différence avec le séquençage classique, c est que l on n utilise pas les amorces universelles décrites plus haut, mais des amorces spécifiques qui permettent de démarrer juste au bord de la zone manquante au lieu du bord de la séquence insérée dans le vecteur. On peut ainsi réduire progressivement la taille du trou jusqu à ce qu il soit accessible à la PCR. 1.8 Le premier de projet de séquençage à grande échelle : le génome de Haemophilus inuenzae Heamophilus influenzae est une petite bactérie qui colonise la sphère ORL chez l homme et cause de nombreuses infections respiratoires et otites, en particulier chez les jeunes enfants. Son génome est constitué d un unique chromosome circulaire long de 1,83 million de paires de bases, soit une taille relativement réduite. Par comparaison, le génome de sa cousine Escherichia coli dépasse 4,3 millions de paires de bases. Cette taille de génome restreinte, conjuguée avec l intérêt thérapeutique de cette bactérie en tant que cible, ont fait de cet organisme le candidat de choix pour le premier séquençage génomique complet par des méthodes automatisées qui a été réalisé en 1995 au TIGR (The Institute of Genome Research) aux Etats-Unis. Le synoptique du programme de séquençage est le suivant : L ADN génomique d H. influenzae aétésoumisàuncassagemécanique par ultrasons et les extrémités des fragments produits sont «égalisées» par un traitement avec une nucléase. Les fragments de taille comprise entre 1,6 et 2,0 kpb ont été sélectivement purifiés par électrophorèse. Ils ont ensuite été ligaturés dans un plasmide portant un marqueur de résistance à l ampicilline, un antibiotique de la famille de la pénicilline. Les plasmides recombinants obtenus ont été transformés dans E. coli.les colonies résultantes, cultivées sur milieu sélectif avec antibiotique, ont été purifiées et isolées. La banque ainsi obtenue contenait clones d E. coli portant chacun un plasmide recombinant ayant incorporé un fragment du génome d H. influenzae. L ADN double brin des plasmides a été préparé par des méthodes semi-robotisées, en utilisant des plaques à 96 puits, permettant 96 pu-
23 ADNc et EST 31 rifications en parallèle. L ADN inséré dans ces plasmides a été ensuite séquencé à partir soit d une seule, soit des deux extrémités, en utilisant des amorces complémentaires de chacun des deux cotés du site d insertion dans le vecteur. En tout séquences ont été réalisées, avec une longueur moyenne de 460 nucléotides lus par séquence. Ce travail a mobilisé 14 séquenceurs automatiques pendant une durée de 3 mois. L ensemble des séquences lues représentait 11,6 millions de nucléotides, soit un taux de couverture du génome de plus d un facteur 6. La reconstruction a été effectuée au moyen d un programme automatique, ce qui a permis d identifier 42 contigs (blocs connexes de séquence), séparés par autant de «trous». Ces trous ont été comblés par des méthodes ad hoc, souventlaborieuses: hybridation moléculaire, PCR, reclonage à partir d une autre banque d ADN contenant des fragments plus grands (15-20 kpb contre 1,6 à 2,0 pour la banque utilisée lors du séquençage aléatoire). La séquence complète de bp a ainsi pu être déterminée, avec un taux d erreur estimé à environ 1/ Le coût, hors infrastructures, a été estimé à environ 0,5 $ US par nucléotide dans la séquence finale (0,9 million de dollars). 1.9 ADNc et EST Toutes les méthodes de séquençage décrites ci-dessus sont appliquées à des grands fragments d ADN, ou même à des génomes entiers. Cette approche apporte une information globale, mais est relativement lourde dans sa mise en œuvre. Dans le cas des très grands génomes des eucaryotes supérieurs, seule une petite partie de celui-ci code effectivement pour des protéines (2 à 5%). En plus des grandes régions non-codantes séparant les gènes, ces derniers sont souvent morcelés par la présence d un grand nombre d introns, qui sont épissés dans l ARN messager mature. Le «meilleur» du génome, les parties codantes, est donc dispersé et minoritaire, c est pourquoi un certain nombre d équipes de recherche a, parallèlement à l approche globale, voulu se concentrer sur cette information essentielle, plus facile à extraire dans un premier temps. L approche suivie consiste à isoler les ARN messagers matures (i.e. débarrassés de leur introns) et à synthétiser des ADN complémentaires (ADNc) à partir de ceux-ci. Le protocole est décrit sur la figure 13 : Les ARN messagers sont purifiés à partir des tissus. Ceux-ci portent une queue poly-a en 3, ce qui permet de les purifier par chromatographie sur
24 32 Chapitre 1. Séquençage et Génome Fig : Construction d un ADNc à partir d un ARNm polyadénylé en 3. des résines où l on a greffé chimiquement du poly-t (le poly-t s apparie au poly-a). En utilisant également du poly-t comme amorce, on effectue la transcription inverse de l ARN en ADN. On utilise pour cela une transcriptase inverse virale qui possède également la propriété d hydrolyser le brin d ARN matrice au fur et à mesure de la polymérisation 2. Lorsque la transcription du premier brin d ADN est terminée, la polymérase «repart» dans l autre sens, en utilisant cette fois-ci l ADN comme matrice. Elle peut profiter pour cela d une structure en épingle à cheveux sur l extrémité du premier brin qui permet l amorçage du second brin. L ADN double brin résultant est une copie de l ARN messager, on l appelle ADNc (c pour complémentaire de l ARN, on dit cdna en anglais). Il est différent de la séquence de l ADN génomique, puisqu il est dépourvu des introns. L intérêt du clonage et du séquençage de ces ADNc est multiple. Il permet tout d abord de savoir quels gènes sont effectivement transcrits en ARNm dans une cellule donnée. Dans le contexte d un organisme complexe composé de cellules différenciées formant des organes et des tissus spécialisés, on peut ainsi identifier les profils de transcription de chaque type cellulaire. 2 Cette activité d hydrolyse spécifique du brin d ARN dans un hétéroduplex ARN : ADN s appelle activité Ribonucléase H.
25 ADNc et EST 33 L autre intérêt de cette approche est de permettre, en conjonction avec la séquence de l ADN génomique, de définir avec précision les bornes des introns et des exons et donc les sites d épissage. De plus, on peut identifier les variations tissulaires du processus d épissage. En fonction du contexte cellulaire, un même gène peut en effet donner naissance à plusieurs ARN messagers et donc à plusieurs variants protéiques, par un mécanisme d épissages alternatifs. On a ainsi baptisé transcriptome (par homologie avec génome), l ensemble des ARN messagers pouvant être transcrits à partir des chromosomes d une cellule. L information relative à ce transcriptome est accessible entre autres par le séquençage systématique des ADNc, mais serait difficile à obtenir à partir de la seule séquence génomique. Lors de la construction et de l analyse des ADNc, deux stratégies assez différentes peuvent être employées : Tout d abord, on peut chercher à obtenir les ADNc les plus longs possibles, afin qu ils couvrent la totalité du cadre ouvert de lecture correspondant au gène étudié. Ceci nécessite beaucoup de précaution lors de l extraction des ARN messagers. Toute dégradation de l ARNm conduira à la production d un ADNc incomplet. L obtention d un ADNc complet permet de déterminer la séquence de la totalité du cadre ouvert de lecture et d en déduire la séquence de la protéine correspondante. Le clonage de l ADNc portant ce cadre ouvert de lecture dans un vecteur génétique adapté permet ensuite la production de la protéine recombinante dans un hôte hétérologue, procaryote ou eucaryote. L ADNc étant en effet dépourvu d introns, il peut être traduit dans n importe quelle cellule, indépendamment de toute machinerie d épissage. Une utilisation alternative d un ADNc consiste à se contenter de déterminer la séquence de ses extrémités, même si celui-ci est incomplet. Ces séquences constituent des «signatures» qui permettent d identifier de manière univoque le gène correspondant. Cette stratégie a été utilisée de manière massive et systématique, pour tenter d identifier tous les gènes transcrits dans un type de cellule donnée. Ces fragments de séquence d ADNc sont appelés des EST (Expressed Sequence Tag, étiquette de séquence exprimée). Chez l homme, organisme le plus étudié, plus de 2 millions d EST ont ainsi été séquencés à ce jour (Printemps 2002), et pour huit autres espèces, le nombre d EST séquencés dépasse les : souris, rat, vache, nématode, drosophile pour le règne animal et arabette, soja et tomate, pour le règne végétal. Dans de nombreux cas, plusieurs EST correspondent à un même gène, et il est possible d appliquer les méthodes de reconstruction (assemblage de contig) décrites plus haut pour reconstituer la séquence de l ARN messager correspondant.
26 34 Chapitre 1. Séquençage et Génome Bibliographie Bonfield, J. K., Smith, K. & Staden, R. (1995). AnewDNAsequence assembly program. Nucleic Acids Res 23, Broder, S. & Venter, J. C. (2000). Whole genomes : the foundation of new biology and medicine. Curr Opin Biotechnol 11, Dear, S. & Staden, R. (1991). A sequence assembly and editing program for efficient management of large projects. Nucleic Acids Res 19, Fleischmann, R. D., Adams, M. D., White, O., Clayton, R. A., Kirkness, E. F., Kerlavage, A. R., Bult, C. J., Tomb, J. F., Dougherty, B. A., Merrick, J. M. & et al. (1995). Whole-genome random sequencing and assembly of Haemophilus influenzae Rd. Science 269, Lander, E. S., et al. (2001). Initial sequencing and analysis of the human genome. Nature409, Lander, E. S. & Waterman, M. S. (1988). Genomic mapping by fingerprinting random clones : a mathematical analysis. Genomics2, Myers, E. W., et al. (2000). A whole-genome assembly of Drosophila. Science287, Venter, J. C., et al. (2001). The sequence of the human genome. Science 291, Venter, J. C., Adams, M. D., Sutton, G. G., Kerlavage, A. R., Smith, H. O. & Hunkapiller, M. (1998). Shotgun sequencing of the human genome. Science 280,
3: Clonage d un gène dans un plasmide
 3: Clonage d un gène dans un plasmide Le clonage moléculaire est une des bases du génie génétique. Il consiste à insérer un fragment d'adn (dénommé insert) dans un vecteur approprié comme un plasmide par
3: Clonage d un gène dans un plasmide Le clonage moléculaire est une des bases du génie génétique. Il consiste à insérer un fragment d'adn (dénommé insert) dans un vecteur approprié comme un plasmide par
CHAPITRE 3 LA SYNTHESE DES PROTEINES
 CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
CHAITRE 3 LA SYNTHESE DES ROTEINES On sait qu un gène détient dans sa séquence nucléotidique, l information permettant la synthèse d un polypeptide. Ce dernier caractérisé par sa séquence d acides aminés
TD de Biochimie 4 : Coloration.
 TD de Biochimie 4 : Coloration. Synthèse de l expérience 2 Les questions posées durant l expérience 2 Exposé sur les méthodes de coloration des molécules : Générique Spécifique Autres Questions Pourquoi
TD de Biochimie 4 : Coloration. Synthèse de l expérience 2 Les questions posées durant l expérience 2 Exposé sur les méthodes de coloration des molécules : Générique Spécifique Autres Questions Pourquoi
Les OGM. 5 décembre 2008. Nicole Mounier
 Les OGM 5 décembre 2008 Nicole Mounier Université Claude Bernard Lyon 1 CGMC, bâtiment Gregor Mendel 43, boulevard du 11 Novembre 1918 69622 Villeurbanne Cedex OGM Organismes Génétiquement Modifiés Transfert
Les OGM 5 décembre 2008 Nicole Mounier Université Claude Bernard Lyon 1 CGMC, bâtiment Gregor Mendel 43, boulevard du 11 Novembre 1918 69622 Villeurbanne Cedex OGM Organismes Génétiquement Modifiés Transfert
Dr E. CHEVRET UE2.1 2013-2014. Aperçu général sur l architecture et les fonctions cellulaires
 Aperçu général sur l architecture et les fonctions cellulaires I. Introduction II. Les microscopes 1. Le microscope optique 2. Le microscope à fluorescence 3. Le microscope confocal 4. Le microscope électronique
Aperçu général sur l architecture et les fonctions cellulaires I. Introduction II. Les microscopes 1. Le microscope optique 2. Le microscope à fluorescence 3. Le microscope confocal 4. Le microscope électronique
TEST ELISA (ENZYME-LINKED IMMUNOSORBENT ASSEY)
 TEST ELISA (ENZYME-LINKED IMMUNOSORBENT ASSEY) Lise Vézina, technicienne de laboratoire Michel Lacroix, agronome-phytopathologiste Direction de l innovation scientifique et technologique Au Laboratoire
TEST ELISA (ENZYME-LINKED IMMUNOSORBENT ASSEY) Lise Vézina, technicienne de laboratoire Michel Lacroix, agronome-phytopathologiste Direction de l innovation scientifique et technologique Au Laboratoire
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST.
 La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
La gestion de données dans le cadre d une application de recherche d alignement de séquence : BLAST. Gaël Le Mahec - p. 1/12 L algorithme BLAST. Basic Local Alignment Search Tool est un algorithme de recherche
Plateforme Transgenèse/Zootechnie/Exploration Fonctionnelle IBiSA. «Anexplo» Service Transgenèse. Catalogue des prestations
 Plateforme Transgenèse/Zootechnie/Exploration Fonctionnelle IBiSA «Anexplo» Service Transgenèse Catalogue des prestations 04/01/12 - Page 1 sur 8 Présentation du service de Transgenèse Le service de Transgenèse
Plateforme Transgenèse/Zootechnie/Exploration Fonctionnelle IBiSA «Anexplo» Service Transgenèse Catalogue des prestations 04/01/12 - Page 1 sur 8 Présentation du service de Transgenèse Le service de Transgenèse
Biologie Appliquée. Dosages Immunologiques TD9 Mai 2015. Stéphanie Sigaut INSERM U1141 [email protected]
 Biologie Appliquée Dosages Immunologiques TD9 Mai 2015 Stéphanie Sigaut INSERM U1141 [email protected] 1 ELISA 2 3 4 [Ac] 5 6 7 8 9 Correction : Faire la moyenne D0-1 et D0-2 pour toute les valeurs
Biologie Appliquée Dosages Immunologiques TD9 Mai 2015 Stéphanie Sigaut INSERM U1141 [email protected] 1 ELISA 2 3 4 [Ac] 5 6 7 8 9 Correction : Faire la moyenne D0-1 et D0-2 pour toute les valeurs
Conférence technique internationale de la FAO
 Décembre 2009 ABDC-10/7.2 F Conférence technique internationale de la FAO Biotechnologies agricoles dans les pays en développement: choix et perspectives pour les cultures, les forêts, l élevage, les pêches
Décembre 2009 ABDC-10/7.2 F Conférence technique internationale de la FAO Biotechnologies agricoles dans les pays en développement: choix et perspectives pour les cultures, les forêts, l élevage, les pêches
Ne laissez pas le stockage cloud pénaliser votre retour sur investissement
 Ne laissez pas le stockage cloud pénaliser votre retour sur investissement Préparé par : George Crump, analyste senior Préparé le : 03/10/2012 L investissement qu une entreprise fait dans le domaine de
Ne laissez pas le stockage cloud pénaliser votre retour sur investissement Préparé par : George Crump, analyste senior Préparé le : 03/10/2012 L investissement qu une entreprise fait dans le domaine de
Gènes Diffusion - EPIC 2010
 Gènes Diffusion - EPIC 2010 1. Contexte. 2. Notion de génétique animale. 3. Profil de l équipe plateforme. 4. Type et gestion des données biologiques. 5. Environnement Matériel et Logiciel. 6. Analyses
Gènes Diffusion - EPIC 2010 1. Contexte. 2. Notion de génétique animale. 3. Profil de l équipe plateforme. 4. Type et gestion des données biologiques. 5. Environnement Matériel et Logiciel. 6. Analyses
IMMUNOLOGIE. La spécificité des immunoglobulines et des récepteurs T. Informations scientifiques
 IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
IMMUNOLOGIE La spécificité des immunoglobulines et des récepteurs T Informations scientifiques L infection par le VIH entraîne des réactions immunitaires de l organisme qui se traduisent par la production
Critères pour les méthodes de quantification des résidus potentiellement allergéniques de protéines de collage dans le vin (OIV-Oeno 427-2010)
 Méthode OIV- -MA-AS315-23 Type de méthode : critères Critères pour les méthodes de quantification des résidus potentiellement allergéniques de protéines de collage (OIV-Oeno 427-2010) 1 Définitions des
Méthode OIV- -MA-AS315-23 Type de méthode : critères Critères pour les méthodes de quantification des résidus potentiellement allergéniques de protéines de collage (OIV-Oeno 427-2010) 1 Définitions des
Big data et sciences du Vivant L'exemple du séquençage haut débit
 Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard [email protected] INRA - MIAT - Plate-forme
Big data et sciences du Vivant L'exemple du séquençage haut débit C. Gaspin, C. Hoede, C. Klopp, D. Laborie, J. Mariette, C. Noirot, MS. Trotard [email protected] INRA - MIAT - Plate-forme
MAB Solut. vos projets. MABLife Génopole Campus 1 5 rue Henri Desbruères 91030 Evry Cedex. www.mabsolut.com. intervient à chaque étape de
 Mabsolut-DEF-HI:Mise en page 1 17/11/11 17:45 Page1 le département prestataire de services de MABLife de la conception à la validation MAB Solut intervient à chaque étape de vos projets Création d anticorps
Mabsolut-DEF-HI:Mise en page 1 17/11/11 17:45 Page1 le département prestataire de services de MABLife de la conception à la validation MAB Solut intervient à chaque étape de vos projets Création d anticorps
Biomarqueurs en Cancérologie
 Biomarqueurs en Cancérologie Définition, détermination, usage Biomarqueurs et Cancer: définition Anomalie(s) quantitative(s) ou qualitative(s) Indicative(s) ou caractéristique(s) d un cancer ou de certaines
Biomarqueurs en Cancérologie Définition, détermination, usage Biomarqueurs et Cancer: définition Anomalie(s) quantitative(s) ou qualitative(s) Indicative(s) ou caractéristique(s) d un cancer ou de certaines
Les outils de génétique moléculaire Les techniques liées aux acides nucléiques
 Les outils de génétique moléculaire Les techniques liées aux acides nucléiques Sommaire Preparation des acides nucléiques Extraction / purification Les enzymes agissant sur les acides nucléiques Les enzymes
Les outils de génétique moléculaire Les techniques liées aux acides nucléiques Sommaire Preparation des acides nucléiques Extraction / purification Les enzymes agissant sur les acides nucléiques Les enzymes
Cellules procaryotes Service histologie Pr.k.mebarek
 Cellules procaryotes Service histologie Pr.k.mebarek I) Les cellules procaryotes II) Les cellules eucaryotes o 1) Caractéristiques générales des cellules eucaryotes o 2) Organisation des cellules eucaryotes
Cellules procaryotes Service histologie Pr.k.mebarek I) Les cellules procaryotes II) Les cellules eucaryotes o 1) Caractéristiques générales des cellules eucaryotes o 2) Organisation des cellules eucaryotes
4 : MÉTHODES D ANALYSE UTILISÉES EN ÉCOLOGIE MICROBIENNE
 4 : MÉTHODES D ANALYSE UTILISÉES EN ÉCOLOGIE MICROBIENNE L écologie microbienne (ou étude des micro-organismes de l environnement) étudie : les relations entre les différentes populations de micro-organismes
4 : MÉTHODES D ANALYSE UTILISÉES EN ÉCOLOGIE MICROBIENNE L écologie microbienne (ou étude des micro-organismes de l environnement) étudie : les relations entre les différentes populations de micro-organismes
Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant
 Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant Parcours: Master 1 : Bioinformatique et biologie des Systèmes dans le Master
Master de Bioinformatique et Biologie des Systèmes Toulouse http://m2pbioinfo.biotoul.fr Responsable : Pr. Gwennaele Fichant Parcours: Master 1 : Bioinformatique et biologie des Systèmes dans le Master
SEQUENÇAGE LI-COR DNA 4200
 SEQUENÇAGE LI-COR DNA 4200 Le gel de séquence contient 64 puits au maximum soit 16 clones traités en parallèle. Les oligos utilisés (modifiés en 5 ) fluorescent à 700/800 nm. Une amorce permet de séquencer
SEQUENÇAGE LI-COR DNA 4200 Le gel de séquence contient 64 puits au maximum soit 16 clones traités en parallèle. Les oligos utilisés (modifiés en 5 ) fluorescent à 700/800 nm. Une amorce permet de séquencer
Vue d ensemble : Office of Cellular, Tissue and Gene Therapies
 Vue d ensemble : Office of Cellular, Tissue and Gene Therapies DIAPOSITIVE 1 Cette présentation fournit une vue d ensemble de l Office of Cellular, Tissue, and Gene Therapies (bureau des thérapies cellulaires,
Vue d ensemble : Office of Cellular, Tissue and Gene Therapies DIAPOSITIVE 1 Cette présentation fournit une vue d ensemble de l Office of Cellular, Tissue, and Gene Therapies (bureau des thérapies cellulaires,
Chapitre 2 : Systèmes radio mobiles et concepts cellulaires
 Chapitre 2 : Systèmes radio mobiles et concepts cellulaires Systèmes cellulaires Réseaux cellulaires analogiques de 1ère génération : AMPS (USA), NMT(Scandinavie), TACS (RU)... Réseaux numériques de 2ème
Chapitre 2 : Systèmes radio mobiles et concepts cellulaires Systèmes cellulaires Réseaux cellulaires analogiques de 1ère génération : AMPS (USA), NMT(Scandinavie), TACS (RU)... Réseaux numériques de 2ème
Biochimie I. Extraction et quantification de l hexokinase dans Saccharomyces cerevisiae 1. Assistants : Tatjana Schwabe Marcy Taylor Gisèle Dewhurst
 Biochimie I Extraction et quantification de l hexokinase dans Saccharomyces cerevisiae 1 Daniel Abegg Sarah Bayat Alexandra Belfanti Assistants : Tatjana Schwabe Marcy Taylor Gisèle Dewhurst Laboratoire
Biochimie I Extraction et quantification de l hexokinase dans Saccharomyces cerevisiae 1 Daniel Abegg Sarah Bayat Alexandra Belfanti Assistants : Tatjana Schwabe Marcy Taylor Gisèle Dewhurst Laboratoire
EXERCICES : MECANISMES DE L IMMUNITE : pages 406 407 408 409 410
 EXERCICES : MECANISMES DE L IMMUNITE : pages 406 407 408 409 410 EXERCICE 1 PAGE 406 : EXPERIENCES A INTERPRETER Question : rôles respectifs du thymus et de la moelle osseuse dans la production des lymphocytes.
EXERCICES : MECANISMES DE L IMMUNITE : pages 406 407 408 409 410 EXERCICE 1 PAGE 406 : EXPERIENCES A INTERPRETER Question : rôles respectifs du thymus et de la moelle osseuse dans la production des lymphocytes.
CATALOGUE DES PRESTATIONS DE LA
 1/23 La plate-forme Biopuces et Séquençage de Strasbourg est équipée des technologies Affymetrix et Agilent pour l étude du transcriptome et du génome sur puces à ADN. SOMMAIRE ANALYSE TRANSCRIPTIONNELLE...
1/23 La plate-forme Biopuces et Séquençage de Strasbourg est équipée des technologies Affymetrix et Agilent pour l étude du transcriptome et du génome sur puces à ADN. SOMMAIRE ANALYSE TRANSCRIPTIONNELLE...
Nombres, mesures et incertitudes en sciences physiques et chimiques. Groupe des Sciences physiques et chimiques de l IGEN
 Nombres, mesures et incertitudes en sciences physiques et chimiques. Groupe des Sciences physiques et chimiques de l IGEN Table des matières. Introduction....3 Mesures et incertitudes en sciences physiques
Nombres, mesures et incertitudes en sciences physiques et chimiques. Groupe des Sciences physiques et chimiques de l IGEN Table des matières. Introduction....3 Mesures et incertitudes en sciences physiques
LES OUTILS DU TRAVAIL COLLABORATIF
 LES OUTILS DU TRAVAIL COLLABORATIF Lorraine L expression «travail collaboratif» peut se définir comme «l utilisation de ressources informatiques dans le contexte d un projet réalisé par les membres d un
LES OUTILS DU TRAVAIL COLLABORATIF Lorraine L expression «travail collaboratif» peut se définir comme «l utilisation de ressources informatiques dans le contexte d un projet réalisé par les membres d un
Hépatite chronique B Moyens thérapeutiques
 Hépatite chronique B Moyens thérapeutiques Dr Olfa BAHRI Laboratoire de Virologie Clinique Institut Pasteur de Tunis INTRODUCTION Plus de 300. 10 6 porteurs chroniques de VHB dans le monde Hépatite chronique
Hépatite chronique B Moyens thérapeutiques Dr Olfa BAHRI Laboratoire de Virologie Clinique Institut Pasteur de Tunis INTRODUCTION Plus de 300. 10 6 porteurs chroniques de VHB dans le monde Hépatite chronique
MYRIAD. l ADN isolé n est à présent plus brevetable!
 MYRIAD La Cour Suprême des Etats-Unis revient sur plus de 30 ans de pratique : l ADN isolé n est à présent plus brevetable! Mauvaise passe pour les inventions en biotechnologies sur le territoire américain.
MYRIAD La Cour Suprême des Etats-Unis revient sur plus de 30 ans de pratique : l ADN isolé n est à présent plus brevetable! Mauvaise passe pour les inventions en biotechnologies sur le territoire américain.
Génétique et génomique Pierre Martin
 Génétique et génomique Pierre Martin Principe de la sélections Repérage des animaux intéressants X Accouplements Programmés Sélection des meilleurs mâles pour la diffusion Index diffusés Indexation simultanée
Génétique et génomique Pierre Martin Principe de la sélections Repérage des animaux intéressants X Accouplements Programmés Sélection des meilleurs mâles pour la diffusion Index diffusés Indexation simultanée
Format de l avis d efficience
 AVIS D EFFICIENCE Format de l avis d efficience Juillet 2013 Commission évaluation économique et de santé publique Ce document est téléchargeable sur www.has-sante.fr Haute Autorité de santé Service documentation
AVIS D EFFICIENCE Format de l avis d efficience Juillet 2013 Commission évaluation économique et de santé publique Ce document est téléchargeable sur www.has-sante.fr Haute Autorité de santé Service documentation
Plateforme. DArT (Diversity Array Technology) Pierre Mournet
 Plateforme DArT (Diversity Array Technology) Pierre Mournet Lundi 8 avril 203 Pourquoi des DArT? Développement rapide et peu onéreux de marqueurs : Pas d information de séquence nécessaire. Pas de pré-requis
Plateforme DArT (Diversity Array Technology) Pierre Mournet Lundi 8 avril 203 Pourquoi des DArT? Développement rapide et peu onéreux de marqueurs : Pas d information de séquence nécessaire. Pas de pré-requis
ANTICORPS POLYCLONAUX ANTI IMMUNOGLOBULINES
 L OUTIL IDEAL POUR TOUTES LES DETECTIONS IMMUNOCHIMIQUES pour toutes les techniques immunodosages (EIA/ELISA) dot/ westernblot immunohistochimie immunocytochimie cytométrie en flux quel que soit le système
L OUTIL IDEAL POUR TOUTES LES DETECTIONS IMMUNOCHIMIQUES pour toutes les techniques immunodosages (EIA/ELISA) dot/ westernblot immunohistochimie immunocytochimie cytométrie en flux quel que soit le système
SOCLE COMMUN - La Compétence 3 Les principaux éléments de mathématiques et la culture scientifique et technologique
 SOCLE COMMUN - La Compétence 3 Les principaux éléments de mathématiques et la culture scientifique et technologique DOMAINE P3.C3.D1. Pratiquer une démarche scientifique et technologique, résoudre des
SOCLE COMMUN - La Compétence 3 Les principaux éléments de mathématiques et la culture scientifique et technologique DOMAINE P3.C3.D1. Pratiquer une démarche scientifique et technologique, résoudre des
5.5.5 Exemple d un essai immunologique
 5.5.5 Exemple d un essai immunologique Test de grossesse Test en forme de bâtonnet destiné à mettre en évidence l'hormone spécifique de la grossesse, la gonadotrophine chorionique humaine (hcg), une glycoprotéine.
5.5.5 Exemple d un essai immunologique Test de grossesse Test en forme de bâtonnet destiné à mettre en évidence l'hormone spécifique de la grossesse, la gonadotrophine chorionique humaine (hcg), une glycoprotéine.
Intrants médicamenteux en agriculture et en santé : les écosystèmes microbiens sont-ils un problème ou une solution?
 Les Rencontres de l Inra au Salon de l agriculture Intrants médicamenteux en agriculture et en santé : les écosystèmes microbiens sont-ils un problème ou une solution? Lundi 23 février 2015 Programme 14h30
Les Rencontres de l Inra au Salon de l agriculture Intrants médicamenteux en agriculture et en santé : les écosystèmes microbiens sont-ils un problème ou une solution? Lundi 23 février 2015 Programme 14h30
Production d une protéine recombinante
 99 Production d une protéine recombinante Lic. B. PIRSON Lic. J-M. SERONT ISICHt - Mons Production de la protéine recombinante GFP (Green Fluorescent Protein d Aequoria victoria) par une bactérie ( E.
99 Production d une protéine recombinante Lic. B. PIRSON Lic. J-M. SERONT ISICHt - Mons Production de la protéine recombinante GFP (Green Fluorescent Protein d Aequoria victoria) par une bactérie ( E.
L immunoenzymologie. Technique puissante couramment utilisée e en recherche et en diagnostic cificité des anticorps pour leurs nes
 L immunoenzymologie Technique puissante couramment utilisée e en recherche et en diagnostic Basée e sur la très s grande spécificit cificité des anticorps pour leurs antigènes nes Test qualitatif Détection
L immunoenzymologie Technique puissante couramment utilisée e en recherche et en diagnostic Basée e sur la très s grande spécificit cificité des anticorps pour leurs antigènes nes Test qualitatif Détection
LES BIOTECHNOLOGIES DANS LE DIAGNOSTIC DES MALADIES INFECTIEUSES ET LE DÉVELOPPEMENT DES VACCINS
 CHAPITRE 1.1.7. LES BIOTECHNOLOGIES DANS LE DIAGNOSTIC DES MALADIES INFECTIEUSES ET LE DÉVELOPPEMENT DES VACCINS INTRODUCTION Les méthodes de biologie moléculaire ont de plus en plus d applications dans
CHAPITRE 1.1.7. LES BIOTECHNOLOGIES DANS LE DIAGNOSTIC DES MALADIES INFECTIEUSES ET LE DÉVELOPPEMENT DES VACCINS INTRODUCTION Les méthodes de biologie moléculaire ont de plus en plus d applications dans
Information génétique
 chapitre 3 Information génétique et division cellulaire L étude de la division cellulaire est abordée pour découvrir comment est transmise et conservée l information génétique portée par les chromosomes.
chapitre 3 Information génétique et division cellulaire L étude de la division cellulaire est abordée pour découvrir comment est transmise et conservée l information génétique portée par les chromosomes.
Comment suivre l évolution d une transformation chimique? + S 2 O 8 = I 2 + 2 SO 4
 Afin d optimiser leurs procédés, les industries chimiques doivent contrôler le bon déroulement de la réaction de synthèse menant aux espèces voulues. Comment suivre l évolution d une transformation chimique?
Afin d optimiser leurs procédés, les industries chimiques doivent contrôler le bon déroulement de la réaction de synthèse menant aux espèces voulues. Comment suivre l évolution d une transformation chimique?
évaluation des risques professionnels
 évaluation des professionnels Inventaire des Etablissement : Faculté de Médecine Unité de travail : Laboratoire de Biochimie Médicale Année : 2013 Locaux Bureaux Salle de Microscopie Culture cellulaire
évaluation des professionnels Inventaire des Etablissement : Faculté de Médecine Unité de travail : Laboratoire de Biochimie Médicale Année : 2013 Locaux Bureaux Salle de Microscopie Culture cellulaire
Ebauche Rapport finale
 Ebauche Rapport finale Sommaire : 1 - Introduction au C.D.N. 2 - Définition de la problématique 3 - Etat de l'art : Présentatio de 3 Topologies streaming p2p 1) INTRODUCTION au C.D.N. La croissance rapide
Ebauche Rapport finale Sommaire : 1 - Introduction au C.D.N. 2 - Définition de la problématique 3 - Etat de l'art : Présentatio de 3 Topologies streaming p2p 1) INTRODUCTION au C.D.N. La croissance rapide
Chapitre III Le phénotype immunitaire au cours de la vie
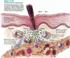 Chapitre III Le phénotype immunitaire au cours de la vie Le phénotype immunitaire d un individu caractérise sa capacité à répondre, grâce aux effecteurs de l immunité adaptative, aux différents agents
Chapitre III Le phénotype immunitaire au cours de la vie Le phénotype immunitaire d un individu caractérise sa capacité à répondre, grâce aux effecteurs de l immunité adaptative, aux différents agents
MABioVis. Bio-informatique et la
 MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
MABioVis Modèles et Algorithmes pour la Bio-informatique et la Visualisation Visite ENS Cachan 5 janvier 2011 MABioVis G GUY MELANÇON (PR UFR Maths Info / EPI GRAVITE) (là, maintenant) - MABioVis DAVID
www.gbo.com/bioscience 1 Culture Cellulaire Microplaques 2 HTS- 3 Immunologie/ HLA 4 Microbiologie/ Bactériologie Containers 5 Tubes/ 6 Pipetage
 2 HTS 3 Immunologie / Immunologie Informations Techniques 3 I 2 ELISA 96 Puits 3 I 4 ELISA 96 Puits en Barrettes 3 I 6 en Barrettes de 8 Puits 3 I 7 en Barrettes de 12 Puits 3 I 8 en Barrettes de 16 Puits
2 HTS 3 Immunologie / Immunologie Informations Techniques 3 I 2 ELISA 96 Puits 3 I 4 ELISA 96 Puits en Barrettes 3 I 6 en Barrettes de 8 Puits 3 I 7 en Barrettes de 12 Puits 3 I 8 en Barrettes de 16 Puits
Cours n 12. Technologies WAN 2nd partie
 Cours n 12 Technologies WAN 2nd partie 1 Sommaire Aperçu des technologies WAN Technologies WAN Conception d un WAN 2 Lignes Louées Lorsque des connexions dédiées permanentes sont nécessaires, des lignes
Cours n 12 Technologies WAN 2nd partie 1 Sommaire Aperçu des technologies WAN Technologies WAN Conception d un WAN 2 Lignes Louées Lorsque des connexions dédiées permanentes sont nécessaires, des lignes
Spécialité auxiliaire en prothèse dentaire du brevet d études professionnelles. ANNEXE IIb DEFINITION DES EPREUVES
 ANNEXE IIb DEFINITION DES EPREUVES 51 Epreuve EP1 : ANALYSE ET COMMUNICATION TECHNOLOGIQUES UP1 Coefficient 4 Finalité et objectifs de l épreuve L épreuve vise à évaluer la capacité du candidat à mobiliser
ANNEXE IIb DEFINITION DES EPREUVES 51 Epreuve EP1 : ANALYSE ET COMMUNICATION TECHNOLOGIQUES UP1 Coefficient 4 Finalité et objectifs de l épreuve L épreuve vise à évaluer la capacité du candidat à mobiliser
LIVRE BLANC Décembre 2014
 PARSING MATCHING EQUALITY SEARCH LIVRE BLANC Décembre 2014 Introduction L analyse des tendances du marché de l emploi correspond à l évidence à une nécessité, surtout en période de tension comme depuis
PARSING MATCHING EQUALITY SEARCH LIVRE BLANC Décembre 2014 Introduction L analyse des tendances du marché de l emploi correspond à l évidence à une nécessité, surtout en période de tension comme depuis
2 e partie de la composante majeure (8 points) Les questions prennent appui sur six documents A, B, C, D, E, F (voir pages suivantes).
 SUJET DE CONCOURS Sujet Exploitation d une documentation scientifique sur le thème de l énergie 2 e partie de la composante majeure (8 points) Les questions prennent appui sur six documents A, B, C, D,
SUJET DE CONCOURS Sujet Exploitation d une documentation scientifique sur le thème de l énergie 2 e partie de la composante majeure (8 points) Les questions prennent appui sur six documents A, B, C, D,
Contrôle de l'expression génétique : Les régulations post-transcriptionnelles
 Contrôle de l'expression génétique : Les régulations post-transcriptionnelles http://perso.univ-rennes1.fr/serge.hardy/ utilisateur : biochimie mot de passe : 2007 L'ARNm, simple intermédiaire entre le
Contrôle de l'expression génétique : Les régulations post-transcriptionnelles http://perso.univ-rennes1.fr/serge.hardy/ utilisateur : biochimie mot de passe : 2007 L'ARNm, simple intermédiaire entre le
DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION
 DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION PRINCIPES DE BASE SUR LES DONNEES ET LE CALCUL HAUTE PERFORMANCE Lois de Gray sur l ingénierie des données 1 : Les calculs scientifiques traitent des volumes considérables
DÉFIS DU SÉQUENÇAGE NOUVELLE GÉNÉRATION PRINCIPES DE BASE SUR LES DONNEES ET LE CALCUL HAUTE PERFORMANCE Lois de Gray sur l ingénierie des données 1 : Les calculs scientifiques traitent des volumes considérables
Environmental Research and Innovation ( ERIN )
 DÉpartEment RDI Environmental Research and Innovation ( ERIN ) Le département «Environmental Research and Innovation» (ERIN) du LIST élabore des stratégies, des technologies et des outils visant à mieux
DÉpartEment RDI Environmental Research and Innovation ( ERIN ) Le département «Environmental Research and Innovation» (ERIN) du LIST élabore des stratégies, des technologies et des outils visant à mieux
Chapitre 1 : Introduction aux bases de données
 Chapitre 1 : Introduction aux bases de données Les Bases de Données occupent aujourd'hui une place de plus en plus importante dans les systèmes informatiques. Les Systèmes de Gestion de Bases de Données
Chapitre 1 : Introduction aux bases de données Les Bases de Données occupent aujourd'hui une place de plus en plus importante dans les systèmes informatiques. Les Systèmes de Gestion de Bases de Données
Sciences de Gestion Spécialité : SYSTÈMES D INFORMATION DE GESTION
 Sciences de Gestion Spécialité : SYSTÈMES D INFORMATION DE GESTION Classe de terminale de la série Sciences et Technologie du Management et de la Gestion Préambule Présentation Les technologies de l information
Sciences de Gestion Spécialité : SYSTÈMES D INFORMATION DE GESTION Classe de terminale de la série Sciences et Technologie du Management et de la Gestion Préambule Présentation Les technologies de l information
Thème sélection génétique des plantes hybridation et génie génétique
 Thème sélection génétique des plantes hybridation et génie génétique Exemple d activité : recenser, extraire et exploiter des informations afin de comprendre les caractéristiques et les limites de la modification
Thème sélection génétique des plantes hybridation et génie génétique Exemple d activité : recenser, extraire et exploiter des informations afin de comprendre les caractéristiques et les limites de la modification
Maps Tableau de bord avec MindManager et Power Markers
 Maps Tableau de bord avec MindManager et Power Markers Nick Duffill, Harport Consulting traduction Martine Jésus Qu est-ce exactement qu une map tableau de bord? Qu est-ce qu une map tableau de bord? N
Maps Tableau de bord avec MindManager et Power Markers Nick Duffill, Harport Consulting traduction Martine Jésus Qu est-ce exactement qu une map tableau de bord? Qu est-ce qu une map tableau de bord? N
Infolettre #18 : Les graphiques avec Excel 2010
 Infolettre #18 : Les graphiques avec Excel 2010 Table des matières Introduction... 1 Hourra! Le retour du double-clic... 1 Modifier le graphique... 4 Onglet Création... 4 L onglet Disposition... 7 Onglet
Infolettre #18 : Les graphiques avec Excel 2010 Table des matières Introduction... 1 Hourra! Le retour du double-clic... 1 Modifier le graphique... 4 Onglet Création... 4 L onglet Disposition... 7 Onglet
Procédure d utilisation du Beckman CEQ 2000 XL pour la réalisation de programmes de séquençage ou de génotypage.
 MODE OPERATOIRE Code : SSG / 003 UMR 1229 MGS Microbiologie et Géochimie des Sols 17 rue Sully BP86510 21065 Dijon Cedex Rédigé par : D. Bru / S. Hallet. Procédure d utilisation du Beckman CEQ 2000 XL
MODE OPERATOIRE Code : SSG / 003 UMR 1229 MGS Microbiologie et Géochimie des Sols 17 rue Sully BP86510 21065 Dijon Cedex Rédigé par : D. Bru / S. Hallet. Procédure d utilisation du Beckman CEQ 2000 XL
Réseaux grande distance
 Chapitre 5 Réseaux grande distance 5.1 Définition Les réseaux à grande distance (WAN) reposent sur une infrastructure très étendue, nécessitant des investissements très lourds. Contrairement aux réseaux
Chapitre 5 Réseaux grande distance 5.1 Définition Les réseaux à grande distance (WAN) reposent sur une infrastructure très étendue, nécessitant des investissements très lourds. Contrairement aux réseaux
LA MITOSE CUEEP - USTL DÉPARTEMENT SCIENCES BAHIJA DELATTRE
 Biologie LA MITOSE CUEEP - USTL DÉPARTEMENT SCIENCES BAHIJA DELATTRE Février 2006 I. L'INTRODUCTION Chaque cellule d'un organisme supérieur provient de la multiplication d'une cellule préexistante (cellule
Biologie LA MITOSE CUEEP - USTL DÉPARTEMENT SCIENCES BAHIJA DELATTRE Février 2006 I. L'INTRODUCTION Chaque cellule d'un organisme supérieur provient de la multiplication d'une cellule préexistante (cellule
Rapport Scientifique Seine-Aval 3
 Rapport Scientifique Seine-Aval 3 Séminaire Seine-Aval 2008 Fiches de synthèse des propositions SA4 THEME 3 : TABLEAU DE BORD ET INDICATEURS OPERATIONNELS PUCES A ADN CHEZ MYTILUS EDULIS - 2005 - Danger
Rapport Scientifique Seine-Aval 3 Séminaire Seine-Aval 2008 Fiches de synthèse des propositions SA4 THEME 3 : TABLEAU DE BORD ET INDICATEURS OPERATIONNELS PUCES A ADN CHEZ MYTILUS EDULIS - 2005 - Danger
Consensus Scientifique sur. les. Champs statiques
 page 1/8 Consensus Scientifique sur Source : OMS (2006) les Champs statiques Résumé & Détails: GreenFacts Contexte - Les équipements d imagerie médicale par résonance magnétique (IRM), les trains et les
page 1/8 Consensus Scientifique sur Source : OMS (2006) les Champs statiques Résumé & Détails: GreenFacts Contexte - Les équipements d imagerie médicale par résonance magnétique (IRM), les trains et les
AGRÉGATION DE SCIENCES DE LA VIE - SCIENCES DE LA TERRE ET DE L UNIVERS
 AGRÉGATION DE SCIENCES DE LA VIE - SCIENCES DE LA TERRE ET DE L UNIVERS CONCOURS EXTERNE ÉPREUVES D ADMISSION session 2010 TRAVAUX PRATIQUES DE CONTRE-OPTION DU SECTEUR A CANDIDATS DES SECTEURS B ET C
AGRÉGATION DE SCIENCES DE LA VIE - SCIENCES DE LA TERRE ET DE L UNIVERS CONCOURS EXTERNE ÉPREUVES D ADMISSION session 2010 TRAVAUX PRATIQUES DE CONTRE-OPTION DU SECTEUR A CANDIDATS DES SECTEURS B ET C
ATELIER IMAGEJ. Différentes applications vous sont proposées pour apprendre à utiliser quelques fonctions d ImageJ :
 Différentes applications vous sont proposées pour apprendre à utiliser quelques fonctions d ImageJ : 1. ANALYSE QUANTITATIVE D UN GEL D ELECTROPHORESE... 2 2. NUMERATION DE COLONIES BACTERIENNES SUR UNE
Différentes applications vous sont proposées pour apprendre à utiliser quelques fonctions d ImageJ : 1. ANALYSE QUANTITATIVE D UN GEL D ELECTROPHORESE... 2 2. NUMERATION DE COLONIES BACTERIENNES SUR UNE
AGREGATION DE BIOCHIMIE GENIE BIOLOGIQUE
 AGREGATION DE BIOCHIMIE GENIE BIOLOGIQUE CONCOURS EXTERNE Session 2005 TRAVAUX PRATIQUES DE BIOCHIMIE PHYSIOLOGIE ALCOOL ET FOIE L éthanol, psychotrope puissant, est absorbé passivement dans l intestin
AGREGATION DE BIOCHIMIE GENIE BIOLOGIQUE CONCOURS EXTERNE Session 2005 TRAVAUX PRATIQUES DE BIOCHIMIE PHYSIOLOGIE ALCOOL ET FOIE L éthanol, psychotrope puissant, est absorbé passivement dans l intestin
Mise en place d une solution automatique de stockage et de visualisation de données de capture des interactions chromatiniennes à l échelle génomique
 Rapport de stage de deuxième année de DUT Génie Biologique option Bioinformatique Mise en place d une solution automatique de stockage et de visualisation de données de capture des interactions chromatiniennes
Rapport de stage de deuxième année de DUT Génie Biologique option Bioinformatique Mise en place d une solution automatique de stockage et de visualisation de données de capture des interactions chromatiniennes
Mise en pratique : Etude de spectres
 Mise en pratique : Etude de spectres Introduction La nouvelle génération de spectromètre à détecteur CCD permet de réaliser n importe quel spectre en temps réel sur toute la gamme de longueur d onde. La
Mise en pratique : Etude de spectres Introduction La nouvelle génération de spectromètre à détecteur CCD permet de réaliser n importe quel spectre en temps réel sur toute la gamme de longueur d onde. La
LES OUTILS D ALIMENTATION DU REFERENTIEL DE DB-MAIN
 LES OUTILS D ALIMENTATION DU REFERENTIEL DE DB-MAIN Les contenues de ce document sont la propriété exclusive de la société REVER. Ils ne sont transmis qu à titre d information et ne peuvent en aucun cas
LES OUTILS D ALIMENTATION DU REFERENTIEL DE DB-MAIN Les contenues de ce document sont la propriété exclusive de la société REVER. Ils ne sont transmis qu à titre d information et ne peuvent en aucun cas
Travaux dirigés de Microbiologie Master I Sciences des Génomes et des Organismes Janvier 2015
 Andrew Tolonen [email protected] Travaux dirigés de Microbiologie Master I Sciences des Génomes et des Organismes Janvier 2015 A- Généralités I- La vie sur terre telle que nous la connaissons ne
Andrew Tolonen [email protected] Travaux dirigés de Microbiologie Master I Sciences des Génomes et des Organismes Janvier 2015 A- Généralités I- La vie sur terre telle que nous la connaissons ne
Comprendre l Univers grâce aux messages de la lumière
 Seconde / P4 Comprendre l Univers grâce aux messages de la lumière 1/ EXPLORATION DE L UNIVERS Dans notre environnement quotidien, les dimensions, les distances sont à l échelle humaine : quelques mètres,
Seconde / P4 Comprendre l Univers grâce aux messages de la lumière 1/ EXPLORATION DE L UNIVERS Dans notre environnement quotidien, les dimensions, les distances sont à l échelle humaine : quelques mètres,
Détection et prise en charge de la résistance aux antirétroviraux
 Détection et prise en charge de la résistance aux antirétroviraux Jean Ruelle, PhD AIDS Reference Laboratory, UCLouvain, Bruxelles Corata 2011, Namur, 10 juin 2011 Laboratoires de référence SIDA (Belgique)
Détection et prise en charge de la résistance aux antirétroviraux Jean Ruelle, PhD AIDS Reference Laboratory, UCLouvain, Bruxelles Corata 2011, Namur, 10 juin 2011 Laboratoires de référence SIDA (Belgique)
Mesures et incertitudes
 En physique et en chimie, toute grandeur, mesurée ou calculée, est entachée d erreur, ce qui ne l empêche pas d être exploitée pour prendre des décisions. Aujourd hui, la notion d erreur a son vocabulaire
En physique et en chimie, toute grandeur, mesurée ou calculée, est entachée d erreur, ce qui ne l empêche pas d être exploitée pour prendre des décisions. Aujourd hui, la notion d erreur a son vocabulaire
INTERSYSTEMS CACHÉ COMME ALTERNATIVE AUX BASES DE DONNÉES RÉSIDENTES EN MÉMOIRE
 I N T E RS Y S T E M S INTERSYSTEMS CACHÉ COMME ALTERNATIVE AUX BASES DE DONNÉES RÉSIDENTES EN MÉMOIRE David Kaaret InterSystems Corporation INTERSySTEMS CAChé CoMME ALTERNATIvE AUx BASES de données RéSIdENTES
I N T E RS Y S T E M S INTERSYSTEMS CACHÉ COMME ALTERNATIVE AUX BASES DE DONNÉES RÉSIDENTES EN MÉMOIRE David Kaaret InterSystems Corporation INTERSySTEMS CAChé CoMME ALTERNATIvE AUx BASES de données RéSIdENTES
TESTS D HYPOTHÈSE FONDÉS SUR LE χ². http://fr.wikipedia.org/wiki/eugénisme
 TESTS D HYPOTHÈSE FONDÉS SUR LE χ² http://fr.wikipedia.org/wiki/eugénisme Logo du Second International Congress of Eugenics 1921. «Comme un arbre, l eugénisme tire ses constituants de nombreuses sources
TESTS D HYPOTHÈSE FONDÉS SUR LE χ² http://fr.wikipedia.org/wiki/eugénisme Logo du Second International Congress of Eugenics 1921. «Comme un arbre, l eugénisme tire ses constituants de nombreuses sources
Excel 2007 Niveau 3 Page 1 www.admexcel.com
 Excel 2007 Niveau 3 Page 1 TABLE DES MATIERES UTILISATION DE LISTES DE DONNEES... 4 REMARQUES PREALABLES SUR LES LISTES DE DONNEES... 4 METTRE EN FORME LE TABLEAU... 6 METTRE LA LISTE A JOUR... 7 a/ Directement
Excel 2007 Niveau 3 Page 1 TABLE DES MATIERES UTILISATION DE LISTES DE DONNEES... 4 REMARQUES PREALABLES SUR LES LISTES DE DONNEES... 4 METTRE EN FORME LE TABLEAU... 6 METTRE LA LISTE A JOUR... 7 a/ Directement
Enseignement Informatique. Classe de Bac Pro SAPAT -----------------------------------------------------------------------
 Enseignement Informatique Classe de Bac Pro SAPAT ----------------------------------------------------------------------- MG4: Culture Scientifique et Technologique. -----------------------------------------------------------------------
Enseignement Informatique Classe de Bac Pro SAPAT ----------------------------------------------------------------------- MG4: Culture Scientifique et Technologique. -----------------------------------------------------------------------
les outils du travail collaboratif
 les outils du travail collaboratif Sommaire Qu est-ce que le travail collaboratif? A chaque usage ses outils L échange d informations Le partage d informations La gestion de projet La conception collaborative
les outils du travail collaboratif Sommaire Qu est-ce que le travail collaboratif? A chaque usage ses outils L échange d informations Le partage d informations La gestion de projet La conception collaborative
Conseil d administration Genève, novembre 2002 LILS
 BUREAU INTERNATIONAL DU TRAVAIL GB.285/LILS/1 285 e session Conseil d administration Genève, novembre 2002 Commission des questions juridiques et des normes internationales du travail LILS PREMIÈRE QUESTION
BUREAU INTERNATIONAL DU TRAVAIL GB.285/LILS/1 285 e session Conseil d administration Genève, novembre 2002 Commission des questions juridiques et des normes internationales du travail LILS PREMIÈRE QUESTION
Contrôle de l'expression génétique :
 Contrôle de l'expression génétique : Les régulations post-transcriptionnelles L'ARNm, simple intermédiaire entre le génome et les protéines? gène protéine L'ARNm, simple intermédiaire entre le génome et
Contrôle de l'expression génétique : Les régulations post-transcriptionnelles L'ARNm, simple intermédiaire entre le génome et les protéines? gène protéine L'ARNm, simple intermédiaire entre le génome et
ISO/CEI 11172-3 NORME INTERNATIONALE
 NORME INTERNATIONALE ISO/CEI 11172-3 Première édition 1993-08-01 Technologies de l information - Codage de l image animée et du son associé pour les supports de stockage numérique jusqu à environ Ii5 Mbit/s
NORME INTERNATIONALE ISO/CEI 11172-3 Première édition 1993-08-01 Technologies de l information - Codage de l image animée et du son associé pour les supports de stockage numérique jusqu à environ Ii5 Mbit/s
d évaluation Objectifs Processus d élaboration
 Présentation du Programme pancanadien d évaluation Le Programme pancanadien d évaluation (PPCE) représente le plus récent engagement du Conseil des ministres de l Éducation du Canada (CMEC) pour renseigner
Présentation du Programme pancanadien d évaluation Le Programme pancanadien d évaluation (PPCE) représente le plus récent engagement du Conseil des ministres de l Éducation du Canada (CMEC) pour renseigner
Séquençage et Assemblage. de Génomes. François Denizot Emmanuel Talla LCB-IBSM CNRS [email protected] [email protected].
 Séquençage et Assemblage de Génomes François Denizot Emmanuel Talla LCB-IBSM CNRS [email protected] [email protected] Projet de séquençage d un génome Séquençage aléatoire Assemblage Annotation
Séquençage et Assemblage de Génomes François Denizot Emmanuel Talla LCB-IBSM CNRS [email protected] [email protected] Projet de séquençage d un génome Séquençage aléatoire Assemblage Annotation
Insert System. BRANDplates. Un design intelligent et innovant jusque dans les moindres détails
 Un design intelligent et innovant jusque dans les moindres détails BRANDplates Insert System H I G H - T E C H D I S P O S A B L E S Plaques de culture cellulaire et inserts innovants de BRAND pour la
Un design intelligent et innovant jusque dans les moindres détails BRANDplates Insert System H I G H - T E C H D I S P O S A B L E S Plaques de culture cellulaire et inserts innovants de BRAND pour la
10 REPÈRES «PLUS DE MAÎTRES QUE DE CLASSES» JUIN 2013 POUR LA MISE EN ŒUVRE DU DISPOSITIF
 10 REPÈRES POUR LA MISE EN ŒUVRE DU DISPOSITIF «PLUS DE MAÎTRES QUE DE CLASSES» JUIN 2013 MEN-DGESCO 2013 Sommaire 1. LES OBJECTIFS DU DISPOSITIF 2. LES ACQUISITIONS PRIORITAIREMENT VISÉES 3. LES LIEUX
10 REPÈRES POUR LA MISE EN ŒUVRE DU DISPOSITIF «PLUS DE MAÎTRES QUE DE CLASSES» JUIN 2013 MEN-DGESCO 2013 Sommaire 1. LES OBJECTIFS DU DISPOSITIF 2. LES ACQUISITIONS PRIORITAIREMENT VISÉES 3. LES LIEUX
Modernisation et gestion de portefeuilles d applications bancaires
 Modernisation et gestion de portefeuilles d applications bancaires Principaux défis et facteurs de réussite Dans le cadre de leurs plans stratégiques à long terme, les banques cherchent à tirer profit
Modernisation et gestion de portefeuilles d applications bancaires Principaux défis et facteurs de réussite Dans le cadre de leurs plans stratégiques à long terme, les banques cherchent à tirer profit
Introduction aux bases de données: application en biologie
 Introduction aux bases de données: application en biologie D. Puthier 1 1 ERM206/Technologies Avancées pour le Génome et la Clinique, http://tagc.univ-mrs.fr/staff/puthier, [email protected] ESIL,
Introduction aux bases de données: application en biologie D. Puthier 1 1 ERM206/Technologies Avancées pour le Génome et la Clinique, http://tagc.univ-mrs.fr/staff/puthier, [email protected] ESIL,
La résistance d'agents infectieux aux médicaments antimicrobiens
 DECLARATION COMMUNE DES ACADEMIES DU G SCIENCE 2013 La résistance d'agents infectieux aux médicaments antimicrobiens Une menace globale pour l'humanité Depuis l introduction dans les années 40 du premier
DECLARATION COMMUNE DES ACADEMIES DU G SCIENCE 2013 La résistance d'agents infectieux aux médicaments antimicrobiens Une menace globale pour l'humanité Depuis l introduction dans les années 40 du premier
vaccin pneumococcique polyosidique conjugué (13-valent, adsorbé)
 EMA/90006/2015 EMEA/H/C/001104 Résumé EPAR à l intention du public vaccin pneumococcique polyosidique conjugué (13-valent, adsorbé) Le présent document est un résumé du rapport européen public d évaluation
EMA/90006/2015 EMEA/H/C/001104 Résumé EPAR à l intention du public vaccin pneumococcique polyosidique conjugué (13-valent, adsorbé) Le présent document est un résumé du rapport européen public d évaluation
Isolement automatisé d ADN génomique à partir de culots de cellules sanguines à l aide de l appareil Tecan Freedom EVO -HSM Workstation
 PROTOCOLE AUTOMATISÉ Isolement automatisé d ADN génomique à partir de culots de cellules sanguines à l aide de l appareil Tecan Freedom EVO -HSM Workstation Mode d emploi des produits A1751 et A2751 Réservé
PROTOCOLE AUTOMATISÉ Isolement automatisé d ADN génomique à partir de culots de cellules sanguines à l aide de l appareil Tecan Freedom EVO -HSM Workstation Mode d emploi des produits A1751 et A2751 Réservé
Objectifs. Clustering. Principe. Applications. Applications. Cartes de crédits. Remarques. Biologie, Génomique
 Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
Objectifs Clustering On ne sait pas ce qu on veut trouver : on laisse l algorithme nous proposer un modèle. On pense qu il existe des similarités entre les exemples. Qui se ressemble s assemble p. /55
Chapitre 5 LE MODELE ENTITE - ASSOCIATION
 Chapitre 5 LE MODELE ENTITE - ASSOCIATION 1 Introduction Conception d une base de données Domaine d application complexe : description abstraite des concepts indépendamment de leur implémentation sous
Chapitre 5 LE MODELE ENTITE - ASSOCIATION 1 Introduction Conception d une base de données Domaine d application complexe : description abstraite des concepts indépendamment de leur implémentation sous
GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010
 GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010 Analyse de la diversité moléculaire des régions génomiques de 30 gènes du développement méristématique dans une core collection
GénoToul 2010, Hôtel de Région Midi Pyrénées, Toulouse, 10 décembre 2010 Analyse de la diversité moléculaire des régions génomiques de 30 gènes du développement méristématique dans une core collection
UE : GENE 2208. Responsable : Enseignant : ECUE 1. Enseignant : ECUE 2. Dr COULIBALY Foungotin Hamidou
 UE : GENE 2208 Responsable : Enseignant : ECUE 1 Enseignant : ECUE 2 Dr COULIBALY Foungotin Hamidou Spécialités : Génétique Humaine Biologie de la Procréation Cytogénétique Spermiologie Essais Cliniques
UE : GENE 2208 Responsable : Enseignant : ECUE 1 Enseignant : ECUE 2 Dr COULIBALY Foungotin Hamidou Spécialités : Génétique Humaine Biologie de la Procréation Cytogénétique Spermiologie Essais Cliniques
Electron S.R.L. - MERLINO - MILAN ITALIE Tel (++ 39 02) 90659200 Fax 90659180 Web www.electron.it, e-mail [email protected]
 Electron S.R.L. Design Production & Trading of Educational Equipment B3510--II APPLIICATIIONS DE TRANSDUCTEURS A ULTRASONS MANUEL D IINSTRUCTIIONS POUR L ETUDIIANT Electron S.R.L. - MERLINO - MILAN ITALIE
Electron S.R.L. Design Production & Trading of Educational Equipment B3510--II APPLIICATIIONS DE TRANSDUCTEURS A ULTRASONS MANUEL D IINSTRUCTIIONS POUR L ETUDIIANT Electron S.R.L. - MERLINO - MILAN ITALIE
VI- Expression du génome
 VI- Expression du génome VI-1.- EXPRESSION DU GÉNOME- PRINCIPES GÉNÉRAUX DOGME CENTRAL Les gènes et l information génétique sont conservés sous forme d acides nucléiques La perpétuation à l identique de
VI- Expression du génome VI-1.- EXPRESSION DU GÉNOME- PRINCIPES GÉNÉRAUX DOGME CENTRAL Les gènes et l information génétique sont conservés sous forme d acides nucléiques La perpétuation à l identique de
SOCLE COMMUN: LA CULTURE SCIENTIFIQUE ET TECHNOLOGIQUE. alain salvadori IA IPR Sciences de la vie et de la Terre 2009-2010 ALAIN SALVADORI IA-IPR SVT
 SOCLE COMMUN: LA CULTURE SCIENTIFIQUE ET TECHNOLOGIQUE alain salvadori IA IPR Sciences de la vie et de la Terre 2009-2010 ALAIN SALVADORI IA-IPR SVT SOCLE COMMUN ET PROGRAMMES La référence pour la rédaction
SOCLE COMMUN: LA CULTURE SCIENTIFIQUE ET TECHNOLOGIQUE alain salvadori IA IPR Sciences de la vie et de la Terre 2009-2010 ALAIN SALVADORI IA-IPR SVT SOCLE COMMUN ET PROGRAMMES La référence pour la rédaction
